DA RA
|
|
|
- よしお なかきむら
- 7 years ago
- Views:
Transcription
1 II 3ADT
2 DA RA SAMBA k FASTA BLAST lustalw
3 1 DA o o o o SAMBA eedleman-wunsch eedleman-wunsch ( ) eedleman-wunsch DDBJ Search and Analysis FASTA BI P BLAST
4 DA (i, j)
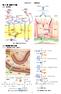
5 1 DA DA DA DA 2.1 DA DA Deoxyriboucleic Acid( ) DA RA DA RA DA ( 1 ) 2 (Bioinformatics) Biology( ) Information science( ) 1: DA Adenine( ) ytosine( ) Guanine( ) Thymine( ) 5
6 2: O 3: O 4: O O 5: 6
7 4 A(Adenine) (ytosine) G(Guanine) O T(Thymine) A T G mra DA O DA(cDA; complementary DA) DA bp(base pair; ) kbp(1,000bp) Mbp(1,000kbp) Gbp(1,000Mbp) 4 6: (omenclature ommittee of the International Union of Biochemistry) 1 3 mra(messenger RA; RA) tra(transfer RA; RA) 2.2 RA RA Riboucleic Acid( ) 4 4 Adenine( ) ytosine( ) Guanine( ) Uracil( ) DA DA DA A G U(Uracil) 6 rra(ribosome RA; RA) RA sira(small interfering RA) mira(micro RA) mra RA tra RA rra RA RA mra 7
8 G G (Guanine) A A (Adenine) T T (Thymine) (ytosine) R A G (purine) Y T (pyrimidine) M A (amino) K G T (Keto) S G 3 (Strong interactioins) W A T 2 (Weak interactioins) A T G = B G T A =B V A G T U (U RA )=V D A G T =D A G T (ay base) 1: 2.3 mra ( ) DA RA DA Geprge I. Bell 1974 (Los Alamos atioinal Laboratory) (Theoretical Biology and Biophysics Group) GenBank 4 8
9 1 2 3 U A G Phe Ser Tyr ys U U Phe Ser Tyr ys Leu Ser STOP STOP A Leu Ser STOP Trp G Leu Pro is Arg U Leu Pro is Arg Leu Pro Gln Arg A Leu Pro Gln Arg G Ile Thr Asn Ser U A Ile Thr Asn Ser Ile Thr Lys Arg A Met(START) Thr Lys Arg G Val Ala Asp Gly U G Val Ala Asp Gly Val Ala Glu Gly A Val Ala Glu Gly G 2: 1974 George I. Bell 1979 Goad GenBank 1980 (EMBL) GenBank 1984 DA (DDBJ) 1989 AceDB GenBank,EMBL,DDBJ 4: DA 9
10 1 3 A Ala ys D Asp E Glu F Phe G Gly is I Ile K Lys L Leu M Met Asn P Pro Q Gln R Arg S Ser T Thr V Val W Trp X Xxx Y Tyr Z Glx ( ) 3: DA 2.5 Fred Blattner amilton Smith 5 10
11 [kb] 1995 KW20 1, K12 4, , S288 12, , , , O157 4, , T18 4, ,100, , ,700, ,000 5: 3 DA (A G T) DA DA 1977 Maxam Gilbert Sanger (i 1, j 1) + d(x i, y j ) (i, j) = Max (1) (i 1, j) g (i, j 1) g (i, 0) = (0, j) = 0 d(x i, y j ) : x i y j g : 1 x i y j 2 3 X = (x 1, x 2,, x i,, x n ) (2) Y = (y 1, y 2,, y j,, y m ) (3) 6 (i, j) x i y j 11
12 X Y y 1 y 2 y j y m X Y A T G x 1 G (1, 1) (1, 2) (1, j) (1, m) x 2 T (2, 1) (2, 2) (2, j) (2, m) x i (i, 1) (i, 2) (i, j) (i, m) x n A (n, 1) (n, 2) (n, j) (n, m) 6: (i, j) 7 ATGGAG GTAGGTGAT A T G G A G G T A G G T G A T : 2 g d(x i, y j ) g = 1 +2, x i = y j d(x i, y j ) = 1, 7 ( 10 ) 8 2 ( ) T G G A G T A G G T G 8: 1 (i, j) (i 1, j 1) (i 1, j) (i, j 1) ( 9 ) 1 1 P i,j P i,j t 4 12
13 P( i - 2, j - 2) P( i - 2, j - 1) P( i - 2-1, j ) P( i - 1, j - 2) P( i - 1, j - 1) P( i - 1, j ) ( i - 1, j - 1) ( i - 1, j ) ( i, j +1) P( i, j - 2) P( i, j - 1) ( i, j - 1) P( i, j ) P( i, j +1) ( i +1, j ) ( i +1, j +1) P( i +1, j ) P( i +1, j +1) 9: 13
14 10: o1 11: o2 12: o3 13: o4 Sp = 5 l g : l b : : (l g l b ) l g + (l b ) 1 l g (5) SAMBA SAMBA Systolic Accelerator for Molecular Biological Applicatioins 14 SAMBA SAMBA 1995 SAMBA Web SAMBA BLAST t = i + j + 1 (4) t = Sp 5 FASTA SSEAR BISP BioSA 14
15 LSI DB FPGA 14: SAMBA KESTREL RAPID-2 FPGA SPLAS-2 ReRLe-1 FPGA SAMBA SAMBA 1 FPGA SAMBA k- 2 15
16 / / 1 eedleman Wunsch Smith Waterman 2 Dayhoff PAM250 DA k- FASTA BLAST k- 2 FASTA BLAST A.J. Gibbs G.A. McIntire ATTGGT GTTAGGTGAT 16
17 A T T G G T G T T A G G T G A T 15: 15 GGT RA Gibbs McIntyre 2 2 DA DA 20 DA 4 DA PAM250 BLOSUM
18 ?? ( 16 2 ) A A T G G A B A G A G 16: 6 g(l) = α beta (L 1) (6) 6 L: α: β: α β α 1/10 α β Dayhoff / eedleman Wunsch 2 eedleman- Wunsch ( 0 ) GTTAGGTGAT ATTGGT GTTAGGTGAT ATTGGT 19 18
19 A T T G G T G T T A 1 G G 1 1 T G 1 1 A 1 T : eedleman-wunsch A T T G G T G T T A 1 G G 1 5 T G 2 1 A 1 T : eedleman-wunsch ( ) T T G G T T T A G G T 19: eedleman-wunsch TT GGT( ) ( ) Smith Waterman DA Smith Waterman eedleman- Wunsch ( ) Smith-Waterman
20 3.2.3 k- k- ( k- ) 2 3.3?? eedleman-wunsch Smith-Waterman DA W. Pearson D. Lipman 1988 FASTA FASTA ( ) 2 BLAST 1990 S. Altschul BLAST D BI BLAST FASTA 2 BLAST DA BLAST GAPPED-BLAST BLAST 3 BLAST PSI-BLAST(Position-Specific-Iterated BLAST; BLAST) FASTA FASTA DA 2 k- k FASTA DA 20
21 FASTA 20 BI BLAST ( 20 ) FASTA k FASTA k- 1( ) k- FASTA FASTA3 FASTA FASTA3(FASTA version34) FASTA FASTA ( ;E) E (Smith-Waterman SSEAR ) u λ FASTA FASTA (z ) z ( 7 21
22 20: DDBJ Search and Analysis FASTA 22
23 Z ) z ( 7 Z ) P (Z > z) = 1 exp( e z ) (7) D z e DP z 1 E = 1 e DP P < 0.1 E = DP 8 E(Z > z) = D P (Z > z) (8) 8. z = z BLAST BLAST(Basic Local Alignment Search Tool) FASTA BLAST BI 21 BI Web BLAST ( 21 ) BLAST BI BLAST BLAST WU-BLAST WU-BLAST BI-BLAST BI BLAST BLAST FASTA k- FASTA BLAST BLOSUM62 BLAST 3 11( 6 3) FASTA FASTA BLAST BLOSUM (T )
24 21: BI P BLAST 24
25 (seed) 8. BI BLAST2 gapped BLAST BLAST T 9. SP(high-scoring segment pair; ) S S SP 10. SP 2 SP S x ρ 9 P (S x) = 1 exp( e λ(x u) ) (9) u = [log(km n )]/λ K λ BLAST n m m m (ln Kmn)/ (10) n n (ln Kmn)/ (11) 2 = (ln Kmn)/l l m n l D S x E E 1 e p(s>x)d (12) 12 p < 0.1 E = pd E D 2 E = SP 12. Smith-Waterman 25
26 SP E E GG PILEUP LUSTALW T-OFFEE ( ) PSSM(Positioin-Specific Scoring Matrix; ) 2 DA DA RA DA lustalw 1 lustalw lustalw eighbor Joining 26
27 lustalw 2 lustalw 27
28 [1] Dominique Lavenier: Speeding Up Genome omputatioins With a Systolic Accelerator [2] ova Ahmed, Yi Pan, Art Vandenberg: Parallel Algorithm for Multiple Genome Alignment on the Grid Environment [3] Gong-Xin Yu, Al Geist, George Ostrouchov, agiza F. Samatova: An SVM-based Algorithm for Identificatioin of Photosynthesis-specific Genome Features [4] David W. Mount: [5] yntbia Gibas, Per Jambeck: [6] : [7] : [8], : [9], : DA [10] DDBJ Searh and Analysis [11] BI 28
1_alignment.ppt
 " " " " n " n n " n " n n n " n n n n " n LGPSSKQTGKGW-SRIWDN! + +! LN-ITKSAGKGAIMRLGDA! " n -------TGKG--------!! -------AGKG--------! " n w w w " n w w " " " 11 12 " n w w w " n w w A! M! O! A!
" " " " n " n n " n " n n n " n n n n " n LGPSSKQTGKGW-SRIWDN! + +! LN-ITKSAGKGAIMRLGDA! " n -------TGKG--------!! -------AGKG--------! " n w w w " n w w " " " 11 12 " n w w w " n w w A! M! O! A!
Perl + α. : DNA, mrna,,
 2009 Perl + α. : DNA, mrna,, DNA .. DNA A C G T DNA 2 A-T, C-G DNA NH 2 NH 2 O - O O N P O - O CH 2 O N N O - O P O CH 2 O N O - O O P O NH 2 O - O - N CH 2 O N O OH OH OH DNA or RNA (U) (A) (G) (C)
2009 Perl + α. : DNA, mrna,, DNA .. DNA A C G T DNA 2 A-T, C-G DNA NH 2 NH 2 O - O O N P O - O CH 2 O N N O - O P O CH 2 O N O - O O P O NH 2 O - O - N CH 2 O N O OH OH OH DNA or RNA (U) (A) (G) (C)
™·”õ/sec3_p63_84/fiü“eflÅ
 Section3 64 1 65 Section 3 1 2 3 4 5 6 7 8 9 10 11 12 13 14 1 Babara E. Ainsworth et al. Med. Sci. Sports. Exevc.1993 66 67 Section 3 1 10 9 8 1 2 3 4 7 6 5 6 7 5 1 2 3 4 68 69 Section 3 2 70 Section 3
Section3 64 1 65 Section 3 1 2 3 4 5 6 7 8 9 10 11 12 13 14 1 Babara E. Ainsworth et al. Med. Sci. Sports. Exevc.1993 66 67 Section 3 1 10 9 8 1 2 3 4 7 6 5 6 7 5 1 2 3 4 68 69 Section 3 2 70 Section 3
狂牛病調査第2巻1章,2章.doc
 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 2.1 A LD50/30mg () 10 ND 1 2.8 ND ND ND 11 ND 2.3 1.0 ND ND 11 ND ND 1.3 1.7 ND 13 ND
21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 2.1 A LD50/30mg () 10 ND 1 2.8 ND ND ND 11 ND 2.3 1.0 ND ND 11 ND ND 1.3 1.7 ND 13 ND
日本化学療法学会雑誌第57巻第1号
 In vitro Streptococcus pneumoniae Escherichia coli in vitro Streptococcus pneumoniae Escherichia coli µs. pneumoniae E. coli µ Key words in vitrostreptococcus pneumoniae Escherichia coli Escherichia coli
In vitro Streptococcus pneumoniae Escherichia coli in vitro Streptococcus pneumoniae Escherichia coli µs. pneumoniae E. coli µ Key words in vitrostreptococcus pneumoniae Escherichia coli Escherichia coli
Nov12_2009.pptx
 h"p://www.ebi.ac.uk/tools/emboss/align/index.html UNIPROT TPIS_HUMAN, TPIS_RABIT FASTA Run #########################################! Program: needle# Rundate: Tue Nov 10 02:40:01 2009! #########################################!
h"p://www.ebi.ac.uk/tools/emboss/align/index.html UNIPROT TPIS_HUMAN, TPIS_RABIT FASTA Run #########################################! Program: needle# Rundate: Tue Nov 10 02:40:01 2009! #########################################!
10D16.dvi
 D IEEJ Transactions on Industry Applications Vol.136 No.10 pp.686 691 DOI: 10.1541/ieejias.136.686 NW Accelerating Techniques for Sequence Alignment based on an Extended NW Algorithm Jin Okaze, Non-member,
D IEEJ Transactions on Industry Applications Vol.136 No.10 pp.686 691 DOI: 10.1541/ieejias.136.686 NW Accelerating Techniques for Sequence Alignment based on an Extended NW Algorithm Jin Okaze, Non-member,
ŁRŁ¸•ñŁŁŁ‘
 16 11 5 25mg 14 11 18 1 25mg 1 150,000 934 1 2 : 1-235-tumor necrosis factor receptor (human) fusion protein with 236-467-immunoglobulin G1 (human γ1-chain Fc fragment), dimmer : : 1 235 236 467 G 1 γ1
16 11 5 25mg 14 11 18 1 25mg 1 150,000 934 1 2 : 1-235-tumor necrosis factor receptor (human) fusion protein with 236-467-immunoglobulin G1 (human γ1-chain Fc fragment), dimmer : : 1 235 236 467 G 1 γ1
untitled
 1 1 2 CADLIVE E-CELL 3 GEM CE/MS MGF WT MGF 4 2 WT E-Cell MASK 5 CE-MS, CE- TOFMS LC-MS 24, 5 9 2 0 6 3 CE-MS CE MS Soga, T. et al. (2002) Anal Chem. 74, 2233-2239 7 CE-MS 859 452, 269, 86 8 4 LC-MS 9
1 1 2 CADLIVE E-CELL 3 GEM CE/MS MGF WT MGF 4 2 WT E-Cell MASK 5 CE-MS, CE- TOFMS LC-MS 24, 5 9 2 0 6 3 CE-MS CE MS Soga, T. et al. (2002) Anal Chem. 74, 2233-2239 7 CE-MS 859 452, 269, 86 8 4 LC-MS 9
スライド 1
 タンパク質 ( 生化学 1) 平成 29 年 4 月 20 日病態生化学分野 分子酵素化学分野教授 山縣和也 生化学 1のスケジュール 4 月 20 日 講義開始 6 月 1 日 中間試験 9 月 25 日 生化学 1 試験 講義日程 内容は一部変更があります 講義資料 ( 山縣 吉澤分 ): 熊本大学病態生化学 で検索 ID: Biochem2 パスワード :76TgFD3Xc 生化学 1 の合否判定は
タンパク質 ( 生化学 1) 平成 29 年 4 月 20 日病態生化学分野 分子酵素化学分野教授 山縣和也 生化学 1のスケジュール 4 月 20 日 講義開始 6 月 1 日 中間試験 9 月 25 日 生化学 1 試験 講義日程 内容は一部変更があります 講義資料 ( 山縣 吉澤分 ): 熊本大学病態生化学 で検索 ID: Biochem2 パスワード :76TgFD3Xc 生化学 1 の合否判定は
3. 生化学的検査 >> 3C. 低分子窒素化合物 >> 3C045. 検体採取 患者の検査前準備 検体採取のタイミング 記号添加物 ( キャップ色等 ) 採取材料採取量測定材料ネ丸底プレイン ( 白 ) 尿 9 ml 注 外 N60 セイカ 検体ラベル ( 単項目オーダー時 ) ホンハ
 3. 生化学的検査 >> 3C. 低分子窒素化合物 >> 3C045. amino acid fractionation 基本情報 3C045 分析物 連絡先 : 3764 JLAC10 診療報酬 識別 材料 001 尿 ( 含むその他 ) 測定法 204 高速液体クラマトグラフィー (HPLC) 結果識別 第 2 章 特掲診療料 D010 5 アミノ酸 第 3 部 検査 D010 5ロ 5 種類以上
3. 生化学的検査 >> 3C. 低分子窒素化合物 >> 3C045. amino acid fractionation 基本情報 3C045 分析物 連絡先 : 3764 JLAC10 診療報酬 識別 材料 001 尿 ( 含むその他 ) 測定法 204 高速液体クラマトグラフィー (HPLC) 結果識別 第 2 章 特掲診療料 D010 5 アミノ酸 第 3 部 検査 D010 5ロ 5 種類以上
HOE901 A S S Gly Ile Val Glu Gln Cys Cys Thr Ser Ile Cys Ser Leu Tyr Gln Leu Glu Asn Tyr Cys Gly
 HOE901 A S S Gly Ile Val Glu Gln Cys Cys Thr Ser Ile Cys Ser Leu Tyr Gln Leu Glu Asn Tyr Cys Gly 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 S S S Phe Val Asn Gln His Leu Cys Gly Ser His Leu
HOE901 A S S Gly Ile Val Glu Gln Cys Cys Thr Ser Ile Cys Ser Leu Tyr Gln Leu Glu Asn Tyr Cys Gly 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 S S S Phe Val Asn Gln His Leu Cys Gly Ser His Leu
ウェブ23Brev2
 23B 23B.1 23B.1.1 23B.1.2 23B.2 23B.2.1 23B.2.2 a b 23B.3 23B.3.1 23B.3.2 23B.4 23B.4.1 23B.4.2 23B.4.3 23B.5 23B.5.1 23B.5.2 23B.6 23B.6.1 a b c Edman d e 23B.6.2 23B.6.3 23B.7 23B.7.1 a b c 23B.7.2 23B.7.3
23B 23B.1 23B.1.1 23B.1.2 23B.2 23B.2.1 23B.2.2 a b 23B.3 23B.3.1 23B.3.2 23B.4 23B.4.1 23B.4.2 23B.4.3 23B.5 23B.5.1 23B.5.2 23B.6 23B.6.1 a b c Edman d e 23B.6.2 23B.6.3 23B.7 23B.7.1 a b c 23B.7.2 23B.7.3
B0B820DFD845F9DE49256B7D0002B34
 平成 13 年 ( 行ケ ) 第 238 号特許取消決定取消請求事件 ( 平成 13 年 11 月 2 9 日口頭弁論終結 ) 判決原告バイオ-ラッドラボラトリーズ インコーポレイティド ( 旧表示ジェネティックシステムズコーポレイション ) 訴訟代理人弁護士上谷清同宇井正一同笹本摂同弁理士福本積被告特許庁長官及川耕造指定代理人後藤千恵子同森田ひとみ同茂木静代主文特許庁が平成 10 年異議第 73683
平成 13 年 ( 行ケ ) 第 238 号特許取消決定取消請求事件 ( 平成 13 年 11 月 2 9 日口頭弁論終結 ) 判決原告バイオ-ラッドラボラトリーズ インコーポレイティド ( 旧表示ジェネティックシステムズコーポレイション ) 訴訟代理人弁護士上谷清同宇井正一同笹本摂同弁理士福本積被告特許庁長官及川耕造指定代理人後藤千恵子同森田ひとみ同茂木静代主文特許庁が平成 10 年異議第 73683
Nakamura
 FASTA, BLAST, PSI-BLAST, HMMPFAM 4-1 4-2 4-3 MEDSI (2003) 4-4 DOROTHYCROWFOOTHODGKIN DOROTHY--------HODGKIN MEDSI (2003) 4-5 4-6 !Altschul, S.F., Gish, W., Miller, W., Myers, E.W. & Lipman, D.J. (1990)!
FASTA, BLAST, PSI-BLAST, HMMPFAM 4-1 4-2 4-3 MEDSI (2003) 4-4 DOROTHYCROWFOOTHODGKIN DOROTHY--------HODGKIN MEDSI (2003) 4-5 4-6 !Altschul, S.F., Gish, W., Miller, W., Myers, E.W. & Lipman, D.J. (1990)!
ナノの技術をバイオに応用
 本日まで お試し期間 なので 出席は取りません 現代生物学概論 2 遺伝子 ( プログラム ) と蛋白質 ( ナノマシン ) 先進理工学科 化学生物学研究室 准教授 生体機能システムコース 瀧真清 1 本日の概要 : 蛋白質生合成の全スキーム D から蛋白質への情報の流れ アミノ酸から蛋白質への物質の流れ 転写 D 本日は詳細は省略 アミノアシル tr 合成酵素 (RS) 翻訳 mr コドンーアンチコドンの対合
本日まで お試し期間 なので 出席は取りません 現代生物学概論 2 遺伝子 ( プログラム ) と蛋白質 ( ナノマシン ) 先進理工学科 化学生物学研究室 准教授 生体機能システムコース 瀧真清 1 本日の概要 : 蛋白質生合成の全スキーム D から蛋白質への情報の流れ アミノ酸から蛋白質への物質の流れ 転写 D 本日は詳細は省略 アミノアシル tr 合成酵素 (RS) 翻訳 mr コドンーアンチコドンの対合
Polyoxometalateが有するcysteine検出能の検討及びジスルフィド結合形成反応への展開
 チオールを特異的に酸化し 発色する 山形大学理工学研究科バイオ化学工学専攻准教授今野博行 金属酸化物クラスターアニオン ポリオキソメタレート Polyoxometalate (POM) 金属元素に酸素が6個配位した MO6を基本骨格とする金属酸化物 1 nm クラスターアニオン MO6 (octahedron) ( PxMyOz )n 構造 構成元素 対カチオン等を 制御することで 様々な機能性を
チオールを特異的に酸化し 発色する 山形大学理工学研究科バイオ化学工学専攻准教授今野博行 金属酸化物クラスターアニオン ポリオキソメタレート Polyoxometalate (POM) 金属元素に酸素が6個配位した MO6を基本骨格とする金属酸化物 1 nm クラスターアニオン MO6 (octahedron) ( PxMyOz )n 構造 構成元素 対カチオン等を 制御することで 様々な機能性を
,328 C 6426 H 9900 N 1700 O 2008 S , ,
 2458 13 4 24 1 11 9 17 1,328 C 6426 H 9900 N 1700 O 2008 S 44 145,000 148,000 10 11 27 2 13 4 24 11 9 17 (1) CD20 B CD20 (2) infusion reaction infusion-associated symptom CD20 B 1 375mg/m 2 1 4 3 Heavy
2458 13 4 24 1 11 9 17 1,328 C 6426 H 9900 N 1700 O 2008 S 44 145,000 148,000 10 11 27 2 13 4 24 11 9 17 (1) CD20 B CD20 (2) infusion reaction infusion-associated symptom CD20 B 1 375mg/m 2 1 4 3 Heavy
 1 911 9001030 9:00 A B C D E F G H I J K L M 1A0900 1B0900 1C0900 1D0900 1E0900 1F0900 1G0900 1H0900 1I0900 1J0900 1K0900 1L0900 1M0900 9:15 1A0915 1B0915 1C0915 1D0915 1E0915 1F0915 1G0915 1H0915 1I0915
1 911 9001030 9:00 A B C D E F G H I J K L M 1A0900 1B0900 1C0900 1D0900 1E0900 1F0900 1G0900 1H0900 1I0900 1J0900 1K0900 1L0900 1M0900 9:15 1A0915 1B0915 1C0915 1D0915 1E0915 1F0915 1G0915 1H0915 1I0915
Microsoft PowerPoint - protein1.ppt [互換モード]
![Microsoft PowerPoint - protein1.ppt [互換モード] Microsoft PowerPoint - protein1.ppt [互換モード]](/thumbs/92/108226396.jpg) 生物物理化学 1 セントラルドグマ (central doguma) DA から蛋白質が作られるまでの道筋 フランシス クリックが提唱した 原核生物と真核生物では 若干関与するタンパク質が異なるが 基本的には同じメカニズムで転写 翻訳 タンパク合成が行われる 原核生物 : 核膜が無い ( 構造的に区別出来る核を持たない ) 細胞 ( これを原核細胞という ) から成る生物で 細菌類や藍藻類がこれに属する
生物物理化学 1 セントラルドグマ (central doguma) DA から蛋白質が作られるまでの道筋 フランシス クリックが提唱した 原核生物と真核生物では 若干関与するタンパク質が異なるが 基本的には同じメカニズムで転写 翻訳 タンパク合成が行われる 原核生物 : 核膜が無い ( 構造的に区別出来る核を持たない ) 細胞 ( これを原核細胞という ) から成る生物で 細菌類や藍藻類がこれに属する
遺伝子発現データの クラスタリングの理論的背景
 自己組織化マップ Self-Organization Map (SOM) 自己組織化マップとは? K 平均アルゴリズムは あらかじめクラスター数 K を設定し 互いに近い値を持った各要素が同一クラスターに所属するように所属クラスターを決めてゆく 自己組織化マップは互いに近い値を持った各要素が近くなるように低い次元上にマップする 自己組織化マップは 1988 年に Kohonen が提案した (Kohonen
自己組織化マップ Self-Organization Map (SOM) 自己組織化マップとは? K 平均アルゴリズムは あらかじめクラスター数 K を設定し 互いに近い値を持った各要素が同一クラスターに所属するように所属クラスターを決めてゆく 自己組織化マップは互いに近い値を持った各要素が近くなるように低い次元上にマップする 自己組織化マップは 1988 年に Kohonen が提案した (Kohonen
スライド 1
 生化学への導入 平成 30 年 4 月 12 日 病態生化学分野教授 ( 生化学 2) 山縣和也 生化学とは 細胞の化学的構成成分 およびそれらが示す化学反応と代謝機序を取り扱う学問 ハーパー生化学 生化学 1 生化学 2 生化学 炭水化物 / 糖 脂質 アミノ酸について勉強します 糖 脂質生化学 2 脂質 アミノ酸 核酸 生化学 1 生化学と医学は密接に関係する 炭水化物 脂質 生化学 蛋白質 核酸
生化学への導入 平成 30 年 4 月 12 日 病態生化学分野教授 ( 生化学 2) 山縣和也 生化学とは 細胞の化学的構成成分 およびそれらが示す化学反応と代謝機序を取り扱う学問 ハーパー生化学 生化学 1 生化学 2 生化学 炭水化物 / 糖 脂質 アミノ酸について勉強します 糖 脂質生化学 2 脂質 アミノ酸 核酸 生化学 1 生化学と医学は密接に関係する 炭水化物 脂質 生化学 蛋白質 核酸
< > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ [ [ [ [ KULASIS
 < > Introduction to Basic Organic Chemistry 1,2 [ 2 [ 10 11 12 13 14 15 [ [ [ [ KULASIS < > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ 11 12 14 15 [ [ [ [ KULASIS < > Introduction to Basic
< > Introduction to Basic Organic Chemistry 1,2 [ 2 [ 10 11 12 13 14 15 [ [ [ [ KULASIS < > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ 11 12 14 15 [ [ [ [ KULASIS < > Introduction to Basic
A A = a 41 a 42 a 43 a 44 A (7) 1 (3) A = M 12 = = a 41 (8) a 41 a 43 a 44 (3) n n A, B a i AB = A B ii aa
 1 2 21 2 2 [ ] a 11 a 12 A = a 21 a 22 (1) A = a 11 a 22 a 12 a 21 (2) 3 3 n n A A = n ( 1) i+j a ij M ij i =1 n (3) j=1 M ij A i j (n 1) (n 1) 2-1 3 3 A A = a 11 a 12 a 13 a 21 a 22 a 23 a 31 a 32 a 33
1 2 21 2 2 [ ] a 11 a 12 A = a 21 a 22 (1) A = a 11 a 22 a 12 a 21 (2) 3 3 n n A A = n ( 1) i+j a ij M ij i =1 n (3) j=1 M ij A i j (n 1) (n 1) 2-1 3 3 A A = a 11 a 12 a 13 a 21 a 22 a 23 a 31 a 32 a 33
(1.2) T D = 0 T = D = 30 kn 1.2 (1.4) 2F W = 0 F = W/2 = 300 kn/2 = 150 kn 1.3 (1.9) R = W 1 + W 2 = = 1100 N. (1.9) W 2 b W 1 a = 0
 1 1 1.1 1.) T D = T = D = kn 1. 1.4) F W = F = W/ = kn/ = 15 kn 1. 1.9) R = W 1 + W = 6 + 5 = 11 N. 1.9) W b W 1 a = a = W /W 1 )b = 5/6) = 5 cm 1.4 AB AC P 1, P x, y x, y y x 1.4.) P sin 6 + P 1 sin 45
1 1 1.1 1.) T D = T = D = kn 1. 1.4) F W = F = W/ = kn/ = 15 kn 1. 1.9) R = W 1 + W = 6 + 5 = 11 N. 1.9) W b W 1 a = a = W /W 1 )b = 5/6) = 5 cm 1.4 AB AC P 1, P x, y x, y y x 1.4.) P sin 6 + P 1 sin 45
Microsoft PowerPoint 熊大 城野.ppt
 平成 20 年度新技術説明会 アミロイド線維形成阻害剤を用いた アミロイドーシスの治療 熊本大学大学院医学薬学研究部 病態情報解析学分野 講師 城野博史 研究背景 アミロイドーシス 生体内にアミロイド ( ナイロン様の重合物質 ) が沈着し臓器障害を引き起こす病気 ーナイロンの配列に似ているー アミロイドーシスの分類 [ 全身性アミロイドーシス ] 免疫グロブリン性アミロイドーシス 1) AL アミロイドーシス
平成 20 年度新技術説明会 アミロイド線維形成阻害剤を用いた アミロイドーシスの治療 熊本大学大学院医学薬学研究部 病態情報解析学分野 講師 城野博史 研究背景 アミロイドーシス 生体内にアミロイド ( ナイロン様の重合物質 ) が沈着し臓器障害を引き起こす病気 ーナイロンの配列に似ているー アミロイドーシスの分類 [ 全身性アミロイドーシス ] 免疫グロブリン性アミロイドーシス 1) AL アミロイドーシス
< > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ [ [ [ [ KULASIS
 < > Introduction to Basic Organic Chemistry 1,2 [ 2 [ 10 11 12 13 14 15 [ [ [ [ KULASIS < > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ 11 12 14 15 [ [ [ [ KULASIS < > Introduction to Basic
< > Introduction to Basic Organic Chemistry 1,2 [ 2 [ 10 11 12 13 14 15 [ [ [ [ KULASIS < > Introduction to Basic Physical Chemistry 1,2 2 [ advanced [ 11 12 14 15 [ [ [ [ KULASIS < > Introduction to Basic
201711grade1ouyou.pdf
 2017 11 26 1 2 52 3 12 13 22 23 32 33 42 3 5 3 4 90 5 6 A 1 2 Web Web 3 4 1 2... 5 6 7 7 44 8 9 1 2 3 1 p p >2 2 A 1 2 0.6 0.4 0.52... (a) 0.6 0.4...... B 1 2 0.8-0.2 0.52..... (b) 0.6 0.52.... 1 A B 2
2017 11 26 1 2 52 3 12 13 22 23 32 33 42 3 5 3 4 90 5 6 A 1 2 Web Web 3 4 1 2... 5 6 7 7 44 8 9 1 2 3 1 p p >2 2 A 1 2 0.6 0.4 0.52... (a) 0.6 0.4...... B 1 2 0.8-0.2 0.52..... (b) 0.6 0.52.... 1 A B 2
色覚(初組4).pm
 733 3 1 520 1 8 4 0.2500 1 30012 1 AB 3 22 3 1 2 http://www.nig.ac.jp/labs/devgen/mou.html 734 1.1 360nm 830nm 380nm 780nm 540nm 580nm 660nm 540nm 660nm Log relative corneal sensitivity 0 1 2 400 500 600
733 3 1 520 1 8 4 0.2500 1 30012 1 AB 3 22 3 1 2 http://www.nig.ac.jp/labs/devgen/mou.html 734 1.1 360nm 830nm 380nm 780nm 540nm 580nm 660nm 540nm 660nm Log relative corneal sensitivity 0 1 2 400 500 600
日本化学療法学会雑誌第66巻第2号
 173 Table 1.Detection rates of Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Ureaplasma urealyticum, Haemophilus influenzae and adenovirus from male urethritis in Urethritis No.
173 Table 1.Detection rates of Neisseria gonorrhoeae, Chlamydia trachomatis, Mycoplasma genitalium, Ureaplasma urealyticum, Haemophilus influenzae and adenovirus from male urethritis in Urethritis No.
本文/YAY116K
 pp. GH GH GH β- - D- β- β- β- - EC J Alkalitolerant mechanism and improvement of alkalitolerancy of glycoside hydrolases Satoshi Nakamura Department of Bioengineering, Tokyo Institute of Technology, J
pp. GH GH GH β- - D- β- β- β- - EC J Alkalitolerant mechanism and improvement of alkalitolerancy of glycoside hydrolases Satoshi Nakamura Department of Bioengineering, Tokyo Institute of Technology, J
GID1, Hiroaki KATO, Tomomi SATO and Miyako UEGUCHI-TANAKA: Gibberellin Receptor GID1: Gibberellin Recognition and Molecular Evolution
 52 31-36 2010 2 GID1, Hiroaki KATO, Tomomi SATO and Miyako UEGUCHI-TANAKA: Gibberellin Receptor GID1: Gibberellin Recognition and Molecular Evolution Gibberellins GAs are phytohormones essential for many
52 31-36 2010 2 GID1, Hiroaki KATO, Tomomi SATO and Miyako UEGUCHI-TANAKA: Gibberellin Receptor GID1: Gibberellin Recognition and Molecular Evolution Gibberellins GAs are phytohormones essential for many
<4D F736F F F696E74202D203692B98EE691E58A C946E8D758E74205B8CDD8AB B83685D>
 1 アミノペプチダダーゼを用いた多様なジペプチドの生産法 鳥取大学 農学学部 生物資源環境学科講師有馬二朗 研究背景 短鎖ペプチドの有用性 機能及び物性改善 構成アミノ酸の栄養機能を有する 単体アミノ酸の物性改善 単体のアミノ酸には無い優れた生理機能を発揮 医療 食品分野での幅広い応用が期待!! < 食品 > 塩味 L-Ala- L- Lys HCl 甘味 L-Asp- L- Phe-OMe ( アスパルテーム
1 アミノペプチダダーゼを用いた多様なジペプチドの生産法 鳥取大学 農学学部 生物資源環境学科講師有馬二朗 研究背景 短鎖ペプチドの有用性 機能及び物性改善 構成アミノ酸の栄養機能を有する 単体アミノ酸の物性改善 単体のアミノ酸には無い優れた生理機能を発揮 医療 食品分野での幅広い応用が期待!! < 食品 > 塩味 L-Ala- L- Lys HCl 甘味 L-Asp- L- Phe-OMe ( アスパルテーム
Vol. 32, Special Issue, S 51 S 64 (2011) 1. Web Site 1 gene chromosome Arthur, 2002;, 2002;, 2004; Web Site 2 Knudesen, 20
 Vol. 32, Special Issue, S 51 S 64 (2011) e-mail:mmatsuura@jfcr.or.jp 1. Web Site 1 gene chromosome Arthur, 2002;, 2002;, 2004; Web Site 2 Knudesen, 2002;, 2005, 2000a, 2004;, 2004, 2004, 2005a,b; Ushijima
Vol. 32, Special Issue, S 51 S 64 (2011) e-mail:mmatsuura@jfcr.or.jp 1. Web Site 1 gene chromosome Arthur, 2002;, 2002;, 2004; Web Site 2 Knudesen, 2002;, 2005, 2000a, 2004;, 2004, 2004, 2005a,b; Ushijima
小角散乱を用いて多機能タンパク質の 機能発現の分子機構を探る
 PF 研 究 会 /2008.09.18 多 機 能 タンパク 質 の 機 能 発 現 の 機 構 解 明 における 小 角 散 乱 の 役 割 Role of small-angle scattering in unraveling the recognition mechanism by a multifunctional protein 山 形 大 院 理 工, Yamagata Univ.,
PF 研 究 会 /2008.09.18 多 機 能 タンパク 質 の 機 能 発 現 の 機 構 解 明 における 小 角 散 乱 の 役 割 Role of small-angle scattering in unraveling the recognition mechanism by a multifunctional protein 山 形 大 院 理 工, Yamagata Univ.,
質量分析計を用いて腸内細菌叢が産生するD-アミノ酸を新発見―高感度ハイスループット・キラルアミノ酸解析でD-アミノ酸研究に新展開―
 参考資料 (1) 研究背景 腸内細菌叢と宿主のクロストークにおいて 腸内常在菌により代謝産生される低分子化合物は重要因子である これらの一部は 腸管上皮細胞を直接的に刺激し 大腸管腔内から吸収され血中に移行すれば宿主の細胞に直接的に影響を与える可能性が高い 長年 生体を構成しているのは L- アミノ酸のみと考えられていたが D- アミノ酸が微量ながら存在することが明らかになり 近年 その様々な生理機能や疾病との関わりに注目が集まっている
参考資料 (1) 研究背景 腸内細菌叢と宿主のクロストークにおいて 腸内常在菌により代謝産生される低分子化合物は重要因子である これらの一部は 腸管上皮細胞を直接的に刺激し 大腸管腔内から吸収され血中に移行すれば宿主の細胞に直接的に影響を与える可能性が高い 長年 生体を構成しているのは L- アミノ酸のみと考えられていたが D- アミノ酸が微量ながら存在することが明らかになり 近年 その様々な生理機能や疾病との関わりに注目が集まっている
 26 2 27 1 2 NFE 13.5 8.0 3.8 71.7 1.7 11.5 12.1 1.8 70.5 2.4 11.8 10.6 2.1 68.8 4.4 13.8 7.9 2.3 73.7 0.9 11.7 46.1 1.3 29.4 5.6 3 Defa Li Asian-Aust. J. Anim. Sci. 2002, 15(8), 1191-1197 Corn and brown
26 2 27 1 2 NFE 13.5 8.0 3.8 71.7 1.7 11.5 12.1 1.8 70.5 2.4 11.8 10.6 2.1 68.8 4.4 13.8 7.9 2.3 73.7 0.9 11.7 46.1 1.3 29.4 5.6 3 Defa Li Asian-Aust. J. Anim. Sci. 2002, 15(8), 1191-1197 Corn and brown
第十四改正日本薬局方第二追補
 461 3 3 3 5 5 6 6 10 18 19 79 81 101 113 127 133 139 145 147 152 153 156 158 165 3 19 20 21 22 22 23 24 25 26 26 27 28 29 29 30 31 dl 32 dl 33 34 34 36 36 37 38 38 38 39 40 42 42 42 43 44 45 46 46 47 48
461 3 3 3 5 5 6 6 10 18 19 79 81 101 113 127 133 139 145 147 152 153 156 158 165 3 19 20 21 22 22 23 24 25 26 26 27 28 29 29 30 31 dl 32 dl 33 34 34 36 36 37 38 38 38 39 40 42 42 42 43 44 45 46 46 47 48
I 2 ESI - Ion Trap
 I 2 ESI - Ion Trap 2011 35 ESI 520l/min 35V V Taylor 20 R N- ESI) Intens. x10 8 1.0 14+ 15+ 883.9 825.1 Cytochrome c 12000 Da 09123cyto mass00003.d: +MS 0.8 0.6 0.4 0.2 0.0 17+ 728.1 16+ 773.6 13+ 951.8
I 2 ESI - Ion Trap 2011 35 ESI 520l/min 35V V Taylor 20 R N- ESI) Intens. x10 8 1.0 14+ 15+ 883.9 825.1 Cytochrome c 12000 Da 09123cyto mass00003.d: +MS 0.8 0.6 0.4 0.2 0.0 17+ 728.1 16+ 773.6 13+ 951.8
 20 4 20 i 1 1 1.1............................ 1 1.2............................ 4 2 11 2.1................... 11 2.2......................... 11 2.3....................... 19 3 25 3.1.............................
20 4 20 i 1 1 1.1............................ 1 1.2............................ 4 2 11 2.1................... 11 2.2......................... 11 2.3....................... 19 3 25 3.1.............................
III 1 (X, d) d U d X (X, d). 1. (X, d).. (i) d(x, y) d(z, y) d(x, z) (ii) d(x, y) d(z, w) d(x, z) + d(y, w) 2. (X, d). F X.. (1), X F, (2) F 1, F 2 F
 III 1 (X, d) d U d X (X, d). 1. (X, d).. (i) d(x, y) d(z, y) d(x, z) (ii) d(x, y) d(z, w) d(x, z) + d(y, w) 2. (X, d). F X.. (1), X F, (2) F 1, F 2 F F 1 F 2 F, (3) F λ F λ F λ F. 3., A λ λ A λ. B λ λ
III 1 (X, d) d U d X (X, d). 1. (X, d).. (i) d(x, y) d(z, y) d(x, z) (ii) d(x, y) d(z, w) d(x, z) + d(y, w) 2. (X, d). F X.. (1), X F, (2) F 1, F 2 F F 1 F 2 F, (3) F λ F λ F λ F. 3., A λ λ A λ. B λ λ
‚åŁÎ“·„´Šš‡ðŠp‡¢‡½‹âfi`fiI…A…‰…S…−…Y…•‡ÌMarkovŸA“½fiI›ð’Í
 Markov 2009 10 2 Markov 2009 10 2 1 / 25 1 (GA) 2 GA 3 4 Markov 2009 10 2 2 / 25 (GA) (GA) L ( 1) I := {0, 1} L f : I (0, ) M( 2) S := I M GA (GA) f (i) i I Markov 2009 10 2 3 / 25 (GA) ρ(i, j), i, j I
Markov 2009 10 2 Markov 2009 10 2 1 / 25 1 (GA) 2 GA 3 4 Markov 2009 10 2 2 / 25 (GA) (GA) L ( 1) I := {0, 1} L f : I (0, ) M( 2) S := I M GA (GA) f (i) i I Markov 2009 10 2 3 / 25 (GA) ρ(i, j), i, j I
配列アラインメント Sequence alignment
 Traceback 以下の図に示すように 最大重みパスを D[ を用いて頂点 (m,n) から逆にたどりながら アラインメントを後ろから前へと計算していけばよい トレースバック i-,j-)=9 j-)= d=- トレースバック i-,j-)=8 j-)= d=- Traceback Procedure Traceback ( ) k ; i m; j n; while i or j do if i
Traceback 以下の図に示すように 最大重みパスを D[ を用いて頂点 (m,n) から逆にたどりながら アラインメントを後ろから前へと計算していけばよい トレースバック i-,j-)=9 j-)= d=- トレースバック i-,j-)=8 j-)= d=- Traceback Procedure Traceback ( ) k ; i m; j n; while i or j do if i
研修コーナー
 l l l l l l l l l l l α α β l µ l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l
l l l l l l l l l l l α α β l µ l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l l
国立遺伝学研究所におけるDNAデータバンク:DDBJ
 DNA DDBJ Introduction of the DNA Data Bank of Japan (DDBJ) DNA DDBJ DNA Data Bank of Japan 1986 DNA DDBJ GenBankEMBL 3 1984 19952001 4DDBJDDBJ DDBJ VPP5000 HPCDDBJ DNA DDBJ SE DDBJ Abstract The DNA Data
DNA DDBJ Introduction of the DNA Data Bank of Japan (DDBJ) DNA DDBJ DNA Data Bank of Japan 1986 DNA DDBJ GenBankEMBL 3 1984 19952001 4DDBJDDBJ DDBJ VPP5000 HPCDDBJ DNA DDBJ SE DDBJ Abstract The DNA Data
Microsoft PowerPoint 説明資料(きのこを活用してGABA富化素材を作る)林産試験場
 きのこを活用して GABA 富化素材を作る 説明内容 はじめに きのこと GABA のこと 本技術の開発と技術内容 GABA に着目した理由 きのこのGABA 生産能素材の機能性評価素材生産プロセスの効率化 本技術の活用 素材の多様化 用途企業等への期待 課題 展開 施設栽培により生産されているきのこ GABA( ギャバ ) の機能性 血圧上昇抑制精神安定成長ホルモン分泌促進その他記憶学習促進体重低減血中の中性脂肪低減
きのこを活用して GABA 富化素材を作る 説明内容 はじめに きのこと GABA のこと 本技術の開発と技術内容 GABA に着目した理由 きのこのGABA 生産能素材の機能性評価素材生産プロセスの効率化 本技術の活用 素材の多様化 用途企業等への期待 課題 展開 施設栽培により生産されているきのこ GABA( ギャバ ) の機能性 血圧上昇抑制精神安定成長ホルモン分泌促進その他記憶学習促進体重低減血中の中性脂肪低減
(1) GGT阻害剤
 (GGT) (glutathione, GSH, -L-Glu-L-Cys-Gly) Cys 1 10 mm GSSG GSH 98% - + H 3 N COO - γ- O H N O N H SH O γ-glu Cys Gly OH - (GGT, EC 2.3.2.2) GGT (GSH) GSX - Glu Cys(X)-Gly 1-2) GGT - 1 hydrolases transferases
(GGT) (glutathione, GSH, -L-Glu-L-Cys-Gly) Cys 1 10 mm GSSG GSH 98% - + H 3 N COO - γ- O H N O N H SH O γ-glu Cys Gly OH - (GGT, EC 2.3.2.2) GGT (GSH) GSX - Glu Cys(X)-Gly 1-2) GGT - 1 hydrolases transferases
gr09.dvi
 .1, θ, ϕ d = A, t dt + B, t dtd + C, t d + D, t dθ +in θdϕ.1.1 t { = f1,t t = f,t { D, t = B, t =.1. t A, tdt e φ,t dt, C, td e λ,t d.1.3,t, t d = e φ,t dt + e λ,t d + dθ +in θdϕ.1.4 { = f1,t t = f,t {
.1, θ, ϕ d = A, t dt + B, t dtd + C, t d + D, t dθ +in θdϕ.1.1 t { = f1,t t = f,t { D, t = B, t =.1. t A, tdt e φ,t dt, C, td e λ,t d.1.3,t, t d = e φ,t dt + e λ,t d + dθ +in θdϕ.1.4 { = f1,t t = f,t {
PowerPoint プレゼンテーション
 アミノ酸代謝 (1) 平成 30 年度 6 月 14 日 1 限病態生化学分野 吉澤達也 アミノ酸代謝 アミノ酸の修飾 食物タンパク質 消化吸収 分解 タンパク質 (20 種類 +α) 遊離アミノ酸 生合成 アミノ酸の生合成 ( 栄養学的非必須アミノ酸 ) 窒素 分解 炭素骨格 非タンパク質性誘導体 ( 神経伝達物質 ホルモン アミノ糖など ) 尿素サイクル 代謝中間体 尿素 糖質 脂質 エネルギー
アミノ酸代謝 (1) 平成 30 年度 6 月 14 日 1 限病態生化学分野 吉澤達也 アミノ酸代謝 アミノ酸の修飾 食物タンパク質 消化吸収 分解 タンパク質 (20 種類 +α) 遊離アミノ酸 生合成 アミノ酸の生合成 ( 栄養学的非必須アミノ酸 ) 窒素 分解 炭素骨格 非タンパク質性誘導体 ( 神経伝達物質 ホルモン アミノ糖など ) 尿素サイクル 代謝中間体 尿素 糖質 脂質 エネルギー
ii 3.,. 4. F. (), ,,. 8.,. 1. (75%) (25%) =7 20, =7 21 (. ). 1.,, (). 3.,. 1. ().,.,.,.,.,. () (12 )., (), 0. 2., 1., 0,.
 24(2012) (1 C106) 4 11 (2 C206) 4 12 http://www.math.is.tohoku.ac.jp/~obata,.,,,.. 1. 2. 3. 4. 5. 6. 7.,,. 1., 2007 (). 2. P. G. Hoel, 1995. 3... 1... 2.,,. ii 3.,. 4. F. (),.. 5... 6.. 7.,,. 8.,. 1. (75%)
24(2012) (1 C106) 4 11 (2 C206) 4 12 http://www.math.is.tohoku.ac.jp/~obata,.,,,.. 1. 2. 3. 4. 5. 6. 7.,,. 1., 2007 (). 2. P. G. Hoel, 1995. 3... 1... 2.,,. ii 3.,. 4. F. (),.. 5... 6.. 7.,,. 8.,. 1. (75%)
I z n+1 = zn 2 + c (c ) c pd L.V. K. 2
 I 2012 00-1 I : October 1, 2012 Version : 1.1 3. 10 1 10 15 10 22 1: 10 29 11 5 11 12 11 19 2: 11 26 12 3 12 10 12 17 3: 12 25 1 9 1 21 3 1 I 2012 00-2 z n+1 = zn 2 + c (c ) c http://www.math.nagoya-u.ac.jp/~kawahira/courses/12w-tenbou.html
I 2012 00-1 I : October 1, 2012 Version : 1.1 3. 10 1 10 15 10 22 1: 10 29 11 5 11 12 11 19 2: 11 26 12 3 12 10 12 17 3: 12 25 1 9 1 21 3 1 I 2012 00-2 z n+1 = zn 2 + c (c ) c http://www.math.nagoya-u.ac.jp/~kawahira/courses/12w-tenbou.html
Frontier Simulation Software for Industrial Science
 地 球 シミュレータ 産 業 利 用 シンポジウム2010 スーパーコンピュータでインフルエンザウイルス の 変 異 の 仕 組 みを 探 る 田 中 成 典 神 戸 大 学 大 学 院 システム 情 報 学 研 究 科 計 算 科 学 専 攻 計 算 生 物 学 講 座 インシリコ バイオロジー(In Silico Biology) コンピュータの 中 で 生 命 現 象 を 再 構 築 する さらに
地 球 シミュレータ 産 業 利 用 シンポジウム2010 スーパーコンピュータでインフルエンザウイルス の 変 異 の 仕 組 みを 探 る 田 中 成 典 神 戸 大 学 大 学 院 システム 情 報 学 研 究 科 計 算 科 学 専 攻 計 算 生 物 学 講 座 インシリコ バイオロジー(In Silico Biology) コンピュータの 中 で 生 命 現 象 を 再 構 築 する さらに
Bethe-Bloch Bethe-Bloch (stopping range) Bethe-Bloch FNAL (Fermi National Accelerator Laboratory) - (SciBooNE ) SciBooNE Bethe-Bloch FNAL - (SciBooNE
 21 2 27 Bethe-Bloch Bethe-Bloch (stopping range) Bethe-Bloch FNAL (Fermi National Accelerator Laboratory) - (SciBooNE ) SciBooNE Bethe-Bloch FNAL - (SciBooNE ) Bethe-Bloch 1 0.1..............................
21 2 27 Bethe-Bloch Bethe-Bloch (stopping range) Bethe-Bloch FNAL (Fermi National Accelerator Laboratory) - (SciBooNE ) SciBooNE Bethe-Bloch FNAL - (SciBooNE ) Bethe-Bloch 1 0.1..............................
_0212_68<5A66><4EBA><79D1>_<6821><4E86><FF08><30C8><30F3><30DC><306A><3057><FF09>.pdf

2 1 1 α = a + bi(a, b R) α (conjugate) α = a bi α (absolute value) α = a 2 + b 2 α (norm) N(α) = a 2 + b 2 = αα = α 2 α (spure) (trace) 1 1. a R aα =
 1 1 α = a + bi(a, b R) α (conjugate) α = a bi α (absolute value) α = a + b α (norm) N(α) = a + b = αα = α α (spure) (trace) 1 1. a R aα = aα. α = α 3. α + β = α + β 4. αβ = αβ 5. β 0 6. α = α ( ) α = α
1 1 α = a + bi(a, b R) α (conjugate) α = a bi α (absolute value) α = a + b α (norm) N(α) = a + b = αα = α α (spure) (trace) 1 1. a R aα = aα. α = α 3. α + β = α + β 4. αβ = αβ 5. β 0 6. α = α ( ) α = α
 ( ) 2002 1 1 1 1.1....................................... 1 1.1.1................................. 1 1.1.2................................. 1 1.1.3................... 3 1.1.4......................................
( ) 2002 1 1 1 1.1....................................... 1 1.1.1................................. 1 1.1.2................................. 1 1.1.3................... 3 1.1.4......................................
CEQ8000 ver7.0-ad 遺伝子解析システム CEQ8000 Tutorial Manual
 CEQ8000 ver7.0-ad 遺伝子解析システム CEQ8000 Tutorial Manual CEQ8000 ver7.0-ad 遺伝子解析システム CEQ8000 Tutorial Manual Chapter 1 サンプル調整 1) 消耗品類に関して 2 2) DNA サンプル調整 3 3) シーケンシング反応 4 4) エタノール沈殿 5 Chapter 2 ソフトウエアとハードウエアの起動
CEQ8000 ver7.0-ad 遺伝子解析システム CEQ8000 Tutorial Manual CEQ8000 ver7.0-ad 遺伝子解析システム CEQ8000 Tutorial Manual Chapter 1 サンプル調整 1) 消耗品類に関して 2 2) DNA サンプル調整 3 3) シーケンシング反応 4 4) エタノール沈殿 5 Chapter 2 ソフトウエアとハードウエアの起動
 (Katsue Ishii) a) Gly, Ala, Ser, Thr, Pro b) Val, Leu, Ile, Trp, Tyr, Phe, Lys, Arg c) Asp, Glu ペ プチ ドの呈 味 に及 ぼ す影 響 乏 し い 牛 肉 エ キ ス に 添 加 し て 調 べ た 分 子 量 1,000 10,000の 画 分 が 酸 味 を抑 制 し ま ろ や か に す る
(Katsue Ishii) a) Gly, Ala, Ser, Thr, Pro b) Val, Leu, Ile, Trp, Tyr, Phe, Lys, Arg c) Asp, Glu ペ プチ ドの呈 味 に及 ぼ す影 響 乏 し い 牛 肉 エ キ ス に 添 加 し て 調 べ た 分 子 量 1,000 10,000の 画 分 が 酸 味 を抑 制 し ま ろ や か に す る
1 Nippon Shokuhin Kagaku Kogaku Kaishi Vol. /., No.1, (,**1) 313 * ** * ** Regional Chemical Composition and Microbial Flora of Commercial Hat
 1 Nippon Shokuhin Kgku Kogku Kishi Vol. /., No.1, -+--+3 (,**1) 313 * ** * ** Regionl Chemicl Composition nd Microbil Flor of Commercil Hthtzushi from Akit Prefecture Kenichi Tsukmoto, Kzuki Toed, Tsutomu
1 Nippon Shokuhin Kgku Kogku Kishi Vol. /., No.1, -+--+3 (,**1) 313 * ** * ** Regionl Chemicl Composition nd Microbil Flor of Commercil Hthtzushi from Akit Prefecture Kenichi Tsukmoto, Kzuki Toed, Tsutomu
1.9 一般的名称に係る文書 1.9 一般的名称に係る文書 国際一般名 (INN) thrombomodulin alfa(r-inn List54, WHO Drug Information, Vol.19, No.3, 2005) 一般的名称 (JAN) 一般的名称 (J
 リコモジュリン点滴静注用 12800 トロンボモデュリンアルファ ( 遺伝子組換え ) 第 1 部 1.9 一般的名称に係る文書 1.9 一般的名称に係る文書 1.9 一般的名称に係る文書 1.9.1 国際一般名 (INN) thrombomodulin alfa(r-inn List54, WHO Drug Information, Vol.19, No.3, 2005) 1.9.2 一般的名称
リコモジュリン点滴静注用 12800 トロンボモデュリンアルファ ( 遺伝子組換え ) 第 1 部 1.9 一般的名称に係る文書 1.9 一般的名称に係る文書 1.9 一般的名称に係る文書 1.9.1 国際一般名 (INN) thrombomodulin alfa(r-inn List54, WHO Drug Information, Vol.19, No.3, 2005) 1.9.2 一般的名称
) ] [ h m x + y + + V x) φ = Eφ 1) z E = i h t 13) x << 1) N n n= = N N + 1) 14) N n n= = N N + 1)N + 1) 6 15) N n 3 n= = 1 4 N N + 1) 16) N n 4
![) ] [ h m x + y + + V x) φ = Eφ 1) z E = i h t 13) x << 1) N n n= = N N + 1) 14) N n n= = N N + 1)N + 1) 6 15) N n 3 n= = 1 4 N N + 1) 16) N n 4 ) ] [ h m x + y + + V x) φ = Eφ 1) z E = i h t 13) x << 1) N n n= = N N + 1) 14) N n n= = N N + 1)N + 1) 6 15) N n 3 n= = 1 4 N N + 1) 16) N n 4](/thumbs/92/107866707.jpg) 1. k λ ν ω T v p v g k = π λ ω = πν = π T v p = λν = ω k v g = dω dk 1) ) 3) 4). p = hk = h λ 5) E = hν = hω 6) h = h π 7) h =6.6618 1 34 J sec) hc=197.3 MeV fm = 197.3 kev pm= 197.3 ev nm = 1.97 1 3 ev
1. k λ ν ω T v p v g k = π λ ω = πν = π T v p = λν = ω k v g = dω dk 1) ) 3) 4). p = hk = h λ 5) E = hν = hω 6) h = h π 7) h =6.6618 1 34 J sec) hc=197.3 MeV fm = 197.3 kev pm= 197.3 ev nm = 1.97 1 3 ev
Powered by TCPDF ( Title 第 11 講 : フィッシャー統計学 II Sub Title Author 石川, 史郎 (Ishikawa, Shiro) Publisher Publication year 2018 Jtitle コペンハーゲン解
 Powered by TCPDF (www.tcpdf.org) Title 第 11 講 : フィッシャー統計学 II Sub Title Author 石川, 史郎 (Ishikawa, Shiro) Publisher Publication year 018 Jtitle コペンハーゲン解釈 ; 量子哲学 (018. 3),p.381-390 Abstract Notes 慶應義塾大学理工学部大学院講義ノート
Powered by TCPDF (www.tcpdf.org) Title 第 11 講 : フィッシャー統計学 II Sub Title Author 石川, 史郎 (Ishikawa, Shiro) Publisher Publication year 018 Jtitle コペンハーゲン解釈 ; 量子哲学 (018. 3),p.381-390 Abstract Notes 慶應義塾大学理工学部大学院講義ノート
,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i
![,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i ,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i](/thumbs/87/95421301.jpg) Armitage.? SAS.2 µ, µ 2, µ 3 a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 µ, µ 2, µ 3 log a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 * 2 2. y t y y y Poisson y * ,, Poisson 3 3. t t y,, y n Nµ,
Armitage.? SAS.2 µ, µ 2, µ 3 a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 µ, µ 2, µ 3 log a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 * 2 2. y t y y y Poisson y * ,, Poisson 3 3. t t y,, y n Nµ,
生物学入門
 第 10 章タンパク質のはたらき これまでの学習で遺伝子はポリペプチド鎖をコードしていることがわかった タンパク質の構造については第 2 章で大まかなことを述べたが ポリペプチド鎖の構造と機能の関係 ポリペプチド鎖とタンパク質の関係を 具体的な例をあげて述べていこう 生物学をあまり知らない人でも ヘモグロビンという名前を聞いたことがあるだろう 赤血球中にある酸素を運搬するタンパク質である 血液を取ってきて赤血球を集めると
第 10 章タンパク質のはたらき これまでの学習で遺伝子はポリペプチド鎖をコードしていることがわかった タンパク質の構造については第 2 章で大まかなことを述べたが ポリペプチド鎖の構造と機能の関係 ポリペプチド鎖とタンパク質の関係を 具体的な例をあげて述べていこう 生物学をあまり知らない人でも ヘモグロビンという名前を聞いたことがあるだろう 赤血球中にある酸素を運搬するタンパク質である 血液を取ってきて赤血球を集めると
ii 3.,. 4. F. ( ), ,,. 8.,. 1. (75% ) (25% ) =7 24, =7 25, =7 26 (. ). 1.,, ( ). 3.,...,.,.,.,.,. ( ) (1 2 )., ( ), 0., 1., 0,.
 (1 C205) 4 10 (2 C206) 4 11 (2 B202) 4 12 25(2013) http://www.math.is.tohoku.ac.jp/~obata,.,,,..,,. 1. 2. 3. 4. 5. 6. 7. 8. 1., 2007 ( ).,. 2. P. G., 1995. 3. J. C., 1988. 1... 2.,,. ii 3.,. 4. F. ( ),..
(1 C205) 4 10 (2 C206) 4 11 (2 B202) 4 12 25(2013) http://www.math.is.tohoku.ac.jp/~obata,.,,,..,,. 1. 2. 3. 4. 5. 6. 7. 8. 1., 2007 ( ).,. 2. P. G., 1995. 3. J. C., 1988. 1... 2.,,. ii 3.,. 4. F. ( ),..
講義資料2012
 第 10 章脂質の代謝 10.1 はじめに 長鎖脂肪トリアシルグリセロール 生物化学 中鎖脂肪トリアシルグリセロール 講義資料 膵リパ - ゼ 1,2- ジアシルグリセロール膵リパ - ゼ 管腔内消化 ミセル 2-アシルグリセロール 1-アシルグリセロールイソメラ-ゼ膵リパ-ゼ 小腸上皮 再エステル化 oa アシルoA oa リンパ管 キロミクロン 図 1 脂質の消化と取り込み 10.2 脂肪酸の異化代謝
第 10 章脂質の代謝 10.1 はじめに 長鎖脂肪トリアシルグリセロール 生物化学 中鎖脂肪トリアシルグリセロール 講義資料 膵リパ - ゼ 1,2- ジアシルグリセロール膵リパ - ゼ 管腔内消化 ミセル 2-アシルグリセロール 1-アシルグリセロールイソメラ-ゼ膵リパ-ゼ 小腸上皮 再エステル化 oa アシルoA oa リンパ管 キロミクロン 図 1 脂質の消化と取り込み 10.2 脂肪酸の異化代謝
本日の道筋 1. 生命体を構成する細胞 2. 遺伝情報の継承 3. 遺伝情報からタンパク質へ 4. 生体内での情報の流れ 5. ゲノムと遺伝子産物の解明と技術 6. 遺伝子多型 遺伝病 関連解析 7. トランスクリプトーム解析 ヒト遺伝子アノテーションデータベース 生化学 発生学 2
 生命体の成り立ちと情報の流れ 理化学研究所ゲノム医科学研究センター統計解析 技術開発グループ統計解析研究チーム山口由美 yyamaguc@src.riken.jp 1 本日の道筋 1. 生命体を構成する細胞 2. 遺伝情報の継承 3. 遺伝情報からタンパク質へ 4. 生体内での情報の流れ 5. ゲノムと遺伝子産物の解明と技術 6. 遺伝子多型 遺伝病 関連解析 7. トランスクリプトーム解析 ヒト遺伝子アノテーションデータベース
生命体の成り立ちと情報の流れ 理化学研究所ゲノム医科学研究センター統計解析 技術開発グループ統計解析研究チーム山口由美 yyamaguc@src.riken.jp 1 本日の道筋 1. 生命体を構成する細胞 2. 遺伝情報の継承 3. 遺伝情報からタンパク質へ 4. 生体内での情報の流れ 5. ゲノムと遺伝子産物の解明と技術 6. 遺伝子多型 遺伝病 関連解析 7. トランスクリプトーム解析 ヒト遺伝子アノテーションデータベース
AJACS18_ ppt
 1, 1, 1, 1, 1, 1,2, 1,2, 1 1 DDBJ 2 AJACS3 2010 6 414:20-15:20 2231 DDBJ DDBJ DDBJ DDBJ NCBI (GenBank) DDBJ EBI (EMBL-Bank) GEO DDBJ Omics ARchive(DOR) ArrayExpress DTA (DDBJ Trace Archive) DRA (DDBJ
1, 1, 1, 1, 1, 1,2, 1,2, 1 1 DDBJ 2 AJACS3 2010 6 414:20-15:20 2231 DDBJ DDBJ DDBJ DDBJ NCBI (GenBank) DDBJ EBI (EMBL-Bank) GEO DDBJ Omics ARchive(DOR) ArrayExpress DTA (DDBJ Trace Archive) DRA (DDBJ
第4章.doc
 101 4 1 102 6) 1 103 104 105 106 107 108 109 110 (Skinner) 111 David1993, p6 112 113 114 115 J.S. Mill 3 116 117 118 4. 119 120 121 122 4.1 (1920) 200115 SCIENCE ART 1) 2) 3) 123 200115 16 124 125 126
101 4 1 102 6) 1 103 104 105 106 107 108 109 110 (Skinner) 111 David1993, p6 112 113 114 115 J.S. Mill 3 116 117 118 4. 119 120 121 122 4.1 (1920) 200115 SCIENCE ART 1) 2) 3) 123 200115 16 124 125 126
…Q…m…•ŒÊŁt‡¯
 Genome Sequence ACEDB (genomic database system) Ace.mbly (graphic interactive program to support shotgun and directed sequencing projects) ARTEMIS II (non-clinical product safety assessment) Bass (lane
Genome Sequence ACEDB (genomic database system) Ace.mbly (graphic interactive program to support shotgun and directed sequencing projects) ARTEMIS II (non-clinical product safety assessment) Bass (lane
I A A441 : April 15, 2013 Version : 1.1 I Kawahira, Tomoki TA (Shigehiro, Yoshida )
 I013 00-1 : April 15, 013 Version : 1.1 I Kawahira, Tomoki TA (Shigehiro, Yoshida) http://www.math.nagoya-u.ac.jp/~kawahira/courses/13s-tenbou.html pdf * 4 15 4 5 13 e πi = 1 5 0 5 7 3 4 6 3 6 10 6 17
I013 00-1 : April 15, 013 Version : 1.1 I Kawahira, Tomoki TA (Shigehiro, Yoshida) http://www.math.nagoya-u.ac.jp/~kawahira/courses/13s-tenbou.html pdf * 4 15 4 5 13 e πi = 1 5 0 5 7 3 4 6 3 6 10 6 17
Bioinformatics2
 バイオインフォマティクス配列データ解析 2 藤 博幸 データベース検索 (1) ブラウザで NCBI を検索 (2)NCBI で配列データの取得 (3)NCBI で BLAST 検索 ブラウザで NCBI を検索 ブラウザで NCBI を検索 クリック ブラウザで NCBI を検索 NCBI トップページ National Center for Biotechnology Information 分
バイオインフォマティクス配列データ解析 2 藤 博幸 データベース検索 (1) ブラウザで NCBI を検索 (2)NCBI で配列データの取得 (3)NCBI で BLAST 検索 ブラウザで NCBI を検索 ブラウザで NCBI を検索 クリック ブラウザで NCBI を検索 NCBI トップページ National Center for Biotechnology Information 分
2 1 κ c(t) = (x(t), y(t)) ( ) det(c (t), c x (t)) = det (t) x (t) y (t) y = x (t)y (t) x (t)y (t), (t) c (t) = (x (t)) 2 + (y (t)) 2. c (t) =
 1 1 1.1 I R 1.1.1 c : I R 2 (i) c C (ii) t I c (t) (0, 0) c (t) c(i) c c(t) 1.1.2 (1) (2) (3) (1) r > 0 c : R R 2 : t (r cos t, r sin t) (2) C f : I R c : I R 2 : t (t, f(t)) (3) y = x c : R R 2 : t (t,
1 1 1.1 I R 1.1.1 c : I R 2 (i) c C (ii) t I c (t) (0, 0) c (t) c(i) c c(t) 1.1.2 (1) (2) (3) (1) r > 0 c : R R 2 : t (r cos t, r sin t) (2) C f : I R c : I R 2 : t (t, f(t)) (3) y = x c : R R 2 : t (t,
新技術説明会 様式例
 1 D- アミノ酸を利用した 旨み増強日本酒の製造方法 関西大学化学生命工学部 生命 生物工学科 教授老川典夫 研究背景 D- アミノ酸と L- アミノ酸 物理化学的性質 ( 融点 沸点 溶解度など ): 同じ生理学的性質 : 異なる 2 3 D- アミノ酸研究の展開 (1) ~ 微生物から動物へ ~ 1940,1950 年代ピリドキサール, 金属, アルブミンの混合物がアミノ酸のラセミ化を触媒することが発見される
1 D- アミノ酸を利用した 旨み増強日本酒の製造方法 関西大学化学生命工学部 生命 生物工学科 教授老川典夫 研究背景 D- アミノ酸と L- アミノ酸 物理化学的性質 ( 融点 沸点 溶解度など ): 同じ生理学的性質 : 異なる 2 3 D- アミノ酸研究の展開 (1) ~ 微生物から動物へ ~ 1940,1950 年代ピリドキサール, 金属, アルブミンの混合物がアミノ酸のラセミ化を触媒することが発見される
2. 背景タンパク質を構成するアミノ酸には L-アミノ酸と D-アミノ酸の 2 つの鏡像異性体が存在します ( 図 1) これまで生物は L-アミノ酸のみを選択的に利用していると考えられてきました ところが分析技術の進歩と共に 生物の体内に少量ながらも D-アミノ酸が存在することが分かってきました
 国立研究開発法人海洋研究開発機構 国立大学法人京都大学 深海にひろがる鏡の向こうの微生物世界 D アミノ酸を好む深海微生物を発見 1. 概要国立研究開発法人海洋研究開発機構 ( 理事長平朝彦 以下 JAMSTEC ) 海洋生命理工学研究開発センターは 国立大学法人京都大学と共同で 有人潜水調査船 しんかい 6500 無人探査機 ハイパードルフィン 等により深海から採取した堆積物から D-アミノ酸を好んで食べて増殖する微生物を発見しました
国立研究開発法人海洋研究開発機構 国立大学法人京都大学 深海にひろがる鏡の向こうの微生物世界 D アミノ酸を好む深海微生物を発見 1. 概要国立研究開発法人海洋研究開発機構 ( 理事長平朝彦 以下 JAMSTEC ) 海洋生命理工学研究開発センターは 国立大学法人京都大学と共同で 有人潜水調査船 しんかい 6500 無人探査機 ハイパードルフィン 等により深海から採取した堆積物から D-アミノ酸を好んで食べて増殖する微生物を発見しました
(5) 75 (a) (b) ( 1 ) v ( 1 ) E E 1 v (a) ( 1 ) x E E (b) (a) (b)
 (5) 74 Re, bondar laer (Prandtl) Re z ω z = x (5) 75 (a) (b) ( 1 ) v ( 1 ) E E 1 v (a) ( 1 ) x E E (b) (a) (b) (5) 76 l V x ) 1/ 1 ( 1 1 1 δ δ = x Re x p V x t V l l (1-1) 1/ 1 δ δ δ δ = x Re p V x t V
(5) 74 Re, bondar laer (Prandtl) Re z ω z = x (5) 75 (a) (b) ( 1 ) v ( 1 ) E E 1 v (a) ( 1 ) x E E (b) (a) (b) (5) 76 l V x ) 1/ 1 ( 1 1 1 δ δ = x Re x p V x t V l l (1-1) 1/ 1 δ δ δ δ = x Re p V x t V
Res. Org. Geochem. 31, 1 17 (2015) 地球 環境有機分子質量分析マニュアル No.29 技術論文 LC/ESI-MS * ** ** ** はじめに Eksborg and Schill, 1973; Gloor and
 Res. rg. Geochem. 31, 1 17 (2015) 地球 環境有機分子質量分析マニュアル No.29 技術論文 LC/ESI-MS * ** ** ** 2014 9 16 2014 12 1 1. はじめに Eksborg and Schill, 1973; Gloor and Johnson, 1977; Chaimbault et al., 2000 Knox and Hartwick,
Res. rg. Geochem. 31, 1 17 (2015) 地球 環境有機分子質量分析マニュアル No.29 技術論文 LC/ESI-MS * ** ** ** 2014 9 16 2014 12 1 1. はじめに Eksborg and Schill, 1973; Gloor and Johnson, 1977; Chaimbault et al., 2000 Knox and Hartwick,
Part () () Γ Part ,
 Contents a 6 6 6 6 6 6 6 7 7. 8.. 8.. 8.3. 8 Part. 9. 9.. 9.. 3. 3.. 3.. 3 4. 5 4.. 5 4.. 9 4.3. 3 Part. 6 5. () 6 5.. () 7 5.. 9 5.3. Γ 3 6. 3 6.. 3 6.. 3 6.3. 33 Part 3. 34 7. 34 7.. 34 7.. 34 8. 35
Contents a 6 6 6 6 6 6 6 7 7. 8.. 8.. 8.3. 8 Part. 9. 9.. 9.. 3. 3.. 3.. 3 4. 5 4.. 5 4.. 9 4.3. 3 Part. 6 5. () 6 5.. () 7 5.. 9 5.3. Γ 3 6. 3 6.. 3 6.. 3 6.3. 33 Part 3. 34 7. 34 7.. 34 7.. 34 8. 35
meiji_resume_1.PDF
 β β β (q 1,q,..., q n ; p 1, p,..., p n ) H(q 1,q,..., q n ; p 1, p,..., p n ) Hψ = εψ ε k = k +1/ ε k = k(k 1) (x, y, z; p x, p y, p z ) (r; p r ), (θ; p θ ), (ϕ; p ϕ ) ε k = 1/ k p i dq i E total = E
β β β (q 1,q,..., q n ; p 1, p,..., p n ) H(q 1,q,..., q n ; p 1, p,..., p n ) Hψ = εψ ε k = k +1/ ε k = k(k 1) (x, y, z; p x, p y, p z ) (r; p r ), (θ; p θ ), (ϕ; p ϕ ) ε k = 1/ k p i dq i E total = E
創薬と結晶構造解析-2.key
 ( 460-377 ( ) ature Reviews Cancer 1, 11 (2001) Arachidonic acid cascade CX-1 / CX-2 cyclooxygenase (magic bullet) (??) (CX) CX E2 (?) BIAP(?) CX Arachidonic acid cascade Phospholipid Aspirin Celecoxib
( 460-377 ( ) ature Reviews Cancer 1, 11 (2001) Arachidonic acid cascade CX-1 / CX-2 cyclooxygenase (magic bullet) (??) (CX) CX E2 (?) BIAP(?) CX Arachidonic acid cascade Phospholipid Aspirin Celecoxib
本文/YA3180D
 pp. D- Frontier Science in Amino Acid and Protein Research D- D- N - D- D- D-DAP, EC D-DaaA Ser β- D D- Discovery of D-stereoselective amino acid amidases, and their use in kinetic resolution of amino
pp. D- Frontier Science in Amino Acid and Protein Research D- D- N - D- D- D-DAP, EC D-DaaA Ser β- D D- Discovery of D-stereoselective amino acid amidases, and their use in kinetic resolution of amino
NL10
 Information September, 2006 1 2 Japanese Association for Molecular Target Therapy of Cancer News Letter No.10 September, 2006 3 2006 4 Japanese Association for Molecular Target Therapy of Cancer News Letter
Information September, 2006 1 2 Japanese Association for Molecular Target Therapy of Cancer News Letter No.10 September, 2006 3 2006 4 Japanese Association for Molecular Target Therapy of Cancer News Letter
K E N Z OU
 K E N Z OU 11 1 1 1.1..................................... 1.1.1............................ 1.1..................................................................................... 4 1.........................................
K E N Z OU 11 1 1 1.1..................................... 1.1.1............................ 1.1..................................................................................... 4 1.........................................
Chapter9 9 LDPC sum-product LDPC 9.1 ( ) 9.2 c 1, c 2, {0, 1, } SUM, PROD : {0, 1, } {0, 1, } SUM(c 1, c 2,, c n ) := { c1 + + c n (c n0 (1 n
 9 LDPC sum-product 9.1 9.2 LDPC 9.1 ( ) 9.2 c 1, c 2, {0, 1, } SUM, PROD : {0, 1, } {0, 1, } SUM(c 1, c 2,, c n ) := { c1 + + c n (c n0 (1 n 0 n)) ( ) 0 (N(0 c) > N(1 c)) PROD(c 1, c 2,, c n ) := 1 (N(0
9 LDPC sum-product 9.1 9.2 LDPC 9.1 ( ) 9.2 c 1, c 2, {0, 1, } SUM, PROD : {0, 1, } {0, 1, } SUM(c 1, c 2,, c n ) := { c1 + + c n (c n0 (1 n 0 n)) ( ) 0 (N(0 c) > N(1 c)) PROD(c 1, c 2,, c n ) := 1 (N(0
2 G(k) e ikx = (ik) n x n n! n=0 (k ) ( ) X n = ( i) n n k n G(k) k=0 F (k) ln G(k) = ln e ikx n κ n F (k) = F (k) (ik) n n= n! κ n κ n = ( i) n n k n
 . X {x, x 2, x 3,... x n } X X {, 2, 3, 4, 5, 6} X x i P i. 0 P i 2. n P i = 3. P (i ω) = i ω P i P 3 {x, x 2, x 3,... x n } ω P i = 6 X f(x) f(x) X n n f(x i )P i n x n i P i X n 2 G(k) e ikx = (ik) n
. X {x, x 2, x 3,... x n } X X {, 2, 3, 4, 5, 6} X x i P i. 0 P i 2. n P i = 3. P (i ω) = i ω P i P 3 {x, x 2, x 3,... x n } ω P i = 6 X f(x) f(x) X n n f(x i )P i n x n i P i X n 2 G(k) e ikx = (ik) n
 21 2 26 i 1 1 1.1............................ 1 1.2............................ 3 2 9 2.1................... 9 2.2.......... 9 2.3................... 11 2.4....................... 12 3 15 3.1..........
21 2 26 i 1 1 1.1............................ 1 1.2............................ 3 2 9 2.1................... 9 2.2.......... 9 2.3................... 11 2.4....................... 12 3 15 3.1..........














