kubo2017sep16a p.1 ( 1 ) : : :55 kubo ( ( 1 ) / 10
|
|
|
- つねたけ かみいしづ
- 4 years ago
- Views:
Transcription
1 kubo2017sep16a p.1 ( 1 ) kubo@ees.hokudai.ac.jp : : :55 kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 :! 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル : GLM GLIM General Linear Model kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 () 1 2 :? 3 GLM: GLM R GLM 4 GLM GLM 1. kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
2 kubo2017sep16a p.2 : :?! ( ) method section p < 0.05 OK kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106? (): kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : 0, 1, 2 (y {0, 1, 2, 3, } ) y x y? kubo ( ( 1 ) / 106 x y ? y 0? x NO! kubo ( ( 1 ) / 106
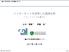
3 kubo2017sep16a p.3? y x YES! kubo ( ( 1 ) / 106 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル kubo ( ( 1 ) / 106 : : (GLM) 3. MCMC 2. : : 1, 2, 3 4. GLM kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : : () : : kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
4 kubo2017sep16a p.4 : () : R i 50 i {1, 2, 3,, 50} y i {y i }! {y i } = {y 1, y 2,, y 50 } {y i } R > data [1] [26] ! kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : R : table() > table(data) ( ) > hist(data, breaks = seq(-0.5, 9.5, 1))! kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : > mean(data) [1] 3.56 > abline(v = mean(data), col = "red") kubo ( ( 1 ) / 106 : : > var(data) [1] (SD = variance) > sd(data) [1] > sqrt(var(data)) [1] : : kubo ( ( 1 ) / 106
5 kubo2017sep16a p.5 :? :?? kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 :? :? > data.table <- table(factor(data, levels = 0:10)) > cbind(y = data.table, prob = data.table / 50) y prob kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 :? :?? () y p(y λ) = λy exp( λ) y! y! y 4! exp( λ) = e λ (e = ) kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
6 kubo2017sep16a p.6 :? :?? R > y <- 0:9 # () > prob <- dpois(y, lambda = 3.56) # > plot(y, prob, type = "b", lty = 2) > # cbind > cbind(y, prob) (λ) 3.56 Poisson distribution y prob kubo ( ( 1 ) / 106 > hist(data, seq(-0.5, 8.5, 0.5)) # > lines(y, prob, type = "b", lty = 2) # kubo ( ( 1 ) / 106 :? :? λ? λ λ λ 0 : λ = = > # cbind > cbind(y, prob) y prob y {0, 1, 2,, } y 1 p(y λ) = 1 y=0 kubo ( ( 1 ) / 106 : y i {0, 1, 2, } y i : kubo ( ( 1 ) / 106 :? : λ p(y λ) = λy exp( λ) y! λ kubo ( ( 1 ) / 106!? kubo ( ( 1 ) / 106
7 kubo2017sep16a p.7 : (likelihood)? : L(λ) λ λ λ 3 {y 1, y 2, y 3 } = {2, 2, 4} = (the likelihood definition for the example): L(λ) = (y 1 2 ) (y 2 2 ) (y 50 3 ) = p(y 1 λ) p(y 2 λ) p(y 3 λ) p(y 50 λ) = p(y i λ) = λ yi exp( λ), y i i i! kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : : λ () (!) (log likelihood function) log L(λ) = i ( ) y i y i log λ λ log k k log L(λ) L(λ) λ kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : : ˆλ log L(λ) = ( i yi log λ λ y i k log k) log likelihood * ˆλ = (ML estimator): i y i/50 d log L dλ (ML estimate): ˆλ = 3.56 = 0! λ λ ˆλ ˆλ λ 50 kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
8 kubo2017sep16a p.8 : : : λ = y ˆλ = y y kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : : λ = y ˆλ = y : ( : y {0, 1, 2, 3, } y : y {0, 1, 2,, N} N y : < y < kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 : : GLMM 線形モデルの発展 階層ベイズモデル もっと自由な統計モデリングを! (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法線形モデル? (GLM) (Poisson regression) (logistic regression) (linear regression) kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
9 kubo2017sep16a p.9 GLM: GLM: i 3. GLM:? : {y i } : {x i } {f i } f i C: T: y i x i (f i = C): 50 sample (i {1, 2, 50}) (f i = T): 50 sample (i {51, 52, 100}) kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: GLM: data frame d : data: data3a.csv CSV (comma separated value) format file R : > d <- read.csv("data3a.csv") d data frame () data frame d > d y x f C C C T T > d$x [1] [9] [97] > d$y [1] [17] [97] kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: data frame d : GLM: R f > d$f [1] C C C C C C C C C C C C C C C C C C C C C C C C C [26] C C C C C C C C C C C C C C C C C C C C C C C C C [51] T T T T T T T T T T T T T T T T T T T T T T T T T [76] T T T T T T T T T T T T T T T T T T T T T T T T T Levels: C T : C T 2 > class(d) # d data.frame [1] "data.frame" > class(d$y) # y integer [1] "integer" > class(d$x) # x numeric [1] "numeric" > class(d$f) # f factor [1] "factor" kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
10 kubo2017sep16a p.10 GLM: data frame summary() GLM:! Generate Data Plots! Always! > summary(d) y x f Min. : 2.00 Min. : C:50 1st Qu.: st Qu.: T:50 Median : 8.00 Median : Mean : 7.83 Mean : rd Qu.: rd Qu.: Max. :15.00 Max. : kubo ( ( 1 ) / 106 > plot(d$x, d$y, pch = c(21, 19)[d$f]) > legend("topleft", legend = c("c", "T"), pch = c(21, 19)) kubo ( ( 1 ) / 106 GLM: GLM: GLM f (box-whisker plot) > plot(d$f, d$y) # note that d$f is factor type! GLM kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: GLM GLM: GLM GLM (GLM)??? : : e.g., β 1 + β 2 x i : () + () x i : kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
11 kubo2017sep16a p.11 GLM: GLM GLM: GLM (?) GLM : (response variable) : (explanatory variable) (linear predictor): () = (, intercept) + ( 1) ( 1) + ( 2) ( 2) : : e.g., β 1 + β 2 x i : ( 3) ( 3) + kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: GLM GLM: GLM GLM logistic R (GLM) GLM () rbinom() glm(family = binomial) : : e.g., β 1 + β 2 x i : logit yi x i rbinom() glm(family = binomial) rpois() glm(family = poisson) rnbinom() glm.nb() in library(mass) () rgamma() glm(family = gamma) rnorm() glm(family = gaussian) glm() GLM kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: GLM Go buck to the seed example y i λ i p(y i λ i ) = λyi i exp( λ i ) y i! i λ i? λ i = exp(β 1 + β 2 x i ) β 1 β 2 () x i i f i kubo ( ( 1 ) / 106 GLM:? i λi GLM λ i = exp(β 1 + β 2 x i ) {β 1, β 2} = { 2, 0.8} {β 1, β 2} = { 1, 0.4} i x i kubo ( ( 1 ) / 106
12 kubo2017sep16a p.12 GLM: GLM GLM: GLM GLM () i λ i λ i = exp(β 1 + β 2 x i ) log(λ i ) = β 1 + β 2 x i log() = : : β 1 + β 2 x i : log kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: R GLM GLM: R GLM glm() R GLM > d y x f C C C T T! > fit <- glm(y ~ x, data = d, family = poisson) kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: R GLM GLM: R GLM glm() glm() > fit <- glm(y ~ x, data = d, family = poisson) all: glm(formula = y ~ x, family = poisson, data = d) Coefficients: (Intercept) x ( z):? link : z (y)? family:? Degrees of Freedom: 99 Total (i.e. Null); Null Deviance: 89.5 Residual Deviance: 85 AIC: Residual kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
13 kubo2017sep16a p.13 GLM: R GLM GLM: R GLM glm() > summary(fit) Call: glm(formula = y ~ x, family = poisson, data = d) Deviance Residuals: Min 1Q Median 3Q Max Coefficients: Estimate Std. Error z value Pr(> z ) (Intercept) x () kubo ( ( 1 ) / 106 () β 2 (Estimate , SE ) (Estimate 1.29, SE 0.364) β p p ˆβ p 0.5 ˆβ (: ) kubo ( ( 1 ) / 106 GLM: R GLM GLM: R GLM (?) β 2 (Estimate , SE ) (Estimate 1.29, SE 0.364) β %? kubo ( ( 1 ) / 106 > fit <- glm(y ~ x, data = d, family = poisson)... Coefficients: (Intercept) x > plot(d$x, d$y, pch = c(21, 19)[d$f]) # data > xp <- seq(min(d$x), max(d$x), length = 100) > lines(xp, exp( * xp)) kubo ( ( 1 ) / 106 GLM: GLM: f i GLM + y i λ i i λ i β 3 f i p(y i λ i ) = λyi i exp( λ i ) y i! λ i = exp(β 1 + β 2 x i + β 3 d i ) d i = { 0 (f i = C ) 1 (f i = T ) kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
14 kubo2017sep16a p.14 GLM: GLM: glm(y x + f,...) > summary(glm(y ~ x + f, data = d, family = poisson))...()... Coefficients: Estimate Std. Error z value Pr(> z ) (Intercept) x ft () x + f > plot(d$x, d$y, pch = c(21, 19)[d$f]) # data > xp <- seq(min(d$x), max(d$x), length = 100) > lines(xp, exp( * xp), col = "blue", lwd = 3) # C > lines(xp, exp( * xp ), col = "red", lwd = 3) # T kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM: GLM: λi f i = C: λ i = exp( x i ) f i = T: λ i = exp( x i 0.032) = exp( x i ) exp( 0.032) x i exp( 0.032)! kubo ( ( 1 ) / 106 λi (A) (B) λ = exp(β 1 + β 2 x + ) λ = β 1 + β 2 x + x i x i kubo ( ( 1 ) / 106 GLM: GLM: GLM: y x log y x 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
15 kubo2017sep16a p.15 GLM GLM : 4. GLM N i = 8 : 8 : i y i = 3 : kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM example data: dead or alive of seeds y i (no individual difference!) kubo ( ( 1 ) / 106 GLM q q i N i y i ) p(y i q) = In this example... ( Ni y i q y i (1 q) N i y i, no individual difference q kubo ( ( 1 ) / 106 GLM : 20 {y i } q 20 q L(q {y i }) = 20 i=1 p(y i q) kubo ( ( 1 ) / 106 GLM L(q ) ˆq 20 log L(q ) = log i=1 ( Ni 20 + {y i log(q) + (N i y i ) log(1 q)} i=1 q kubo ( ( 1 ) / 106 y i )
16 kubo2017sep16a p.16 GLM (MLE) do you remember? GLM 8 y i L(q ) q log L(q ) q 0 ˆq log L(q )/ q = 0 q log likelihood ˆq = = = * ˆq = 0.46 ( ) 8 y 0.46 y y y i kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM GLM GLM GLM GLM logistic GLM : : e.g., β 1 + β 2 x i yi : logit x i kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM GLM GLM GLM N y 8 y! f i C: T: i N i = 8 y i = 3 (alive) (dead) x i data4a.csv CSV (comma separated value) format file R : > d <- read.csv("data4a.csv") or > d <- read.csv( + " d data frame () kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
17 kubo2017sep16a p.17 GLM GLM GLM GLM data frame d > summary(d) N y x f Min. :8 Min. :0.00 Min. : C:50 1st Qu.:8 1st Qu.:3.00 1st Qu.: T:50 Median :8 Median :6.00 Median : Mean :8 Mean :5.08 Mean : rd Qu.:8 3rd Qu.:8.00 3rd Qu.: Max. :8 Max. :8.00 Max. : > plot(d$x, d$y, pch = c(21, 19)[d$f]) > legend("topleft", legend = c("c", "T"), pch = c(21, 19)) yi x i? kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM GLM : N y p(y N, q) = ( ) N q y (1 q) N y y ( N ) y N y logit p(y i 8, q) q = 0.1 q = 0.8 q = y i kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM GLM (z i: e.g. z i = β 1 + β 2x i) 1 q i = logistic(z i ) = 1 + exp( z i ) > logistic <- function(z) 1 / (1 + exp(-z)) # > z <- seq(-6, 6, 0.1) > plot(z, logistic(z), type = "l") q q = 1 1+exp( z) z kubo ( ( 1 ) / 106 {β 1, β 2 } = {0, 2}(A) β 2 = 2 β 1 (B) β 1 = 0 β 2 q (A) β 2 = 2 β 1 = 2 β 1 = x β 1 = (B) β 1 = 0 β 2 = 4 β 2 = x β 2 = 1 {β 1, β 2 } x q 0 q 1 kubo ( ( 1 ) / 106
18 kubo2017sep16a p.18 GLM logit link function GLM R β 1 β 2 logistic 1 q = 1 + exp( (β 1 + β 2 x)) = logistic(β 1 + β 2 x) logit q logit(q) = log 1 q = β 1 + β 2 x logit logistic logistic logit logit is the inverse function of logistic function, vice versa y (A) f i =C x (B) > glm(cbind(y, N - y) ~ x + f, data = d, family = binomial)... Coefficients: (Intercept) x ft x kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106 GLM GLM : : yi (A) f i =C x i (B) f i =T x i (GLM) 3. MCMC 4. GLM kubo ( ( 1 ) / 106 kubo ( ( 1 ) / 106
19 kubo2017sep16b p.1 ( 2 ) GLM kubo@ees.hokudai.ac.jp : : :58 kubo ( ( 2 ) / 90 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル kubo ( ( 2 ) / 90 : (GLM) 3. MCMC () 1 MCMC MCMC: 2 MCMC GLM kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 MCMC MCMC : () 1. MCMC : () y i kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
20 kubo2017sep16b p.2 MCMC q () MCMC (MLE) () i N i y i ) p(y i q) = ( Ni y i q y i (1 q) N i y i, q L(q ) q log L(q ) q 0 ˆq log L(q )/ q = 0 q log likelihood ˆq = = = * kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 MCMC 8 y i () MCMC ˆq = 0.46 ( ) 8 y 0.46 y y y i kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 MCMC MCMC : p(q Y) = () p(y q) p(q) p(y) () p(y q): q Y () p(q): q () p(q Y): Y q () p(y): Y (q!) 1 p(y q) 2 p(q) 3 p(q Y ) 1 p(y q) 2 p(q) 3 p(q Y ) p(y q) 4 p(q)? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
21 kubo2017sep16b p.3 MCMC MCMC log likelihood log L(q) * q L(q) p(y q) L(q) q? p(q Y) Y p(y q) L(q) p(y q) q p(q) OK? ? : q q? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 MCMC MCMC MCMC: : GLM MCMC:? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 MCMC MCMC: MCMC MCMC: : MCMC () Markov chain Monte Carlo (MCMC) Metropolis Method (Metropolis method) :?? MCMC Metropolis Method kubo ( ( 2 ) / 90 MCMC log likelihood log L(q) * : q log likelihood q () kubo ( ( 2 ) / 90
22 kubo2017sep16b p.4 MCMC MCMC: MCMC MCMC: q log likelihood q: seed sruvivorship 1 q 2 q 3 (1), (2) kubo ( ( 2 ) / 90 Metropolis Method : 1 q ( q 0.3) 2 q ( q q new ) 3 q new L(q new ) L(q) L(q new ) L(q) (): q q new L(q new ) < L(q) (): r = L(q new)/l(q) q qnew 1 r q 4 2. (q = 0.01 q = 0.99 ) kubo ( ( 2 ) / 90 MCMC MCMC: MCMC MCMC: Metropolis Method q log likelihood log likelihood (MCMC) Metropolis Method kubo ( ( 2 ) / 90 q Metropolis Method ( MCMC) log likelihood ? q kubo ( ( 2 ) / 90 MCMC MCMC: MCMC MCMC: q q? q q? q MCMC MCMC?? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
23 kubo2017sep16b p.5 MCMC MCMC: MCMC MCMC: q MCMC q q? kubo ( ( 2 ) / 90 MCMC? log L(q) log likelihood * L(q) MCMC () kubo ( ( 2 ) / 90 MCMC MCMC: MCMC MCMC: MCMC q () MCMC p(q) q 95%! kubo ( ( 2 ) / 90 q Metropolis Method p(q Y) L(q) p(q) q kubo ( ( 2 ) / 90 MCMC MCMC MCMC? 2. MCMC Gibbs sampling 1 : : MCMC 2 R package : package : 3 BUGS Gibbs sampler : Gibbs sampler? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
24 kubo2017sep16b p.6 MCMC のためのソフトウェア MCMC のためのソフトウェア さまざまな MCMC アルゴリズム Gibbs sampling とは何か? MCMC アルゴリズムのひとつ いろいろな MCMC Metropolis Method: 試行錯誤で値を変化させて いく MCMC Metropolis-Hastings: その改良版 複数のパラメーターの MCMC サンプリングに使う 例: パラメーター β1 と β2 の Gibbs sampling 1 2 Gibbs sampling: 条件つき確率分布を使った MCMC β2 に何か適当な値を与える β2 の値はそのままにして その条件のもとでの β1 の MCMC sampling をする (条件つき事後分布) 3 複数の変数 (パラメーター 状態) を効率よくサンプリング an β1 の値はそのままにして その条件のもとでの β2 の MCMC sampling をする (条件つき事後分布) efficient method for sampling of parameter values をくりかえす 教科書の第 9 章の例題で説明 kubo ( 統計モデリング入門 (第 2 部) / 90 kubo ( MCMC のためのソフトウェア 図解: Gibbs sampling β2 のサンプリング BUGS 言語 (+ っぽいもの) でベイズモデルを 8 10 記述できるソフトウェア β β β step β WinBUGS 歴史を変えて さようなら? OpenBUGS 予算が足りなくて停滞? JAGS お手軽で良い どんな OS でも動く Stan いま一番の注目 今日は紹介しませんが 8 10 リンク集: β1 3 kubo ( step 3 32 / 90 便利な BUGS 汎用 Gibbs sampler たち (統計モデリング入門の第 9 章) step MCMC のためのソフトウェア β1 のサンプリング MCMC 統計モデリング入門 (第 2 部) β 統計モデリング入門 (第 2 部) えーと BUGS 言語って何? / 90 MCMC のためのソフトウェア kubo ( 統計モデリング入門 (第 2 部) / 90 MCMC のためのソフトウェア いろいろな OS で使える JAGS このベイズモデルを BUGS 言語で記述したい データ Y[i] 種子数8個のうちの生存数 二項分布 dbin(q,8) BUGS 言語コード R core team のひとり Martyn Plummer さんが開発 Just Another Gibbs Sampler C++ で実装されている R がインストールされていることが必要 生存確率 q Linux, Windows, Mac OS X バイナリ版もある 無情報事前分布 開発進行中 R からの使う: library(rjags) 矢印は手順ではなく 依存関係をあらわしている BUGS 言語: ベイズモデルを記述する言語 Spiegelhalter et al BUGS: Bayesian Using Gibbs Sampling version kubo ( 統計モデリング入門 (第 2 部) / 90 kubo ( 統計モデリング入門 (第 2 部) / 90
25 kubo2017sep16b p.7 MCMC JAGS R モデルの構造 データとパラメーターの初期値 サンプリングの詳細 Input BUGS 言語 JAGS MCMC sampling from posterior distributions 事後分布からのランダムサンプル Trace of beta[1] Iterations Trace of beta[2] Iterations Output Density of beta[1] N = 3000 Bandwidth = Density of beta[2] N = 3000 Bandwidth = kubo ( ( 2 ) / 90 MCMC R JAGS (1 / 3) library(rjags) library(r2winbugs) # to use write.model() model.bugs <- function() { for (i in 1:N.data) { Y[i] ~ dbin(q, 8) # } q ~ dunif(0.0, 1.0) # q } file.model <- "model.bug.txt" write.model(model.bugs, file.model) # # kubo ( ( 2 ) / 90 MCMC R JAGS (2 / 3) load("mcmc.rdata") # (data.rdata mcmc.rdata!!) list.data <- list(y = data, N.data = length(data)) inits <- list(q = 0.5) n.burnin < n.chain <- 3 n.thin <- 1 n.iter <- n.thin * 1000 model <- jags.model( file = file.model, data = list.data, inits = inits, n.chain = n.chain ) # kubo ( ( 2 ) / 90 MCMC R JAGS (3 / 3) # burn-in update(model, n.burnin) # burn in # post.mcmc.list post.mcmc.list <- coda.samples( model = model, variable.names = names(inits), n.iter = n.iter, thin = n.thin ) # kubo ( ( 2 ) / 90 MCMC burn in? MCMC step kubo ( ( 2 ) / 90 MCMC MCMC ˆR = MCMC ˆR = MCMC MCMC step! kubo ( ( 2 ) / 90
26 kubo2017sep16b p.8 MCMC ˆR MCMC Gibbs sampling plot(post.mcmc.list) gelman.diag(post.mcmc.list) R-hat Gelman-Rubin ˆR var ˆ + (ψ y) = W var ˆ + (ψ y) = n 1 n W + 1 n B W : variance B : variance Gelman et al Bayesian Data Analysis. Chapman & Hall/CRC Trace of q Density of q Iterations N = 1000 Bandwidth = kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90! GLM ! y i? ( 10 ) kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 (overdispersion) y i 0.5 : overdispersion : Modeling of individual difference i N i y i ) p(y i q i ) = ( Ni y i q y i i (1 q i) N i y i, q i kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
27 kubo2017sep16b p.9 Survivorship: logistic regression (GLM) q i = q(z i ) q(z) = 1/{1 + exp( z)} q(z) z i = a + r i a: r i : i () z r i > (a {r 1, r 2,, r 100 }) /! () kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 {r i } s = 1.0 s = 1.5 s = 3.0 r i ) 1 p(r i s) = ( exp r2 i 2πs 2 2s 2 p(r i s) r i r i r i kubo ( ( 2 ) / 90 : r i (A) I III I II I I y i (B) p(r i s) s = {r i} s = 3.0 r i 1 q i = 1+exp( r i) II I II I II I I I III I II I I I I I p(y i q i) y i kubo ( ( 2 ) / 90 r i r i {r i } 100 r i s = 1.0 s = 1.5 r i p(r i s) = s = πs 2 exp ( r2 i 2s 2 ) (A) (!) (B) (C) s kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
28 kubo2017sep16b p.10 r i! s = 1.0 s = 1.5 r i s = 3.0 p(r i s) = 1 2πs 2 exp ( r2 i 2s 2 kubo ( ( 2 ) / 90 ) 全データ 個体個体 3 3 のデータのデータ個体 1 のデータ個体個体 3 3 のデータのデータ個体 2 のデータ {r 1, r 2, r 3,..., r 100 } s local parameter random effects a global parameter fixed effects? kubo ( ( 2 ) / 90 {r i } s (B) (C) s = 1.0 a, s {r i } s s = 1.5 s = 3.0 s s (non-informative prior) 0 < s < 10 4 kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 a : Hierarchical and non-informative priors ( 0; 1) ( 0; 100) (logit) a a 種子 8 個のうち Y[i] が生存 生存 q[i] 全個体共通の 平均 a r[i] s hyper parameter kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
29 kubo2017sep16b p.11 JAGS R JAGS BUGS model { for (i in 1:N.data) { Y[i] ~ dbin(q[i], 8) logit(q[i]) <- a + r[i] } a ~ dnorm(0, 1.0E-4) for (i in 1:N.data) { r[i] ~ dnorm(0, tau) } tau <- 1 / (s * s) s ~ dunif(0, 1.0E+4) } 種子 8 個のうち Y[i] が生存 生存 q[i] 全個体共通の 平均 a r[i] s hyper parameter kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 JAGS > source("mcmc.list2bugs.r") # > post.bugs <- mcmc.list2bugs(post.mcmc.list) # bugs 80% 10 interval 5 0 for each 5 10 chain 1 R hat a * r[1] [2] [3] [4] [5] [6] [7] [8] [9] [10] [11] [12] [13] [14] [15] [16] [17] [18] [19] [20] [21] [22] [23] [24] [25] [26] [27] [28] [29] [30] [31] [32] [33] [34] [35] [36] [37] [38] [39] [40] [41] [42] [43] [44] [45] [46] [47] [48] [49] [50] [51] [52] [53] [54] [55] [56] [57] [58] [59] [60] [61] [62] [63] [64] [65] [66] [67] [68] [69] [70] [71] [72] [73] [74] [75] s [76] * array truncated for lack of space 3 chains, each with 4000 iterations (first 2000 discarded) a * r s medians and 80% intervals kubo ( ( 2 ) / 90 bugs post.bugs print(post.bugs, digits.summary = 3) 95% 3 chains, each with 4000 iterations (first 2000 discarded), n.thin = 2 n.sims = 3000 iterations saved mean sd 2.5% 25% 50% 75% 97.5% Rhat n.eff a s r[1] r[2] r[3] r[4] r[5] () r[99] r[100] kubo ( ( 2 ) / 90 R Trace of a Density of a post.mcmc <- to.mcmc(post.bugs) Iterations Trace of s Iterations N = 1000 Bandwidth = Density of s N = 1000 Bandwidth = matrix kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
30 kubo2017sep16b p.12 GLMM? 線形モデルの発展推定計算方法階層ベイズモデル (HBM) MCMC もっと自由な統計モデリングを! 一般化線形混合モデル (GLMM) 最尤推定法個体差 場所差といった変量効果をあつかいたい一般化線形モデル (GLM) 正規分布以外の確率分布をあつかいたい 最小二乗法線形モデル (Generalized Linear Mixed Model) 4. + GLMM local parameter GLMM kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 :? pot G pot E pot B pot H pot C pot I pot A pot D pot J pot F y i ( 100 ) (f j = C) 5 ( 50 ) (f j = T) 5 ( 50 ) kubo ( ( 2 ) / 90 pot A pot G pot J pot H pot E pot D pot C pot I pot F pot B y i ( 100 ) (f j = C) 5 ( 50 ) (f j = T) 5 ( 50 ) kubo ( ( 2 ) / 90!! I > d <- read.csv("d1.csv") > head(d) id pot f y id : {1, 2, 3,, 100} 1 1 A C 6 pot : {A, B, C, 2 2 A C 3, J} 3 3 A C 19 f : : C, 4 4 A C 5 T 5 5 A C 0 y : () 6 6 A C 19 d$y D D J B AA D G I D E I A G II A AA A A B DE E G C D E H B C A B C B C CC D EE D E EF F F F I G H H I II J J JJ J d$id plot(d$id, d$y, pch = as.character(d$pot),...)? kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90
31 kubo2017sep16b p.13? I d$y D D J B AA D G I D E I A G II A AA A A B DE E G C D E H B C A B C B C CC D EE D E EF F F F I G H H I II J J JJ J d$id A B C D E F G H I J? () plot(d$pot, d$y, col = rep(c("blue", "red"), each = 5)) random effects kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 () 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル GLM: > summary(glm(y ~ f, data = d, family = poisson))...()... Coefficients: Estimate Std. Error z value Pr(> z ) (Intercept) < 2e-16 ft e-06...()... (f)? AIC kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 GLMM:! > library(glmmml) > summary(glmmml(y ~ f, data = d, family = poisson, + cluster = id))...()... coef se(coef) z Pr(> z ) (Intercept) e-12 ft e-03...()...?? kubo ( ( 2 ) / 90,! kubo ( ( 2 ) / 90
32 kubo2017sep16b p.14 R! Use JAGS! + R GLMM library(glmmml) glmmml() library(lme4) lmer() library(nlme) nlme() () GLMM + () log log(λ i ) = a + bf i + () + () a f i b () ( σ 1, σ 2 ) σ ([0, 10 4 ] ) kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 データ各個体の種子数 Y[i] 個 ポアソン分布平均 lambda[i] 全個体共通 beta[1] 無情報事前分布 データ ( 説明変数 ) 施肥処理 F[i] 植木鉢差 rp[pot[i]] 個体差 r[i] 階層事前分布階層事前分布 s[1] 個体差の s[2] 植木鉢差の beta[2] ばらつきばらつき 無情報事前分布 ( 超事前分布 ) kubo ( ( 2 ) / 90 + BUGS code (1) model { for (i in 1:N.sample) { Y[i] ~ dpois(lambda[i]) log(lambda[i]) <- a + b * F[i] + r[i] + rp[pot[i]] } # BUGS coding f i {C, T} F[i] 0, 1 Pot[i] 1, 2,..., 10 rp[...] kubo ( ( 2 ) / 90 + BUGS code (2) # a ~ dnorm(0, 1.0E-4) # b ~ dnorm(0, 1.0E-4) # for (i in 1:N.sample) { r[i] ~ dnorm(0, tau[1]) # } for (j in 1:N.pot) { rp[j] ~ dnorm(0, tau[2]) # () } for (k in 1:N.tau) { tau[k] <- 1.0 / (sigma[k] * sigma[k]) # sigma[k] ~ dunif(0, 1.0E+4) } } kubo ( ( 2 ) / 90 ( b)? mean sd 2.5% 25% 50% 75% 97.5% Rhat n. a b sigma[1] ()... kubo ( ( 2 ) / 90
33 kubo2017sep16b p.15 () rp[j]! random effects random effects fixed effects! kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 : (GLM) 3. MCMC 4. GLM kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 kubo ( ( 2 ) / 90 階層ベイズモデル もっと自由な統計モデリングを! 線形モデルの発展 (HBM) 一般化線形混合モデル 個体差 場所差といった変量効果をあつかいたい 一般化線形モデル 正規分布以外の確率分布をあつかいたい (GLMM) 推定計算方法 MCMC 最尤推定法 (GLM) 最小二乗法 線形モデル () kubo ( ( 2 ) / 90
kubo2015ngt6 p.2 ( ( (MLE 8 y i L(q q log L(q q 0 ˆq log L(q / q = 0 q ˆq = = = * ˆq = 0.46 ( 8 y 0.46 y y y i kubo (ht
 kubo2015ngt6 p.1 2015 (6 MCMC kubo@ees.hokudai.ac.jp, @KuboBook http://goo.gl/m8hsbm 1 ( 2 3 4 5 JAGS : 2015 05 18 16:48 kubo (http://goo.gl/m8hsbm 2015 (6 1 / 70 kubo (http://goo.gl/m8hsbm 2015 (6 2 /
kubo2015ngt6 p.1 2015 (6 MCMC kubo@ees.hokudai.ac.jp, @KuboBook http://goo.gl/m8hsbm 1 ( 2 3 4 5 JAGS : 2015 05 18 16:48 kubo (http://goo.gl/m8hsbm 2015 (6 1 / 70 kubo (http://goo.gl/m8hsbm 2015 (6 2 /
kubostat2017c p (c) Poisson regression, a generalized linear model (GLM) : :
 kubostat2017c p.1 2017 (c), a generalized linear model (GLM) : kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 14 : 2017 11 07 15:43 kubostat2017c (http://goo.gl/76c4i) 2017 (c) 2017 11 14 1 / 47 agenda
kubostat2017c p.1 2017 (c), a generalized linear model (GLM) : kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 14 : 2017 11 07 15:43 kubostat2017c (http://goo.gl/76c4i) 2017 (c) 2017 11 14 1 / 47 agenda
kubostat1g p. MCMC binomial distribution q MCMC : i N i y i p(y i q = ( Ni y i q y i (1 q N i y i, q {y i } q likelihood q L(q {y i } = i=1 p(y i q 1
 kubostat1g p.1 1 (g Hierarchical Bayesian Model kubo@ees.hokudai.ac.jp http://goo.gl/7ci The development of linear models Hierarchical Bayesian Model Be more flexible Generalized Linear Mixed Model (GLMM
kubostat1g p.1 1 (g Hierarchical Bayesian Model kubo@ees.hokudai.ac.jp http://goo.gl/7ci The development of linear models Hierarchical Bayesian Model Be more flexible Generalized Linear Mixed Model (GLMM
一般化線形 (混合) モデル (2) - ロジスティック回帰と GLMM
 .. ( ) (2) GLMM kubo@ees.hokudai.ac.jp I http://goo.gl/rrhzey 2013 08 27 : 2013 08 27 08:29 kubostat2013ou2 (http://goo.gl/rrhzey) ( ) (2) 2013 08 27 1 / 74 I.1 N k.2 binomial distribution logit link function.3.4!
.. ( ) (2) GLMM kubo@ees.hokudai.ac.jp I http://goo.gl/rrhzey 2013 08 27 : 2013 08 27 08:29 kubostat2013ou2 (http://goo.gl/rrhzey) ( ) (2) 2013 08 27 1 / 74 I.1 N k.2 binomial distribution logit link function.3.4!
k2 ( :35 ) ( k2) (GLM) web web 1 :
 2012 11 01 k2 (2012-10-26 16:35 ) 1 6 2 (2012 11 01 k2) (GLM) kubo@ees.hokudai.ac.jp web http://goo.gl/wijx2 web http://goo.gl/ufq2 1 : 2 2 4 3 7 4 9 5 : 11 5.1................... 13 6 14 6.1......................
2012 11 01 k2 (2012-10-26 16:35 ) 1 6 2 (2012 11 01 k2) (GLM) kubo@ees.hokudai.ac.jp web http://goo.gl/wijx2 web http://goo.gl/ufq2 1 : 2 2 4 3 7 4 9 5 : 11 5.1................... 13 6 14 6.1......................
12/1 ( ) GLM, R MCMC, WinBUGS 12/2 ( ) WinBUGS WinBUGS 12/2 ( ) : 12/3 ( ) :? ( :51 ) 2/ 71
 2010-12-02 (2010 12 02 10 :51 ) 1/ 71 GCOE 2010-12-02 WinBUGS kubo@ees.hokudai.ac.jp http://goo.gl/bukrb 12/1 ( ) GLM, R MCMC, WinBUGS 12/2 ( ) WinBUGS WinBUGS 12/2 ( ) : 12/3 ( ) :? 2010-12-02 (2010 12
2010-12-02 (2010 12 02 10 :51 ) 1/ 71 GCOE 2010-12-02 WinBUGS kubo@ees.hokudai.ac.jp http://goo.gl/bukrb 12/1 ( ) GLM, R MCMC, WinBUGS 12/2 ( ) WinBUGS WinBUGS 12/2 ( ) : 12/3 ( ) :? 2010-12-02 (2010 12
講義のーと : データ解析のための統計モデリング. 第3回
 Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
kubostat2015e p.2 how to specify Poisson regression model, a GLM GLM how to specify model, a GLM GLM logistic probability distribution Poisson distrib
 kubostat2015e p.1 I 2015 (e) GLM kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2015 07 22 2015 07 21 16:26 kubostat2015e (http://goo.gl/76c4i) 2015 (e) 2015 07 22 1 / 42 1 N k 2 binomial distribution logit
kubostat2015e p.1 I 2015 (e) GLM kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2015 07 22 2015 07 21 16:26 kubostat2015e (http://goo.gl/76c4i) 2015 (e) 2015 07 22 1 / 42 1 N k 2 binomial distribution logit
kubostat2017e p.1 I 2017 (e) GLM logistic regression : : :02 1 N y count data or
 kubostat207e p. I 207 (e) GLM kubo@ees.hokudai.ac.jp https://goo.gl/z9ycjy 207 4 207 6:02 N y 2 binomial distribution logit link function 3 4! offset kubostat207e (https://goo.gl/z9ycjy) 207 (e) 207 4
kubostat207e p. I 207 (e) GLM kubo@ees.hokudai.ac.jp https://goo.gl/z9ycjy 207 4 207 6:02 N y 2 binomial distribution logit link function 3 4! offset kubostat207e (https://goo.gl/z9ycjy) 207 (e) 207 4
kubostat2017b p.1 agenda I 2017 (b) probability distribution and maximum likelihood estimation :
 kubostat2017b p.1 agenda I 2017 (b) probabilit distribution and maimum likelihood estimation kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 14 : 2017 11 07 15:43 1 : 2 3? 4 kubostat2017b (http://goo.gl/76c4i)
kubostat2017b p.1 agenda I 2017 (b) probabilit distribution and maimum likelihood estimation kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 14 : 2017 11 07 15:43 1 : 2 3? 4 kubostat2017b (http://goo.gl/76c4i)
講義のーと : データ解析のための統計モデリング. 第2回
 Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
kubostat7f p GLM! logistic regression as usual? N? GLM GLM doesn t work! GLM!! probabilit distribution binomial distribution : : β + β x i link functi
 kubostat7f p statistaical models appeared in the class 7 (f) kubo@eeshokudaiacjp https://googl/z9cjy 7 : 7 : The development of linear models Hierarchical Baesian Model Be more flexible Generalized Linear
kubostat7f p statistaical models appeared in the class 7 (f) kubo@eeshokudaiacjp https://googl/z9cjy 7 : 7 : The development of linear models Hierarchical Baesian Model Be more flexible Generalized Linear
60 (W30)? 1. ( ) 2. ( ) web site URL ( :41 ) 1/ 77
 60 (W30)? 1. ( ) kubo@ees.hokudai.ac.jp 2. ( ) web site URL http://goo.gl/e1cja!! 2013 03 07 (2013 03 07 17 :41 ) 1/ 77 ! : :? 2013 03 07 (2013 03 07 17 :41 ) 2/ 77 2013 03 07 (2013 03 07 17 :41 ) 3/ 77!!
60 (W30)? 1. ( ) kubo@ees.hokudai.ac.jp 2. ( ) web site URL http://goo.gl/e1cja!! 2013 03 07 (2013 03 07 17 :41 ) 1/ 77 ! : :? 2013 03 07 (2013 03 07 17 :41 ) 2/ 77 2013 03 07 (2013 03 07 17 :41 ) 3/ 77!!
kubostat2017j p.2 CSV CSV (!) d2.csv d2.csv,, 286,0,A 85,0,B 378,1,A 148,1,B ( :27 ) 10/ 51 kubostat2017j (http://goo.gl/76c4i
 kubostat2017j p.1 2017 (j) Categorical Data Analsis kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 15 : 2017 11 08 17:11 kubostat2017j (http://goo.gl/76c4i) 2017 (j) 2017 11 15 1 / 63 A B C D E F G
kubostat2017j p.1 2017 (j) Categorical Data Analsis kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2017 11 15 : 2017 11 08 17:11 kubostat2017j (http://goo.gl/76c4i) 2017 (j) 2017 11 15 1 / 63 A B C D E F G
/22 R MCMC R R MCMC? 3. Gibbs sampler : kubo/
 2006-12-09 1/22 R MCMC R 1. 2. R MCMC? 3. Gibbs sampler : kubo@ees.hokudai.ac.jp http://hosho.ees.hokudai.ac.jp/ kubo/ 2006-12-09 2/22 : ( ) : : ( ) : (?) community ( ) 2006-12-09 3/22 :? 1. ( ) 2. ( )
2006-12-09 1/22 R MCMC R 1. 2. R MCMC? 3. Gibbs sampler : kubo@ees.hokudai.ac.jp http://hosho.ees.hokudai.ac.jp/ kubo/ 2006-12-09 2/22 : ( ) : : ( ) : (?) community ( ) 2006-12-09 3/22 :? 1. ( ) 2. ( )
講義のーと : データ解析のための統計モデリング. 第5回
 Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
Title 講義のーと : データ解析のための統計モデリング Author(s) 久保, 拓弥 Issue Date 2008 Doc URL http://hdl.handle.net/2115/49477 Type learningobject Note この講義資料は, 著者のホームページ http://hosho.ees.hokudai.ac.jp/~kub ードできます Note(URL)http://hosho.ees.hokudai.ac.jp/~kubo/ce/EesLecture20
k3 ( :07 ) 2 (A) k = 1 (B) k = 7 y x x 1 (k2)?? x y (A) GLM (k
 2012 11 01 k3 (2012-10-24 14:07 ) 1 6 3 (2012 11 01 k3) kubo@ees.hokudai.ac.jp web http://goo.gl/wijx2 web http://goo.gl/ufq2 1 3 2 : 4 3 AIC 6 4 7 5 8 6 : 9 7 11 8 12 8.1 (1)........ 13 8.2 (2) χ 2....................
2012 11 01 k3 (2012-10-24 14:07 ) 1 6 3 (2012 11 01 k3) kubo@ees.hokudai.ac.jp web http://goo.gl/wijx2 web http://goo.gl/ufq2 1 3 2 : 4 3 AIC 6 4 7 5 8 6 : 9 7 11 8 12 8.1 (1)........ 13 8.2 (2) χ 2....................
/ *1 *1 c Mike Gonzalez, October 14, Wikimedia Commons.
 2010 05 22 1/ 35 2010 2010 05 22 *1 kubo@ees.hokudai.ac.jp *1 c Mike Gonzalez, October 14, 2007. Wikimedia Commons. 2010 05 22 2/ 35 1. 2. 3. 2010 05 22 3/ 35 : 1.? 2. 2010 05 22 4/ 35 1. 2010 05 22 5/
2010 05 22 1/ 35 2010 2010 05 22 *1 kubo@ees.hokudai.ac.jp *1 c Mike Gonzalez, October 14, 2007. Wikimedia Commons. 2010 05 22 2/ 35 1. 2. 3. 2010 05 22 3/ 35 : 1.? 2. 2010 05 22 4/ 35 1. 2010 05 22 5/
kubostat2018d p.2 :? bod size x and fertilization f change seed number? : a statistical model for this example? i response variable seed number : { i
 kubostat2018d p.1 I 2018 (d) model selection and kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2018 06 25 : 2018 06 21 17:45 1 2 3 4 :? AIC : deviance model selection misunderstanding kubostat2018d (http://goo.gl/76c4i)
kubostat2018d p.1 I 2018 (d) model selection and kubo@ees.hokudai.ac.jp http://goo.gl/76c4i 2018 06 25 : 2018 06 21 17:45 1 2 3 4 :? AIC : deviance model selection misunderstanding kubostat2018d (http://goo.gl/76c4i)
(2/24) : 1. R R R
 R? http://hosho.ees.hokudai.ac.jp/ kubo/ce/2004/ : kubo@ees.hokudai.ac.jp (2/24) : 1. R 2. 3. R R (3/24)? 1. ( ) 2. ( I ) : (p ) : cf. (power) p? (4/24) p ( ) I p ( ) I? ( ) (5/24)? 0 2 4 6 8 A B A B (control)
R? http://hosho.ees.hokudai.ac.jp/ kubo/ce/2004/ : kubo@ees.hokudai.ac.jp (2/24) : 1. R 2. 3. R R (3/24)? 1. ( ) 2. ( I ) : (p ) : cf. (power) p? (4/24) p ( ) I p ( ) I? ( ) (5/24)? 0 2 4 6 8 A B A B (control)
1 15 R Part : website:
 1 15 R Part 4 2017 7 24 4 : website: email: http://www3.u-toyama.ac.jp/kkarato/ kkarato@eco.u-toyama.ac.jp 1 2 2 3 2.1............................... 3 2.2 2................................. 4 2.3................................
1 15 R Part 4 2017 7 24 4 : website: email: http://www3.u-toyama.ac.jp/kkarato/ kkarato@eco.u-toyama.ac.jp 1 2 2 3 2.1............................... 3 2.2 2................................. 4 2.3................................
今回 次回の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ Danger!! (危 1) 時系列データの GLM あてはめ (危 2) 時系列Yt 時系列 Xt 各時刻の個体数 気温 とか これは次回)
 生態学の時系列データ解析でよく見る あぶない モデリング 久保拓弥 mailto:kubo@ees.hokudai.ac.jp statistical model for time-series data 2017-07-03 kubostat2017 (h) 1/59 今回 次回の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ Danger!! (危 1) 時系列データの
生態学の時系列データ解析でよく見る あぶない モデリング 久保拓弥 mailto:kubo@ees.hokudai.ac.jp statistical model for time-series data 2017-07-03 kubostat2017 (h) 1/59 今回 次回の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ Danger!! (危 1) 時系列データの
/ 60 : 1. GLM? 2. A: (pwer functin) x y?
 2009-03-17 1/ 60 (2009-03-17) GLM 1. GLM :, link,, deviance (20 ) 2. GLM : (60 ) 3. GLM ( ): ffset (40 ) http://hsh.ees.hkudai.ac.jp/ kub/ce/ecsj2009.html 2009-03-17 2/ 60 : 1. GLM? 2. A: (pwer functin)
2009-03-17 1/ 60 (2009-03-17) GLM 1. GLM :, link,, deviance (20 ) 2. GLM : (60 ) 3. GLM ( ): ffset (40 ) http://hsh.ees.hkudai.ac.jp/ kub/ce/ecsj2009.html 2009-03-17 2/ 60 : 1. GLM? 2. A: (pwer functin)
,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i
![,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i ,, Poisson 3 3. t t y,, y n Nµ, σ 2 y i µ + ɛ i ɛ i N0, σ 2 E[y i ] µ * i y i x i y i α + βx i + ɛ i ɛ i N0, σ 2, α, β *3 y i E[y i ] α + βx i](/thumbs/87/95421301.jpg) Armitage.? SAS.2 µ, µ 2, µ 3 a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 µ, µ 2, µ 3 log a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 * 2 2. y t y y y Poisson y * ,, Poisson 3 3. t t y,, y n Nµ,
Armitage.? SAS.2 µ, µ 2, µ 3 a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 µ, µ 2, µ 3 log a, a 2, a 3 a µ + a 2 µ 2 + a 3 µ 3 µ, µ 2, µ 3 * 2 2. y t y y y Poisson y * ,, Poisson 3 3. t t y,, y n Nµ,
(lm) lm AIC 2 / 1
 W707 s-taiji@is.titech.ac.jp 1 / 1 (lm) lm AIC 2 / 1 : y = β 1 x 1 + β 2 x 2 + + β d x d + β d+1 + ϵ (ϵ N(0, σ 2 )) y R: x R d : β i (i = 1,..., d):, β d+1 : ( ) (d = 1) y = β 1 x 1 + β 2 + ϵ (d > 1) y
W707 s-taiji@is.titech.ac.jp 1 / 1 (lm) lm AIC 2 / 1 : y = β 1 x 1 + β 2 x 2 + + β d x d + β d+1 + ϵ (ϵ N(0, σ 2 )) y R: x R d : β i (i = 1,..., d):, β d+1 : ( ) (d = 1) y = β 1 x 1 + β 2 + ϵ (d > 1) y
kubostat2018a p.1 統計モデリング入門 2018 (a) The main language of this class is 生物多様性学特論 Japanese Sorry An overview: Statistical Modeling 観測されたパターンを説明する統計モデル
 p.1 統計モデリング入門 2018 (a) The main language of this class is 生物多様性学特論 Japanese Sorry An overview: Statistical Modeling 観測されたパターンを説明する統計モデル 久保拓弥 (北海道大 環境科学) Why in Japanese? because even in Japanese, statistics
p.1 統計モデリング入門 2018 (a) The main language of this class is 生物多様性学特論 Japanese Sorry An overview: Statistical Modeling 観測されたパターンを説明する統計モデル 久保拓弥 (北海道大 環境科学) Why in Japanese? because even in Japanese, statistics
統計モデリング入門 2018 (a) 生物多様性学特論 An overview: Statistical Modeling 観測されたパターンを説明する統計モデル 久保拓弥 (北海道大 環境科学) 統計モデリング入門 2018a 1
 統計モデリング入門 2018 (a) 生物多様性学特論 An overview: Statistical Modeling 観測されたパターンを説明する統計モデル 久保拓弥 (北海道大 環境科学) kubo@ees.hokudai.ac.jp 1/56 The main language of this class is Japanese Sorry Why in Japanese? because
統計モデリング入門 2018 (a) 生物多様性学特論 An overview: Statistical Modeling 観測されたパターンを説明する統計モデル 久保拓弥 (北海道大 環境科学) kubo@ees.hokudai.ac.jp 1/56 The main language of this class is Japanese Sorry Why in Japanese? because
今日の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ (危 1) 時系列データの GLM あてはめ (危 2) 時系列Yt 時系列 Xt 各時刻の個体数 気温 とか
 時系列データ解析でよく見る あぶない モデリング 久保拓弥 (北海道大 環境科学) 1/56 今日の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ (危 1) 時系列データの GLM あてはめ (危 2) 時系列Yt 時系列 Xt 各時刻の個体数 気温 とか (危 1) 時系列データを GLM で (危 2) 時系列Yt 時系列 Xt 相関は因果関係ではない 問題の一部
時系列データ解析でよく見る あぶない モデリング 久保拓弥 (北海道大 環境科学) 1/56 今日の要点 あぶない 時系列データ解析は やめましょう! 統計モデル のあてはめ (危 1) 時系列データの GLM あてはめ (危 2) 時系列Yt 時系列 Xt 各時刻の個体数 気温 とか (危 1) 時系列データを GLM で (危 2) 時系列Yt 時系列 Xt 相関は因果関係ではない 問題の一部
untitled
 MCMC 2004 23 1 I. MCMC 1. 2. 3. 4. MH 5. 6. MCMC 2 II. 1. 2. 3. 4. 5. 3 I. MCMC 1. 2. 3. 4. MH 5. 4 1. MCMC 5 2. A P (A) : P (A)=0.02 A B A B Pr B A) Pr B A c Pr B A)=0.8, Pr B A c =0.1 6 B A 7 8 A, :
MCMC 2004 23 1 I. MCMC 1. 2. 3. 4. MH 5. 6. MCMC 2 II. 1. 2. 3. 4. 5. 3 I. MCMC 1. 2. 3. 4. MH 5. 4 1. MCMC 5 2. A P (A) : P (A)=0.02 A B A B Pr B A) Pr B A c Pr B A)=0.8, Pr B A c =0.1 6 B A 7 8 A, :
: Bradley-Terry Burczyk
 58 (W15) 2011 03 09 kubo@ees.hokudai.ac.jp http://goo.gl/edzle 2011 03 09 (2011 03 09 19 :32 ) : Bradley-Terry Burczyk ? ( ) 1999 2010 9 R : 7 (1) 8 7??! 15 http://www.atmarkit.co.jp/fcoding/articles/stat/07/stat07a.html
58 (W15) 2011 03 09 kubo@ees.hokudai.ac.jp http://goo.gl/edzle 2011 03 09 (2011 03 09 19 :32 ) : Bradley-Terry Burczyk ? ( ) 1999 2010 9 R : 7 (1) 8 7??! 15 http://www.atmarkit.co.jp/fcoding/articles/stat/07/stat07a.html
Use R
 Use R! 2008/05/23( ) Index Introduction (GLM) ( ) R. Introduction R,, PLS,,, etc. 2. Correlation coefficient (Pearson s product moment correlation) r = Sxy Sxx Syy :, Sxy, Sxx= X, Syy Y 1.96 95% R cor(x,
Use R! 2008/05/23( ) Index Introduction (GLM) ( ) R. Introduction R,, PLS,,, etc. 2. Correlation coefficient (Pearson s product moment correlation) r = Sxy Sxx Syy :, Sxy, Sxx= X, Syy Y 1.96 95% R cor(x,
80 X 1, X 2,, X n ( λ ) λ P(X = x) = f (x; λ) = λx e λ, x = 0, 1, 2, x! l(λ) = n f (x i ; λ) = i=1 i=1 n λ x i e λ i=1 x i! = λ n i=1 x i e nλ n i=1 x
 80 X 1, X 2,, X n ( λ ) λ P(X = x) = f (x; λ) = λx e λ, x = 0, 1, 2, x! l(λ) = n f (x i ; λ) = n λ x i e λ x i! = λ n x i e nλ n x i! n n log l(λ) = log(λ) x i nλ log( x i!) log l(λ) λ = 1 λ n x i n =
80 X 1, X 2,, X n ( λ ) λ P(X = x) = f (x; λ) = λx e λ, x = 0, 1, 2, x! l(λ) = n f (x i ; λ) = n λ x i e λ x i! = λ n x i e nλ n x i! n n log l(λ) = log(λ) x i nλ log( x i!) log l(λ) λ = 1 λ n x i n =
/ 55 2 : : (GLM) 1. 1/23 ( )? GLM? (GLM ) 2.! 1/25 ( ) ffset (GLM )
 2012 01 25 1/ 55 ( II) : (2012 1 ) 2 2 (GLM) 2012 01 25! kub@ees.hkudai.ac.jp http://g.gl/76c4i 2012 01 25 2/ 55 2 : : (GLM) 1. 1/23 ( )? GLM? (GLM ) 2.! 1/25 ( ) ffset (GLM ) 2012 01 25 3/ 55 1. : 2.
2012 01 25 1/ 55 ( II) : (2012 1 ) 2 2 (GLM) 2012 01 25! kub@ees.hkudai.ac.jp http://g.gl/76c4i 2012 01 25 2/ 55 2 : : (GLM) 1. 1/23 ( )? GLM? (GLM ) 2.! 1/25 ( ) ffset (GLM ) 2012 01 25 3/ 55 1. : 2.
: (GLMM) (pseudo replication) ( ) ( ) & Markov Chain Monte Carlo (MCMC)? /30
 PlotNet 6 ( ) 2006-01-19 TOEF(1998 2004), AM, growth6 DBH growth (mm) 1998 1999 2000 2001 2002 2003 2004 10 20 30 40 50 70 DBH (cm) 1. 2. - - : kubo@ees.hokudai.ac.jp http://hosho.ees.hokudai.ac.jp/ kubo/show/2006/plotnet/
PlotNet 6 ( ) 2006-01-19 TOEF(1998 2004), AM, growth6 DBH growth (mm) 1998 1999 2000 2001 2002 2003 2004 10 20 30 40 50 70 DBH (cm) 1. 2. - - : kubo@ees.hokudai.ac.jp http://hosho.ees.hokudai.ac.jp/ kubo/show/2006/plotnet/
DAA09
 > summary(dat.lm1) Call: lm(formula = sales ~ price, data = dat) Residuals: Min 1Q Median 3Q Max -55.719-19.270 4.212 16.143 73.454 Coefficients: Estimate Std. Error t value Pr(> t ) (Intercept) 237.1326
> summary(dat.lm1) Call: lm(formula = sales ~ price, data = dat) Residuals: Min 1Q Median 3Q Max -55.719-19.270 4.212 16.143 73.454 Coefficients: Estimate Std. Error t value Pr(> t ) (Intercept) 237.1326
最小2乗法
 2 2012 4 ( ) 2 2012 4 1 / 42 X Y Y = f (X ; Z) linear regression model X Y slope X 1 Y (X, Y ) 1 (X, Y ) ( ) 2 2012 4 2 / 42 1 β = β = β (4.2) = β 0 + β (4.3) ( ) 2 2012 4 3 / 42 = β 0 + β + (4.4) ( )
2 2012 4 ( ) 2 2012 4 1 / 42 X Y Y = f (X ; Z) linear regression model X Y slope X 1 Y (X, Y ) 1 (X, Y ) ( ) 2 2012 4 2 / 42 1 β = β = β (4.2) = β 0 + β (4.3) ( ) 2 2012 4 3 / 42 = β 0 + β + (4.4) ( )
1 環境統計学ぷらす 第 5 回 一般 ( 化 ) 線形混合モデル 高木俊 2013/11/21
 1 環境統計学ぷらす 第 5 回 一般 ( 化 ) 線形混合モデル 高木俊 shun.takagi@sci.toho-u.ac.jp 2013/11/21 2 予定 第 1 回 : Rの基礎と仮説検定 第 2 回 : 分散分析と回帰 第 3 回 : 一般線形モデル 交互作用 第 4.1 回 : 一般化線形モデル 第 4.2 回 : モデル選択 (11/29?) 第 5 回 : 一般化線形混合モデル
1 環境統計学ぷらす 第 5 回 一般 ( 化 ) 線形混合モデル 高木俊 shun.takagi@sci.toho-u.ac.jp 2013/11/21 2 予定 第 1 回 : Rの基礎と仮説検定 第 2 回 : 分散分析と回帰 第 3 回 : 一般線形モデル 交互作用 第 4.1 回 : 一般化線形モデル 第 4.2 回 : モデル選択 (11/29?) 第 5 回 : 一般化線形混合モデル
201711grade2.pdf
 2017 11 26 1 2 28 3 90 4 5 A 1 2 3 4 Web Web 6 B 10 3 10 3 7 34 8 23 9 10 1 2 3 1 (A) 3 32.14 0.65 2.82 0.93 7.48 (B) 4 6 61.30 54.68 34.86 5.25 19.07 (C) 7 13 5.89 42.18 56.51 35.80 50.28 (D) 14 20 0.35
2017 11 26 1 2 28 3 90 4 5 A 1 2 3 4 Web Web 6 B 10 3 10 3 7 34 8 23 9 10 1 2 3 1 (A) 3 32.14 0.65 2.82 0.93 7.48 (B) 4 6 61.30 54.68 34.86 5.25 19.07 (C) 7 13 5.89 42.18 56.51 35.80 50.28 (D) 14 20 0.35
Rによる計量分析:データ解析と可視化 - 第3回 Rの基礎とデータ操作・管理
 R 3 R 2017 Email: gito@eco.u-toyama.ac.jp October 23, 2017 (Toyama/NIHU) R ( 3 ) October 23, 2017 1 / 34 Agenda 1 2 3 4 R 5 RStudio (Toyama/NIHU) R ( 3 ) October 23, 2017 2 / 34 10/30 (Mon.) 12/11 (Mon.)
R 3 R 2017 Email: gito@eco.u-toyama.ac.jp October 23, 2017 (Toyama/NIHU) R ( 3 ) October 23, 2017 1 / 34 Agenda 1 2 3 4 R 5 RStudio (Toyama/NIHU) R ( 3 ) October 23, 2017 2 / 34 10/30 (Mon.) 12/11 (Mon.)
2014ESJ.key
 http://www001.upp.so-net.ne.jp/ito-hi/stat/2014esj/ Statistical Software for State Space Models Commandeur et al. (2011) Journal of Statistical Software 41(1) State Space Models in R Petris & Petrone (2011)
http://www001.upp.so-net.ne.jp/ito-hi/stat/2014esj/ Statistical Software for State Space Models Commandeur et al. (2011) Journal of Statistical Software 41(1) State Space Models in R Petris & Petrone (2011)
yamadaiR(cEFA).pdf
 R 2012/10/05 Kosugi,E.Koji (Yamadai.R) Categorical Factor Analysis by using R 2012/10/05 1 / 9 Why we use... 3 5 Kosugi,E.Koji (Yamadai.R) Categorical Factor Analysis by using R 2012/10/05 2 / 9 FA vs
R 2012/10/05 Kosugi,E.Koji (Yamadai.R) Categorical Factor Analysis by using R 2012/10/05 1 / 9 Why we use... 3 5 Kosugi,E.Koji (Yamadai.R) Categorical Factor Analysis by using R 2012/10/05 2 / 9 FA vs
X X X Y R Y R Y R MCAR MAR MNAR Figure 1: MCAR, MAR, MNAR Y R X 1.2 Missing At Random (MAR) MAR MCAR MCAR Y X X Y MCAR 2 1 R X Y Table 1 3 IQ MCAR Y I
 (missing data analysis) - - 1/16/2011 (missing data, missing value) (list-wise deletion) (pair-wise deletion) (full information maximum likelihood method, FIML) (multiple imputation method) 1 missing completely
(missing data analysis) - - 1/16/2011 (missing data, missing value) (list-wise deletion) (pair-wise deletion) (full information maximum likelihood method, FIML) (multiple imputation method) 1 missing completely
 2009 5 1...1 2...3 2.1...3 2.2...3 3...10 3.1...10 3.1.1...10 3.1.2... 11 3.2...14 3.2.1...14 3.2.2...16 3.3...18 3.4...19 3.4.1...19 3.4.2...20 3.4.3...21 4...24 4.1...24 4.2...24 4.3 WinBUGS...25 4.4...28
2009 5 1...1 2...3 2.1...3 2.2...3 3...10 3.1...10 3.1.1...10 3.1.2... 11 3.2...14 3.2.1...14 3.2.2...16 3.3...18 3.4...19 3.4.1...19 3.4.2...20 3.4.3...21 4...24 4.1...24 4.2...24 4.3 WinBUGS...25 4.4...28
AR(1) y t = φy t 1 + ɛ t, ɛ t N(0, σ 2 ) 1. Mean of y t given y t 1, y t 2, E(y t y t 1, y t 2, ) = φy t 1 2. Variance of y t given y t 1, y t
 87 6.1 AR(1) y t = φy t 1 + ɛ t, ɛ t N(0, σ 2 ) 1. Mean of y t given y t 1, y t 2, E(y t y t 1, y t 2, ) = φy t 1 2. Variance of y t given y t 1, y t 2, V(y t y t 1, y t 2, ) = σ 2 3. Thus, y t y t 1,
87 6.1 AR(1) y t = φy t 1 + ɛ t, ɛ t N(0, σ 2 ) 1. Mean of y t given y t 1, y t 2, E(y t y t 1, y t 2, ) = φy t 1 2. Variance of y t given y t 1, y t 2, V(y t y t 1, y t 2, ) = σ 2 3. Thus, y t y t 1,
スライド 1
 WinBUGS 入門 水産資源学におけるベイズ統計の応用ワークショップ 2007 年 8 月 2-3 日, 中央水研 遠洋水産研究所外洋資源部 鯨類管理研究室 岡村寛 WinBUGS とは BUGS (Bayesian Inference Using Gibbs Sampling) の Windows バージョン フリーのソフトウェア Gibbs samplingを利用した事後確率からのサンプリングを行う
WinBUGS 入門 水産資源学におけるベイズ統計の応用ワークショップ 2007 年 8 月 2-3 日, 中央水研 遠洋水産研究所外洋資源部 鯨類管理研究室 岡村寛 WinBUGS とは BUGS (Bayesian Inference Using Gibbs Sampling) の Windows バージョン フリーのソフトウェア Gibbs samplingを利用した事後確率からのサンプリングを行う
Microsoft Word - 計量研修テキスト_第5版).doc
 Q10-2 テキスト P191 1. 記述統計量 ( 変数 :YY95) 表示変数として 平均 中央値 最大値 最小値 標準偏差 観測値 を選択 A. 都道府県別 Descriptive Statistics for YY95 Categorized by values of PREFNUM Date: 05/11/06 Time: 14:36 Sample: 1990 2002 Included
Q10-2 テキスト P191 1. 記述統計量 ( 変数 :YY95) 表示変数として 平均 中央値 最大値 最小値 標準偏差 観測値 を選択 A. 都道府県別 Descriptive Statistics for YY95 Categorized by values of PREFNUM Date: 05/11/06 Time: 14:36 Sample: 1990 2002 Included
第11回:線形回帰モデルのOLS推定
 11 OLS 2018 7 13 1 / 45 1. 2. 3. 2 / 45 n 2 ((y 1, x 1 ), (y 2, x 2 ),, (y n, x n )) linear regression model y i = β 0 + β 1 x i + u i, E(u i x i ) = 0, E(u i u j x i ) = 0 (i j), V(u i x i ) = σ 2, i
11 OLS 2018 7 13 1 / 45 1. 2. 3. 2 / 45 n 2 ((y 1, x 1 ), (y 2, x 2 ),, (y n, x n )) linear regression model y i = β 0 + β 1 x i + u i, E(u i x i ) = 0, E(u i u j x i ) = 0 (i j), V(u i x i ) = σ 2, i
y i OLS [0, 1] OLS x i = (1, x 1,i,, x k,i ) β = (β 0, β 1,, β k ) G ( x i β) 1 G i 1 π i π i P {y i = 1 x i } = G (
![y i OLS [0, 1] OLS x i = (1, x 1,i,, x k,i ) β = (β 0, β 1,, β k ) G ( x i β) 1 G i 1 π i π i P {y i = 1 x i } = G ( y i OLS [0, 1] OLS x i = (1, x 1,i,, x k,i ) β = (β 0, β 1,, β k ) G ( x i β) 1 G i 1 π i π i P {y i = 1 x i } = G (](/thumbs/82/85163103.jpg) 7 2 2008 7 10 1 2 2 1.1 2............................................. 2 1.2 2.......................................... 2 1.3 2........................................ 3 1.4................................................
7 2 2008 7 10 1 2 2 1.1 2............................................. 2 1.2 2.......................................... 2 1.3 2........................................ 3 1.4................................................
²¾ÁÛ¾õ¶·É¾²ÁË¡¤Î¤¿¤á¤Î¥Ñ¥Ã¥±¡¼¥¸DCchoice ¡Ê»ÃÄêÈÇ¡Ë
 DCchoice ( ) R 2013 2013 11 30 DCchoice package R 2013/11/30 1 / 19 1 (CV) CV 2 DCchoice WTP 3 DCchoice package R 2013/11/30 2 / 19 (Contingent Valuation; CV) WTP CV WTP WTP 1 1989 2 DCchoice package R
DCchoice ( ) R 2013 2013 11 30 DCchoice package R 2013/11/30 1 / 19 1 (CV) CV 2 DCchoice WTP 3 DCchoice package R 2013/11/30 2 / 19 (Contingent Valuation; CV) WTP CV WTP WTP 1 1989 2 DCchoice package R
Stata11 whitepapers mwp-037 regress - regress regress. regress mpg weight foreign Source SS df MS Number of obs = 74 F(
 mwp-037 regress - regress 1. 1.1 1.2 1.3 2. 3. 4. 5. 1. regress. regress mpg weight foreign Source SS df MS Number of obs = 74 F( 2, 71) = 69.75 Model 1619.2877 2 809.643849 Prob > F = 0.0000 Residual
mwp-037 regress - regress 1. 1.1 1.2 1.3 2. 3. 4. 5. 1. regress. regress mpg weight foreign Source SS df MS Number of obs = 74 F( 2, 71) = 69.75 Model 1619.2877 2 809.643849 Prob > F = 0.0000 Residual
日心TWS
 2017.09.22 (15:40~17:10) 日本心理学会第 81 回大会 TWS ベイジアンデータ解析入門 回帰分析を例に ベイジアンデータ解析 を体験してみる 広島大学大学院教育学研究科平川真 ベイジアン分析のステップ (p.24) 1) データの特定 2) モデルの定義 ( 解釈可能な ) モデルの作成 3) パラメタの事前分布の設定 4) ベイズ推論を用いて パラメタの値に確信度を再配分ベイズ推定
2017.09.22 (15:40~17:10) 日本心理学会第 81 回大会 TWS ベイジアンデータ解析入門 回帰分析を例に ベイジアンデータ解析 を体験してみる 広島大学大学院教育学研究科平川真 ベイジアン分析のステップ (p.24) 1) データの特定 2) モデルの定義 ( 解釈可能な ) モデルの作成 3) パラメタの事前分布の設定 4) ベイズ推論を用いて パラメタの値に確信度を再配分ベイズ推定
% 10%, 35%( 1029 ) p (a) 1 p 95% (b) 1 Std. Err. (c) p 40% 5% (d) p 1: STATA (1). prtesti One-sample test of pr
 1 1. 2014 6 2014 6 10 10% 10%, 35%( 1029 ) p (a) 1 p 95% (b) 1 Std. Err. (c) p 40% 5% (d) p 1: STATA (1). prtesti 1029 0.35 0.40 One-sample test of proportion x: Number of obs = 1029 Variable Mean Std.
1 1. 2014 6 2014 6 10 10% 10%, 35%( 1029 ) p (a) 1 p 95% (b) 1 Std. Err. (c) p 40% 5% (d) p 1: STATA (1). prtesti 1029 0.35 0.40 One-sample test of proportion x: Number of obs = 1029 Variable Mean Std.
1 Stata SEM LightStone 4 SEM 4.. Alan C. Acock, Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press 3.
 1 Stata SEM LightStone 4 SEM 4.. Alan C. Acock, 2013. Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press 3. 2 4, 2. 1 2 2 Depress Conservative. 3., 3,. SES66 Alien67 Alien71,
1 Stata SEM LightStone 4 SEM 4.. Alan C. Acock, 2013. Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press 3. 2 4, 2. 1 2 2 Depress Conservative. 3., 3,. SES66 Alien67 Alien71,
Dirichlet process mixture Dirichlet process mixture 2 /40 MIRU2008 :
 Dirichlet Process : joint work with: Max Welling (UC Irvine), Yee Whye Teh (UCL, Gatsby) http://kenichi.kurihara.googlepages.com/miru_workshop.pdf 1 /40 MIRU2008 : Dirichlet process mixture Dirichlet process
Dirichlet Process : joint work with: Max Welling (UC Irvine), Yee Whye Teh (UCL, Gatsby) http://kenichi.kurihara.googlepages.com/miru_workshop.pdf 1 /40 MIRU2008 : Dirichlet process mixture Dirichlet process
バイオインフォマティクス特論4
 藤 博幸 1-3-1. ピアソン相関係数 1-3-2. 致性のカッパ係数 1-3-3. 時系列データにおける変化検出 ベイズ統計で実践モデリング 5.1 ピアソン係数 第 5 章データ解析の例 データは n ペアの独 な観測値の対例 : 特定の薬剤の投与量と投与から t 時間後の注 する遺伝 の発現量 2 つの変数間の線形の関係性はピアソンの積率相関係数 r で表現される t 時間後の注 する遺伝
藤 博幸 1-3-1. ピアソン相関係数 1-3-2. 致性のカッパ係数 1-3-3. 時系列データにおける変化検出 ベイズ統計で実践モデリング 5.1 ピアソン係数 第 5 章データ解析の例 データは n ペアの独 な観測値の対例 : 特定の薬剤の投与量と投与から t 時間後の注 する遺伝 の発現量 2 つの変数間の線形の関係性はピアソンの積率相関係数 r で表現される t 時間後の注 する遺伝
ECCS. ECCS,. ( 2. Mac Do-file Editor. Mac Do-file Editor Windows Do-file Editor Top Do-file e
 1 1 2015 4 6 1. ECCS. ECCS,. (https://ras.ecc.u-tokyo.ac.jp/guacamole/) 2. Mac Do-file Editor. Mac Do-file Editor Windows Do-file Editor Top Do-file editor, Do View Do-file Editor Execute(do). 3. Mac System
1 1 2015 4 6 1. ECCS. ECCS,. (https://ras.ecc.u-tokyo.ac.jp/guacamole/) 2. Mac Do-file Editor. Mac Do-file Editor Windows Do-file Editor Top Do-file editor, Do View Do-file Editor Execute(do). 3. Mac System
みっちりGLM
 2015/3/27 12:00-13:00 日本草地学会若手 R 統計企画 ( 信州大学農学部 ) R と一般化線形モデル入門 山梨県富士山科学研究所 安田泰輔 謝辞 : 日本草地学会若手の会の皆様 発表の機会を頂き たいへんありがとうございます! 茨城大学 学生時代 自己紹介 ベータ二項分布を用いた種の空間分布の解析 所属 : 山梨県富士山科学研究所 最近の研究テーマ 近接リモートセンシングによる半自然草地のモニタリング手法開発
2015/3/27 12:00-13:00 日本草地学会若手 R 統計企画 ( 信州大学農学部 ) R と一般化線形モデル入門 山梨県富士山科学研究所 安田泰輔 謝辞 : 日本草地学会若手の会の皆様 発表の機会を頂き たいへんありがとうございます! 茨城大学 学生時代 自己紹介 ベータ二項分布を用いた種の空間分布の解析 所属 : 山梨県富士山科学研究所 最近の研究テーマ 近接リモートセンシングによる半自然草地のモニタリング手法開発
28
 y i = Z i δ i +ε i ε i δ X y i = X Z i δ i + X ε i [ ] 1 δ ˆ i = Z i X( X X) 1 X Z i [ ] 1 σ ˆ 2 Z i X( X X) 1 X Z i Z i X( X X) 1 X y i σ ˆ 2 ˆ σ 2 = [ ] y i Z ˆ [ i δ i ] 1 y N p i Z i δ ˆ i i RSTAT
y i = Z i δ i +ε i ε i δ X y i = X Z i δ i + X ε i [ ] 1 δ ˆ i = Z i X( X X) 1 X Z i [ ] 1 σ ˆ 2 Z i X( X X) 1 X Z i Z i X( X X) 1 X y i σ ˆ 2 ˆ σ 2 = [ ] y i Z ˆ [ i δ i ] 1 y N p i Z i δ ˆ i i RSTAT
¥¤¥ó¥¿¡¼¥Í¥Ã¥È·×¬¤È¥Ç¡¼¥¿²òÀÏ Âè2²ó
 2 2015 4 20 1 (4/13) : ruby 2 / 49 2 ( ) : gnuplot 3 / 49 1 1 2014 6 IIJ / 4 / 49 1 ( ) / 5 / 49 ( ) 6 / 49 (summary statistics) : (mean) (median) (mode) : (range) (variance) (standard deviation) 7 / 49
2 2015 4 20 1 (4/13) : ruby 2 / 49 2 ( ) : gnuplot 3 / 49 1 1 2014 6 IIJ / 4 / 49 1 ( ) / 5 / 49 ( ) 6 / 49 (summary statistics) : (mean) (median) (mode) : (range) (variance) (standard deviation) 7 / 49
: (EQS) /EQUATIONS V1 = 30*V F1 + E1; V2 = 25*V *F1 + E2; V3 = 16*V *F1 + E3; V4 = 10*V F2 + E4; V5 = 19*V99
 218 6 219 6.11: (EQS) /EQUATIONS V1 = 30*V999 + 1F1 + E1; V2 = 25*V999 +.54*F1 + E2; V3 = 16*V999 + 1.46*F1 + E3; V4 = 10*V999 + 1F2 + E4; V5 = 19*V999 + 1.29*F2 + E5; V6 = 17*V999 + 2.22*F2 + E6; CALIS.
218 6 219 6.11: (EQS) /EQUATIONS V1 = 30*V999 + 1F1 + E1; V2 = 25*V999 +.54*F1 + E2; V3 = 16*V999 + 1.46*F1 + E3; V4 = 10*V999 + 1F2 + E4; V5 = 19*V999 + 1.29*F2 + E5; V6 = 17*V999 + 2.22*F2 + E6; CALIS.
1 R Windows R 1.1 R The R project web R web Download [CRAN] CRAN Mirrors Japan Download and Install R [Windows 9
![1 R Windows R 1.1 R The R project web R web Download [CRAN] CRAN Mirrors Japan Download and Install R [Windows 9 1 R Windows R 1.1 R The R project web R web Download [CRAN] CRAN Mirrors Japan Download and Install R [Windows 9](/thumbs/99/142039515.jpg) 1 R 2007 8 19 1 Windows R 1.1 R The R project web http://www.r-project.org/ R web Download [CRAN] CRAN Mirrors Japan Download and Install R [Windows 95 and later ] [base] 2.5.1 R - 2.5.1 for Windows R
1 R 2007 8 19 1 Windows R 1.1 R The R project web http://www.r-project.org/ R web Download [CRAN] CRAN Mirrors Japan Download and Install R [Windows 95 and later ] [base] 2.5.1 R - 2.5.1 for Windows R
1 Stata SEM LightStone 3 2 SEM. 2., 2,. Alan C. Acock, Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press.
 1 Stata SEM LightStone 3 2 SEM. 2., 2,. Alan C. Acock, 2013. Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press. 2 3 2 Conservative Depress. 3.1 2. SEM. 1. x SEM. Depress.
1 Stata SEM LightStone 3 2 SEM. 2., 2,. Alan C. Acock, 2013. Discovering Structural Equation Modeling Using Stata, Revised Edition, Stata Press. 2 3 2 Conservative Depress. 3.1 2. SEM. 1. x SEM. Depress.
バイオインフォマティクス特論12
 藤 博幸 事後予測分布 パラメータの事後分布に従って モデルがどんなデータを期待するかを予測する 予測分布が観測されたデータと 致するかを確認することで モデルの適切さを確認できる 前回と同じ問題で事後予測を う 3-1-1. 個 差を考えない場合 3-1-2. 完全な個 差を考える場合 3-1-3. 構造化された個 差を考える場合 ベイズ統計で実践モデリング 10.1 個 差を考えない場合 第 10
藤 博幸 事後予測分布 パラメータの事後分布に従って モデルがどんなデータを期待するかを予測する 予測分布が観測されたデータと 致するかを確認することで モデルの適切さを確認できる 前回と同じ問題で事後予測を う 3-1-1. 個 差を考えない場合 3-1-2. 完全な個 差を考える場合 3-1-3. 構造化された個 差を考える場合 ベイズ統計で実践モデリング 10.1 個 差を考えない場合 第 10
1 kawaguchi p.1/81
 1 kawaguchi atsushi@kurume-u.ac.jp 2005 7 2 p.1/81 2.1 2.2 2.2.3 2.3 AUC 4.4 p.2/81 X Z X = α + βz + e α : Z = 0 X ( ) β : Z X ( ) e : 0 σ 2 p.3/81 2.1 Z X 1 0.045 2 0.114 4 0.215 6 0.346 7 0.41 8 0.52
1 kawaguchi atsushi@kurume-u.ac.jp 2005 7 2 p.1/81 2.1 2.2 2.2.3 2.3 AUC 4.4 p.2/81 X Z X = α + βz + e α : Z = 0 X ( ) β : Z X ( ) e : 0 σ 2 p.3/81 2.1 Z X 1 0.045 2 0.114 4 0.215 6 0.346 7 0.41 8 0.52
こんにちは由美子です
 1 2 . sum Variable Obs Mean Std. Dev. Min Max ---------+----------------------------------------------------- var1 13.4923077.3545926.05 1.1 3 3 3 0.71 3 x 3 C 3 = 0.3579 2 1 0.71 2 x 0.29 x 3 C 2 = 0.4386
1 2 . sum Variable Obs Mean Std. Dev. Min Max ---------+----------------------------------------------------- var1 13.4923077.3545926.05 1.1 3 3 3 0.71 3 x 3 C 3 = 0.3579 2 1 0.71 2 x 0.29 x 3 C 2 = 0.4386
untitled
 2011/6/22 M2 1*1+2*2 79 2F Y YY 0.0 0.2 0.4 0.6 0.8 0.000 0.002 0.004 0.006 0.008 0.010 0.012 1.0 1.5 2.0 2.5 3.0 3.5 4.0 Y 0 50 100 150 200 250 YY A (Y = X + e A ) B (YY = X + e B ) X 0.00 0.05 0.10
2011/6/22 M2 1*1+2*2 79 2F Y YY 0.0 0.2 0.4 0.6 0.8 0.000 0.002 0.004 0.006 0.008 0.010 0.012 1.0 1.5 2.0 2.5 3.0 3.5 4.0 Y 0 50 100 150 200 250 YY A (Y = X + e A ) B (YY = X + e B ) X 0.00 0.05 0.10
H22 BioS t (i) treat1 treat2 data d1; input patno treat1 treat2; cards; ; run; 1 (i) treat = 1 treat =
 H BioS t (i) treat treat data d; input patno treat treat; cards; 3 8 7 4 8 8 5 5 6 3 ; run; (i) treat treat data d; input group patno period treat y; label group patno period ; cards; 3 8 3 7 4 8 4 8 5
H BioS t (i) treat treat data d; input patno treat treat; cards; 3 8 7 4 8 8 5 5 6 3 ; run; (i) treat treat data d; input group patno period treat y; label group patno period ; cards; 3 8 3 7 4 8 4 8 5
2 と入力すると以下のようになる > x1<-c(1.52,2,3.01,9,2,6.3,5,11.2) > y1<-c(4,0.21,-1.5,8,2,6,9.915,5.2) > cor(x1,y1) [1] > cor.test(x1,y1) Pearson's produ
![2 と入力すると以下のようになる > x1<-c(1.52,2,3.01,9,2,6.3,5,11.2) > y1<-c(4,0.21,-1.5,8,2,6,9.915,5.2) > cor(x1,y1) [1] > cor.test(x1,y1) Pearson's produ 2 と入力すると以下のようになる > x1<-c(1.52,2,3.01,9,2,6.3,5,11.2) > y1<-c(4,0.21,-1.5,8,2,6,9.915,5.2) > cor(x1,y1) [1] > cor.test(x1,y1) Pearson's produ](/thumbs/91/106146056.jpg) 1 統計 データ解析セミナーの予習 2010.11.24 粕谷英一 ( 理 生物 生態 ) GCOE アジア保全生態学 本日のメニュー R 一般化線形モデル (Generalized Linear Models 略して GLM) R で GLM を使う R でグラフを描く 説明しないこと :R でできること全般 たくさんあるので時間的に無理 R でするプログラミング-データ解析なら使いやすい R 起動と終了
1 統計 データ解析セミナーの予習 2010.11.24 粕谷英一 ( 理 生物 生態 ) GCOE アジア保全生態学 本日のメニュー R 一般化線形モデル (Generalized Linear Models 略して GLM) R で GLM を使う R でグラフを描く 説明しないこと :R でできること全般 たくさんあるので時間的に無理 R でするプログラミング-データ解析なら使いやすい R 起動と終了
こんにちは由美子です
 Analysis of Variance 2 two sample t test analysis of variance (ANOVA) CO 3 3 1 EFV1 µ 1 µ 2 µ 3 H 0 H 0 : µ 1 = µ 2 = µ 3 H A : Group 1 Group 2.. Group k population mean µ 1 µ µ κ SD σ 1 σ σ κ sample mean
Analysis of Variance 2 two sample t test analysis of variance (ANOVA) CO 3 3 1 EFV1 µ 1 µ 2 µ 3 H 0 H 0 : µ 1 = µ 2 = µ 3 H A : Group 1 Group 2.. Group k population mean µ 1 µ µ κ SD σ 1 σ σ κ sample mean
GLM PROC GLM y = Xβ + ε y X β ε ε σ 2 E[ε] = 0 var[ε] = σ 2 I σ 2 0 σ 2 =... 0 σ 2 σ 2 I ε σ 2 y E[y] =Xβ var[y] =σ 2 I PROC GLM
![GLM PROC GLM y = Xβ + ε y X β ε ε σ 2 E[ε] = 0 var[ε] = σ 2 I σ 2 0 σ 2 =... 0 σ 2 σ 2 I ε σ 2 y E[y] =Xβ var[y] =σ 2 I PROC GLM GLM PROC GLM y = Xβ + ε y X β ε ε σ 2 E[ε] = 0 var[ε] = σ 2 I σ 2 0 σ 2 =... 0 σ 2 σ 2 I ε σ 2 y E[y] =Xβ var[y] =σ 2 I PROC GLM](/thumbs/84/89087584.jpg) PROC MIXED ( ) An Introdunction to PROC MIXED Junji Kishimoto SAS Institute Japan / Keio Univ. SFC / Univ. of Tokyo e-mail address: jpnjak@jpn.sas.com PROC MIXED PROC GLM PROC MIXED,,,, 1 1.1 PROC MIXED
PROC MIXED ( ) An Introdunction to PROC MIXED Junji Kishimoto SAS Institute Japan / Keio Univ. SFC / Univ. of Tokyo e-mail address: jpnjak@jpn.sas.com PROC MIXED PROC GLM PROC MIXED,,,, 1 1.1 PROC MIXED
第13回:交差項を含む回帰・弾力性の推定
 13 2018 7 27 1 / 31 1. 2. 2 / 31 y i = β 0 + β X x i + β Z z i + β XZ x i z i + u i, E(u i x i, z i ) = 0, E(u i u j x i, z i ) = 0 (i j), V(u i x i, z i ) = σ 2, i = 1, 2,, n x i z i 1 3 / 31 y i = β
13 2018 7 27 1 / 31 1. 2. 2 / 31 y i = β 0 + β X x i + β Z z i + β XZ x i z i + u i, E(u i x i, z i ) = 0, E(u i u j x i, z i ) = 0 (i j), V(u i x i, z i ) = σ 2, i = 1, 2,, n x i z i 1 3 / 31 y i = β
03.Œk’ì
 HRS KG NG-HRS NG-KG AIC Fama 1965 Mandelbrot Blattberg Gonedes t t Kariya, et. al. Nagahara ARCH EngleGARCH Bollerslev EGARCH Nelson GARCH Heynen, et. al. r n r n =σ n w n logσ n =α +βlogσ n 1 + v n w
HRS KG NG-HRS NG-KG AIC Fama 1965 Mandelbrot Blattberg Gonedes t t Kariya, et. al. Nagahara ARCH EngleGARCH Bollerslev EGARCH Nelson GARCH Heynen, et. al. r n r n =σ n w n logσ n =α +βlogσ n 1 + v n w
p.1/22
 p.1/22 & & & & Excel / p.2/22 & & & & Excel / p.2/22 ( ) ( ) p.3/22 ( ) ( ) Baldi Web p.3/22 ( ) ( ) Baldi Web ( ) ( ) ( p.3/22 ) Text Mining for Clementine True Teller Text Mining Studio Text Miner Trustia
p.1/22 & & & & Excel / p.2/22 & & & & Excel / p.2/22 ( ) ( ) p.3/22 ( ) ( ) Baldi Web p.3/22 ( ) ( ) Baldi Web ( ) ( ) ( p.3/22 ) Text Mining for Clementine True Teller Text Mining Studio Text Miner Trustia
Microsoft PowerPoint - GLMMexample_ver pptx
 Linear Mixed Model ( 以下 混合モデル ) の短い解説 この解説のPDFは http://www.lowtem.hokudai.ac.jp/plantecol/akihiro/sumida-index.html の お勉強 のページにあります. ver 20121121 と との間に次のような関係が見つかったとしよう 全体的な傾向に対する回帰直線を点線で示した ところが これらのデータは実は異なる
Linear Mixed Model ( 以下 混合モデル ) の短い解説 この解説のPDFは http://www.lowtem.hokudai.ac.jp/plantecol/akihiro/sumida-index.html の お勉強 のページにあります. ver 20121121 と との間に次のような関係が見つかったとしよう 全体的な傾向に対する回帰直線を点線で示した ところが これらのデータは実は異なる
1 Tokyo Daily Rainfall (mm) Days (mm)
 ( ) r-taka@maritime.kobe-u.ac.jp 1 Tokyo Daily Rainfall (mm) 0 100 200 300 0 10000 20000 30000 40000 50000 Days (mm) 1876 1 1 2013 12 31 Tokyo, 1876 Daily Rainfall (mm) 0 50 100 150 0 100 200 300 Tokyo,
( ) r-taka@maritime.kobe-u.ac.jp 1 Tokyo Daily Rainfall (mm) 0 100 200 300 0 10000 20000 30000 40000 50000 Days (mm) 1876 1 1 2013 12 31 Tokyo, 1876 Daily Rainfall (mm) 0 50 100 150 0 100 200 300 Tokyo,
Stanによるハミルトニアンモンテカルロ法を用いたサンプリングについて
 Stan によるハミルトニアンモンテカルロ法を用いたサンプリングについて 10 月 22 日中村文士 1 目次 1.STANについて 2.RでSTANをするためのインストール 3.STANのコード記述方法 4.STANによるサンプリングの例 2 1.STAN について ハミルトニアンモンテカルロ法に基づいた事後分布からのサンプリングなどができる STAN の HP: mc-stan.org 3 由来
Stan によるハミルトニアンモンテカルロ法を用いたサンプリングについて 10 月 22 日中村文士 1 目次 1.STANについて 2.RでSTANをするためのインストール 3.STANのコード記述方法 4.STANによるサンプリングの例 2 1.STAN について ハミルトニアンモンテカルロ法に基づいた事後分布からのサンプリングなどができる STAN の HP: mc-stan.org 3 由来
tokei01.dvi
 2. :,,,. :.... Apr. - Jul., 26FY Dept. of Mechanical Engineering, Saga Univ., JAPAN 4 3. (probability),, 1. : : n, α A, A a/n. :, p, p Apr. - Jul., 26FY Dept. of Mechanical Engineering, Saga Univ., JAPAN
2. :,,,. :.... Apr. - Jul., 26FY Dept. of Mechanical Engineering, Saga Univ., JAPAN 4 3. (probability),, 1. : : n, α A, A a/n. :, p, p Apr. - Jul., 26FY Dept. of Mechanical Engineering, Saga Univ., JAPAN
R John Fox R R R Console library(rcmdr) Rcmdr R GUI Windows R R SDI *1 R Console R 1 2 Windows XP Windows * 2 R R Console R ˆ R
 R John Fox 2006 8 26 2008 8 28 1 R R R Console library(rcmdr) Rcmdr R GUI Windows R R SDI *1 R Console R 1 2 Windows XP Windows * 2 R R Console R ˆ R GUI R R R Console > ˆ 2 ˆ Fox(2005) jfox@mcmaster.ca
R John Fox 2006 8 26 2008 8 28 1 R R R Console library(rcmdr) Rcmdr R GUI Windows R R SDI *1 R Console R 1 2 Windows XP Windows * 2 R R Console R ˆ R GUI R R R Console > ˆ 2 ˆ Fox(2005) jfox@mcmaster.ca
Microsoft PowerPoint - R-stat-intro_20.ppt [互換モード]
![Microsoft PowerPoint - R-stat-intro_20.ppt [互換モード] Microsoft PowerPoint - R-stat-intro_20.ppt [互換モード]](/thumbs/95/123289731.jpg) と WinBUGS R で統計解析入門 (20) ベイズ統計 超 入門 WinBUGS と R2WinBUGS のセットアップ 1. 本資料で使用するデータを以下からダウンロードする http://www.cwk.zaq.ne.jp/fkhud708/files/r-intro/r-stat-intro_data.zip 2. WinBUGS のホームページから下記ファイルをダウンロードし WinBUGS14.exe
と WinBUGS R で統計解析入門 (20) ベイズ統計 超 入門 WinBUGS と R2WinBUGS のセットアップ 1. 本資料で使用するデータを以下からダウンロードする http://www.cwk.zaq.ne.jp/fkhud708/files/r-intro/r-stat-intro_data.zip 2. WinBUGS のホームページから下記ファイルをダウンロードし WinBUGS14.exe
分布
 (normal distribution) 30 2 Skewed graph 1 2 (variance) s 2 = 1/(n-1) (xi x) 2 x = mean, s = variance (variance) (standard deviation) SD = SQR (var) or 8 8 0.3 0.2 0.1 0.0 0 1 2 3 4 5 6 7 8 8 0 1 8 (probability
(normal distribution) 30 2 Skewed graph 1 2 (variance) s 2 = 1/(n-1) (xi x) 2 x = mean, s = variance (variance) (standard deviation) SD = SQR (var) or 8 8 0.3 0.2 0.1 0.0 0 1 2 3 4 5 6 7 8 8 0 1 8 (probability
> usdata01 と打ち込んでエンター キーを押すと V1 V2 V : : : : のように表示され 読み込まれていることがわかる ここで V1, V2, V3 は R が列のデータに自 動的につけた変数名である ( variable
 R による回帰分析 ( 最小二乗法 ) この資料では 1. データを読み込む 2. 最小二乗法によってパラメーターを推定する 3. データをプロットし 回帰直線を書き込む 4. いろいろなデータの読み込み方について簡単に説明する 1. データを読み込む 以下では read.table( ) 関数を使ってテキストファイル ( 拡張子が.txt のファイル ) のデー タの読み込み方を説明する 1.1
R による回帰分析 ( 最小二乗法 ) この資料では 1. データを読み込む 2. 最小二乗法によってパラメーターを推定する 3. データをプロットし 回帰直線を書き込む 4. いろいろなデータの読み込み方について簡単に説明する 1. データを読み込む 以下では read.table( ) 関数を使ってテキストファイル ( 拡張子が.txt のファイル ) のデー タの読み込み方を説明する 1.1
卒業論文
 Y = ax 1 b1 X 2 b2...x k bk e u InY = Ina + b 1 InX 1 + b 2 InX 2 +...+ b k InX k + u X 1 Y b = ab 1 X 1 1 b 1 X 2 2...X bk k e u = b 1 (ax b1 1 X b2 2...X bk k e u ) / X 1 = b 1 Y / X 1 X 1 X 1 q YX1
Y = ax 1 b1 X 2 b2...x k bk e u InY = Ina + b 1 InX 1 + b 2 InX 2 +...+ b k InX k + u X 1 Y b = ab 1 X 1 1 b 1 X 2 2...X bk k e u = b 1 (ax b1 1 X b2 2...X bk k e u ) / X 1 = b 1 Y / X 1 X 1 X 1 q YX1
H22 BioS (i) I treat1 II treat2 data d1; input group patno treat1 treat2; cards; ; run; I
 H BioS (i) I treat II treat data d; input group patno treat treat; cards; 8 7 4 8 8 5 5 6 ; run; I II sum data d; set d; sum treat + treat; run; sum proc gplot data d; plot sum * group ; symbol c black
H BioS (i) I treat II treat data d; input group patno treat treat; cards; 8 7 4 8 8 5 5 6 ; run; I II sum data d; set d; sum treat + treat; run; sum proc gplot data d; plot sum * group ; symbol c black
BMIdata.txt DT DT <- read.table("bmidata.txt") DT head(dt) names(dt) str(dt)
 ?read.table read.table(file, header = FALSE, sep = "", quote = "\" ", dec = ".", numerals = c("allow.loss", "warn.loss", "no.loss"), row.names, col.names, as.is =!stringsasfactors, na.strings = "NA", colclasses
?read.table read.table(file, header = FALSE, sep = "", quote = "\" ", dec = ".", numerals = c("allow.loss", "warn.loss", "no.loss"), row.names, col.names, as.is =!stringsasfactors, na.strings = "NA", colclasses
2 / 39
 W707 s-taiji@is.titech.ac.jp 1 / 39 2 / 39 1 2 3 3 / 39 q f (x; α) = α j B j (x). j=1 min α R n+2 n ( d (Y i f (X i ; α)) 2 2 ) 2 f (x; α) + λ dx 2 dx. i=1 f B j 4 / 39 : q f (x) = α j B j (x). j=1 : x
W707 s-taiji@is.titech.ac.jp 1 / 39 2 / 39 1 2 3 3 / 39 q f (x; α) = α j B j (x). j=1 min α R n+2 n ( d (Y i f (X i ; α)) 2 2 ) 2 f (x; α) + λ dx 2 dx. i=1 f B j 4 / 39 : q f (x) = α j B j (x). j=1 : x
@i_kiwamu Bayes - -
 Bayes RStan 1 2012 12 1 R @ @i_kiwamu Bayes - - Stan / RStan Bayes Stan Development Team - Andrew Gelman, Bob Carpenter, Matt Hoffman, Ben Goodrich, Michael Malecki, Daniel Lee and Chad Scherrer Open source
Bayes RStan 1 2012 12 1 R @ @i_kiwamu Bayes - - Stan / RStan Bayes Stan Development Team - Andrew Gelman, Bob Carpenter, Matt Hoffman, Ben Goodrich, Michael Malecki, Daniel Lee and Chad Scherrer Open source
NLMIXED プロシジャを用いた生存時間解析 伊藤要二アストラゼネカ株式会社臨床統計 プログラミング グループグルプ Survival analysis using PROC NLMIXED Yohji Itoh Clinical Statistics & Programming Group, A
 NLMIXED プロシジャを用いた生存時間解析 伊藤要二アストラゼネカ株式会社臨床統計 プログラミング グループグルプ Survival analysis using PROC NLMIXED Yohji Itoh Clinical Statistics & Programming Group, AstraZeneca KK 要旨 : NLMIXEDプロシジャの最尤推定の機能を用いて 指数分布 Weibull
NLMIXED プロシジャを用いた生存時間解析 伊藤要二アストラゼネカ株式会社臨床統計 プログラミング グループグルプ Survival analysis using PROC NLMIXED Yohji Itoh Clinical Statistics & Programming Group, AstraZeneca KK 要旨 : NLMIXEDプロシジャの最尤推定の機能を用いて 指数分布 Weibull
Stata 11 Stata ts (ARMA) ARCH/GARCH whitepaper mwp 3 mwp-083 arch ARCH 11 mwp-051 arch postestimation 27 mwp-056 arima ARMA 35 mwp-003 arima postestim
 TS001 Stata 11 Stata ts (ARMA) ARCH/GARCH whitepaper mwp 3 mwp-083 arch ARCH 11 mwp-051 arch postestimation 27 mwp-056 arima ARMA 35 mwp-003 arima postestimation 49 mwp-055 corrgram/ac/pac 56 mwp-009 dfgls
TS001 Stata 11 Stata ts (ARMA) ARCH/GARCH whitepaper mwp 3 mwp-083 arch ARCH 11 mwp-051 arch postestimation 27 mwp-056 arima ARMA 35 mwp-003 arima postestimation 49 mwp-055 corrgram/ac/pac 56 mwp-009 dfgls
Stata 11 Stata ROC whitepaper mwp anova/oneway 3 mwp-042 kwallis Kruskal Wallis 28 mwp-045 ranksum/median / 31 mwp-047 roctab/roccomp ROC 34 mwp-050 s
 BR003 Stata 11 Stata ROC whitepaper mwp anova/oneway 3 mwp-042 kwallis Kruskal Wallis 28 mwp-045 ranksum/median / 31 mwp-047 roctab/roccomp ROC 34 mwp-050 sampsi 47 mwp-044 sdtest 54 mwp-043 signrank/signtest
BR003 Stata 11 Stata ROC whitepaper mwp anova/oneway 3 mwp-042 kwallis Kruskal Wallis 28 mwp-045 ranksum/median / 31 mwp-047 roctab/roccomp ROC 34 mwp-050 sampsi 47 mwp-044 sdtest 54 mwp-043 signrank/signtest
151021slide.dvi
 : Mac I 1 ( 5 Windows (Mac Excel : Excel 2007 9 10 1 4 http://asakura.co.jp/ books/isbn/978-4-254-12172-8/ (1 1 9 1/29 (,,... (,,,... (,,, (3 3/29 (, (F7, Ctrl + i, (Shift +, Shift + Ctrl (, a i (, Enter,
: Mac I 1 ( 5 Windows (Mac Excel : Excel 2007 9 10 1 4 http://asakura.co.jp/ books/isbn/978-4-254-12172-8/ (1 1 9 1/29 (,,... (,,,... (,,, (3 3/29 (, (F7, Ctrl + i, (Shift +, Shift + Ctrl (, a i (, Enter,
インターネットを活用した経済分析 - フリーソフト Rを使おう
 R 1 1 1 2017 2 15 2017 2 15 1/64 2 R 3 R R RESAS 2017 2 15 2/64 2 R 3 R R RESAS 2017 2 15 3/64 2-4 ( ) ( (80%) (20%) 2017 2 15 4/64 PC LAN R 2017 2 15 5/64 R R 2017 2 15 6/64 3-4 R 15 + 2017 2 15 7/64
R 1 1 1 2017 2 15 2017 2 15 1/64 2 R 3 R R RESAS 2017 2 15 2/64 2 R 3 R R RESAS 2017 2 15 3/64 2-4 ( ) ( (80%) (20%) 2017 2 15 4/64 PC LAN R 2017 2 15 5/64 R R 2017 2 15 6/64 3-4 R 15 + 2017 2 15 7/64
Isogai, T., Building a dynamic correlation network for fat-tailed financial asset returns, Applied Network Science (7):-24, 206,
 H28. (TMU) 206 8 29 / 34 2 3 4 5 6 Isogai, T., Building a dynamic correlation network for fat-tailed financial asset returns, Applied Network Science (7):-24, 206, http://link.springer.com/article/0.007/s409-06-0008-x
H28. (TMU) 206 8 29 / 34 2 3 4 5 6 Isogai, T., Building a dynamic correlation network for fat-tailed financial asset returns, Applied Network Science (7):-24, 206, http://link.springer.com/article/0.007/s409-06-0008-x
10
 z c j = N 1 N t= j1 [ ( z t z ) ( )] z t j z q 2 1 2 r j /N j=1 1/ N J Q = N(N 2) 1 N j j=1 r j 2 2 χ J B d z t = z t d (1 B) 2 z t = (z t z t 1 ) (z t 1 z t 2 ) (1 B s )z t = z t z t s _ARIMA CONSUME
z c j = N 1 N t= j1 [ ( z t z ) ( )] z t j z q 2 1 2 r j /N j=1 1/ N J Q = N(N 2) 1 N j j=1 r j 2 2 χ J B d z t = z t d (1 B) 2 z t = (z t z t 1 ) (z t 1 z t 2 ) (1 B s )z t = z t z t s _ARIMA CONSUME
(pdf) (cdf) Matlab χ ( ) F t
 (, ) (univariate) (bivariate) (multi-variate) Matlab Octave Matlab Matlab/Octave --...............3. (pdf) (cdf)...3.4....4.5....4.6....7.7. Matlab...8.7.....9.7.. χ ( )...0.7.3.....7.4. F....7.5. t-...3.8....4.8.....4.8.....5.8.3....6.8.4....8.8.5....8.8.6....8.9....9.9.....9.9.....0.9.3....0.9.4.....9.5.....0....3
(, ) (univariate) (bivariate) (multi-variate) Matlab Octave Matlab Matlab/Octave --...............3. (pdf) (cdf)...3.4....4.5....4.6....7.7. Matlab...8.7.....9.7.. χ ( )...0.7.3.....7.4. F....7.5. t-...3.8....4.8.....4.8.....5.8.3....6.8.4....8.8.5....8.8.6....8.9....9.9.....9.9.....0.9.3....0.9.4.....9.5.....0....3
¥¤¥ó¥¿¡¼¥Í¥Ã¥È·×¬¤È¥Ç¡¼¥¿²òÀÏ Âè2²ó
 2 212 4 13 1 (4/6) : ruby 2 / 35 ( ) : gnuplot 3 / 35 ( ) 4 / 35 (summary statistics) : (mean) (median) (mode) : (range) (variance) (standard deviation) 5 / 35 (mean): x = 1 n (median): { xr+1 m, m = 2r
2 212 4 13 1 (4/6) : ruby 2 / 35 ( ) : gnuplot 3 / 35 ( ) 4 / 35 (summary statistics) : (mean) (median) (mode) : (range) (variance) (standard deviation) 5 / 35 (mean): x = 1 n (median): { xr+1 m, m = 2r
Microsoft PowerPoint - Rを利用した回帰分析.pptx
 R を利 した回帰分析 中央水産研究所 岡村 寛 水産資源学における統計解析 漁業 調査データ解析 CPUE 標準化 資源のトレンド 体 組成のモード分解 成 式などの生物パラメータの推定 資源評価モデルによる個体群評価 ほとんどがパラメータの推定問題 今日の概要 前半 ( 岡村 ) 単回帰 重回帰モデル一般化線形 ( 混合 加法 ) モデルプロダクションモデル,VPA など 最小二乗法 最尤法 ベイズ推定
R を利 した回帰分析 中央水産研究所 岡村 寛 水産資源学における統計解析 漁業 調査データ解析 CPUE 標準化 資源のトレンド 体 組成のモード分解 成 式などの生物パラメータの推定 資源評価モデルによる個体群評価 ほとんどがパラメータの推定問題 今日の概要 前半 ( 岡村 ) 単回帰 重回帰モデル一般化線形 ( 混合 加法 ) モデルプロダクションモデル,VPA など 最小二乗法 最尤法 ベイズ推定
untitled
 2 : n =1, 2,, 10000 0.5125 0.51 0.5075 0.505 0.5025 0.5 0.4975 0.495 0 2000 4000 6000 8000 10000 2 weak law of large numbers 1. X 1,X 2,,X n 2. µ = E(X i ),i=1, 2,,n 3. σi 2 = V (X i ) σ 2,i=1, 2,,n ɛ>0
2 : n =1, 2,, 10000 0.5125 0.51 0.5075 0.505 0.5025 0.5 0.4975 0.495 0 2000 4000 6000 8000 10000 2 weak law of large numbers 1. X 1,X 2,,X n 2. µ = E(X i ),i=1, 2,,n 3. σi 2 = V (X i ) σ 2,i=1, 2,,n ɛ>0
.3 ˆβ1 = S, S ˆβ0 = ȳ ˆβ1 S = (β0 + β1i i) β0 β1 S = (i β0 β1i) = 0 β0 S = (i β0 β1i)i = 0 β1 β0, β1 ȳ β0 β1 = 0, (i ȳ β1(i ))i = 0 {(i ȳ)(i ) β1(i ))
 Copright (c) 004,005 Hidetoshi Shimodaira 1.. 3. 4. 004-10-01 16:15:07 shimo cat(" 1: "); c(mea(), mea()) cat(" : "); mmea
Copright (c) 004,005 Hidetoshi Shimodaira 1.. 3. 4. 004-10-01 16:15:07 shimo cat(" 1: "); c(mea(), mea()) cat(" : "); mmea
JMP V4 による生存時間分析
 V4 1 SAS 2000.11.18 4 ( ) (Survival Time) 1 (Event) Start of Study Start of Observation Died Died Died Lost End Time Censor Died Died Censor Died Time Start of Study End Start of Observation Censor
V4 1 SAS 2000.11.18 4 ( ) (Survival Time) 1 (Event) Start of Study Start of Observation Died Died Died Lost End Time Censor Died Died Censor Died Time Start of Study End Start of Observation Censor

