PowerPoint プレゼンテーション
|
|
|
- あゆみ むらかわ
- 9 years ago
- Views:
Transcription
1 平成 28 年度 NGS ハンズオン講習会 ChIP-seq 2016 年 7 月 28 日
2 本講義にあたって 代表的な解析の流れを紹介します 論文でよく使用されているツールを使用します コマンドを沢山実行します タイプミスが心配な方は コマンド例がありますのでコピーして実行してください 実行が遅れてもあせらずに 課題や休憩の間に追い付いてください Amelieff Corporation All Rights Reserved 2
3 本講義の内容 ChIP-seqとは ChIP-seq 解析の流れ 公開データの取得 クオリティコントロール マッピング ピーク検出 ピークアノテーション モチーフ探索 まとめ 最後に Amelieff Corporation All Rights Reserved 3
4 ChIP-seq とは ChIP(Chromatin Immuno Precipitation) + NGS sequencing クロマチン免疫沈降により濃縮したゲノム領域をシーケンスする手法 主な解析対象 タンパクとDNAの相互作用 ヒストン修飾 Licensed under CC-BY 4.0 Togo picture gallery by DBCLS Amelieff Corporation All Rights Reserved 4
5 ChIP-seq とは ChIP-seq で主に解析されるのは転写調節領域 A User s Guide to the Encyclopedia of DNA Elements (ENCODE), 2011 より Amelieff Corporation All Rights Reserved 5
6 ChIP-seq とは input と IP ChIP-seq では 免疫沈降のバックグラウンドノイズを削減するため コントロールを使用することが多い 免疫沈降 (IP) を行っていないサンプルをコントロールとして使用し 検出したピークを抗体に非特異的なものとして取り除くために用いる 一般にこのコントロールを input と呼ぶ IP input Licensed under CC-BY 4.0 Togo picture gallery by DBCLS Amelieff Corporation All Rights Reserved 6
7 ChIP-seq とは ChIP-seq では 抗体が特異的に結合した領域をピークとして得る : シーケンスリード A User s Guide to the Encyclopedia of DNA Elements (ENCODE), 2011 より Amelieff Corporation All Rights Reserved 7
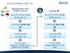
8 ChIP-seq 解析の流れ 代表的なソフト クオリティコントロール Trimmomatic, fastqc, FASTX_Toolkit... マッピング Bowtie, Bowtie2, bwa... ピーク検出 MACS, MACS2, SICER... ピークアノテーション SnpEff, ChIPpeakAnno... モチーフ探索 rgadem... ChIP-seq 解析の一般的な流れであり 全ての ChIP-seq で同一の解析を行うわけではない 研究の目的やデータに合わせて 最適な解析を設計 Amelieff Corporation All Rights Reserved 8
9 クオリティコントロール 他の NGS データ解析と同様に 解析前のクオリティコントロールを実施 本日使用するソフト トリミング 低クオリティリードの除去 Trimmomatic クオリティチェック Fastqc ChIP-seqにおけるポイント リード長に注意する (75 bp 以下など短い場合が多い ) Amelieff Corporation All Rights Reserved 9
10 マッピング Reseqでも使用されるマッピングソフトがChIP-seqでよく使用される 本日使用するソフト bowtie2 ギャップアラインメントに対応 マッピング精度が高い この他に使用されるソフト bowtie ギャップアラインメントに非対応 bwa Amelieff Corporation All Rights Reserved 10
11 ピーク検出 ピーク検出ソフトは IP で濃縮した領域のリードの頂点を検出する 本日使用するソフト MACS2( デファクトスタンダード ) 被引用数 2750 件 (2016 年 7 月 20 日時点 ) この他に使用されるソフト SICER 被引用数 425 件 (2016 年 7 月 20 日時点 ) MACS Amelieff Corporation All Rights Reserved 11
12 ピークアノテーション ピーク検出後に ピークがゲノム上のどのような位置に存在するのか アノテーションする 本日使用するソフト SnpEff 遺伝子名を付与 遺伝子上のドメイン ( エキソン 上流など ) を付与 様々な生物種に対応 この他に使用されるソフト ChIPpeakAnno Rパッケージ Amelieff Corporation All Rights Reserved 12
13 モチーフ探索 検出されたピークに共通のモチーフを探索する モチーフは 抗体と結合する短い配列で ピーク配列に共通して見られる 本日使用するソフト rgadem Artistic License 2.0( 改変 再配布 商用可 ) なので利用しやすい この他に使用されるソフト MEME 商用利用不可 Amelieff Corporation All Rights Reserved 13
14 公開データの取得 今回の解析に必要なデータ リファレンスゲノム ( 実行済み ) 解析対象のシーケンスデータ ( 実行済み ) Amelieff Corporation All Rights Reserved 14
15 公開データの取得 酵母のリファレンスゲノムデータの取得方法 $ wget ftp://igenome:[email protected]/saccharomyces_cerevisiae/ncbi/build3.1/saccha romyces_cerevisiae_ncbi_build3.1.tar.gz $ tar zxvf Saccharomyces_cerevisiae_NCBI_build3.1.tar.gz Saccharomyces cerevisiae のリファレンスゲノムをイルミナの Web ページからダウンロードし解凍 ( 実行済み ) $ ls -l /home/ ユーザ名 /Desktop/amelieff/sacCer3/ : -rwxr-xr-x. 1 root root 月 23 11: genome.fa -rwxr-xr-x. 1 root root 月 23 11: genome.fa.fai -rwxr--r--. 1 root root 月 23 11: mask.gtf -rwxr-xr-x. 1 root root 月 23 11: refgene.txt /home/ ユーザ名 /Desktop/amelieff/Scerevisiae/ の 解凍したファイル ( 今回使用するもののみ ) を確認 Amelieff Corporation All Rights Reserved 15
16 公開データの取得 fasta ファイルの中身の確認 $ less /home/ ユーザ名 /Desktop/amelieff/Scerevisiae/genome.fa >chri CCACACCACACCCACACACCCACACACCACACCACACACCACACCACACC CACACACACACATCCTAACACTACCCTAACACAGCCCTAATCTAACCCTG GCCAACCTGTCTCTCAACTTACCCTCCATTACCCTGCCTCCACTCGTTAC CCTGTCCCATTCAACCATACCACTCCGAACCACCATCCATCCCTCTACTT ACTACCACTCACCCACCGTTACCCTCCAATTACCCATATCCAACCCACTG : 1 行目 : コンティグ名 2 行目以降 : 実際の配列情報 q で閲覧を終了する Amelieff Corporation All Rights Reserved 16
17 公開データの取得 データの探し方 解析対象のシーケンスデータの取得方法 NCBI SRA( へアクセスする Amelieff Corporation All Rights Reserved 17
18 公開データの取得 データの探し方 論文中などから得られたアクセッション番号の ERR を検索する Amelieff Corporation All Rights Reserved 18
19 公開データの取得 データの探し方 研究情報 サンプル情報 ライブラリ情報 Amelieff Corporation All Rights Reserved 19
20 公開データの取得 データの探し方 All runs を選択同じ Study でシーケンスした全てのデータを確認 Amelieff Corporation All Rights Reserved 20
21 公開データの取得 データの探し方 SRA Run Selector でデータを確認する 47Runs 合計 4.42GB のデータ量 Amelieff Corporation All Rights Reserved 21
22 公開データの取得 データの探し方 ERR (input) と ERR (sample) を選択 コントロール (input) サンプル (H3K56ac) ダウンロードするデータにチェック Amelieff Corporation All Rights Reserved 22
23 公開データの取得 データの探し方 Accession List をダウンロード アクセッション番号のリスト ( テキストファイル ) がダウンロードされる Amelieff Corporation All Rights Reserved 23
24 公開データの取得 ダウンロード方法 SRA のダウンロードには SRA-Tools を使用する SRA-Tools( 主な用途 実行コマンド NCBI SRA からのデータダウンロード prefetch SRA FASTQ のフォーマット変換 fastq-dump Amelieff Corporation All Rights Reserved 24
25 公開データの取得 ダウンロード方法 SRA-Tools のインストール 本日はデータを用意済みのため実施しません $ wget $ tar xf sratoolkit centos_linux64.tar.gz $ ln -s sratoolkit centos_linux64/bin/prefetch /usr/local/bin/ $ ln -s sratoolkit centos_linux64/bin/fastq-dump /usr/local/bin/ 参考 : Amelieff Corporation All Rights Reserved 25
26 公開データの取得 ダウンロード方法 SRA-Tools の prefetch コマンドでまとめて SRA をダウンロード ダウンロードした Accession List(SRR_Acc_List.txt) を --option-file で指定 $ prefetch --option-file SRR_Acc_List.txt デフォルトで SRA は ~/ncbi/public/sra/ に保存される $ ls ~/ncbi/public/sra/ ERR sra ERR sra Amelieff Corporation All Rights Reserved 26
27 公開データの取得 SRA の変換方法 SRA-Tools の fastq-dump を使用して SRA から FASTQ へ変換する 変換データを保存するディレクトリ (data) を作成する ( 実行済み ) $ mkdir data $ cd data --split-files を付けてペアエンドのファイルを分割しながら FASTQ に変換する ( 実行済み ) どこでペアエンドかシングルエンドかを確認するのか ( 次のスライドで解説 ) $ fastq-dump ~/ncbi/public/sra/err sra --split-files $ fastq-dump ~/ncbi/public/sra/err sra --split-files Amelieff Corporation All Rights Reserved 27
28 公開データの取得 SRA の変換方法 Run selector の LibraryLayout でペアエンドであることを確認できる 本日使用するデータは全てペアエンド (PAIRED) Amelieff Corporation All Rights Reserved 28
29 公開データの取得 SRA の変換方法 SRA-Tools の fastq-dump を使用して SRA から FASTQ へ変換する 変換した FASTQ を確認する $ ls ERR _1.fastq ERR _2.fastq ERR _1.fastq ERR _2.fastq Amelieff Corporation All Rights Reserved 29
30 公開データの取得 実習用データの作成 seqtk( を使用し 実習用に FASTQ からデータの一部を抜粋する seqtk のインストール ( 今回は実施しません ) $ wget $ tar xf v1.2.tar.gz $ cd seqtk-1.2 $ ln -s ~/src/seqtk-1.2/seqtk /usr/local/bin/ Amelieff Corporation All Rights Reserved 30
31 公開データの取得 実習用データの作成 seqtk を使用し 実習用に FASTQ からデータの一部を抜粋する seqtk の実行 $ seqtk sample -s 100 ERR _1.fastq > input_1.fastq $ seqtk sample -s 100 ERR _2.fastq > input_2.fastq $ seqtk sample -s 100 ERR _1.fastq > sample_1.fastq $ seqtk sample -s 100 ERR _2.fastq > sample_2.fastq -s 100: シード値を100に指定ペアで同じシード値を使うことで ランダムに抽出するリードのペアを保つ事ができる :50 万リード抽出 Amelieff Corporation All Rights Reserved 31
32 実習パート Amelieff Corporation All Rights Reserved 32
33 公開データの取得 解析対象のシーケンスデータの取得方法 ( 実行済み ) ダウンロード SRA FASTQ 変換 $ prefetch --option-file SRR_Acc_List.txt $ fastq-dump ~/ncbi/public/sra/err12315*.sra --split-files 実習用の軽量なデータを作成 ( 実行済み ) $ seqtk sample -s 100 ERR _1.fastq > input_1.fastq $ seqtk sample -s 100 ERR _2.fastq > input_2.fastq $ seqtk sample -s 100 ERR _1.fastq > sample_1.fastq $ seqtk sample -s 100 ERR _2.fastq > sample_2.fastq Amelieff Corporation All Rights Reserved 33
34 公開データの取得 解析対象のシーケンスデータの確認 $ cd /home/iu/chipseq $ ls data input_1.fastq.gz sample_1.fastq.gz input_2.fastq.gz sample_2.fastq.gz アクセッション番号との対応 input_1 ERR _1.fastq.gz input_2 ERR _2.fastq.gz sample_1 ERR _1.fastq.gz sample_2 ERR _2.fastq.gz それぞれ 500,000 リードのデータ Amelieff Corporation All Rights Reserved 34
35 クオリティコントロール QC 前の品質確認 シーケンスクオリティチェックソフトウェア FastQC の実行 $ mkdir fastqc_before $ fastqc --nogroup -t 2 -o./fastqc_before data/input_1.fastq.gz data/input_2.fastq.gz data/sample_1.fastq.gz data/sample_2.fastq.gz $ ls fastqc_before input_1_fastqc input_2_fastqc.zip sample_2_fastqc input_1_fastqc.zip sample_1_fastqc sample_2_fastqc.zip input_2_fastqc sample_1_fastqc.zip Amelieff Corporation All Rights Reserved 35
36 クオリティコントロール QC 前の品質確認 FastQC の結果確認 (QC 前 ) 解析結果の html ファイルをブラウザ (firefox) で確認 $ firefox fastqc_before/input_1_fastqc/fastqc_report.html fastqc_before/input_2_fastqc/fastqc_report.html fastqc_before/sample_1_fastqc/fastqc_report.html fastqc_before/sample_2_fastqc/fastqc_report.html ブラウザでタブが 4 つ開かれ クオリティチェックの解析結果が確認できる Amelieff Corporation All Rights Reserved 36
37 クオリティコントロール QC 前の品質確認 fastqc summary (QC 前 ) input1 input2 sample1 sample2 Amelieff Corporation All Rights Reserved 37
38 クオリティコントロール QC 処理 今回のデータに対する処理 (Trimmomatic を用いた一括処理 1) $ mkdir trimmed_data $ java -jar /usr/local/bin/trimmomatic-0.36.jar PE -threads 2 -phred33 data/input_1.fastq.gz data/input_2.fastq.gz trimmed_data/input_1_paired.fastq trimmed_data/input_1_unpaired.fastq trimmed_data/input_2_paired.fastq trimmed_data/input_2_unpaired.fastq LEADING:20 TRAILING:20 SLIDINGWINDOW:4:15 MINLEN:36 sample~ でも同様の処理を実行 Amelieff Corporation All Rights Reserved 38
39 クオリティコントロール QC 処理 今回のデータに対する処理 (Trimmomatic を用いた一括処理 2) $ java -jar /usr/local/bin/trimmomatic-0.36.jar PE -threads 2 -phred33 data/sample_1.fastq.gz data/sample_2.fastq.gz trimmed_data/sample_1_paired.fastq trimmed_data/sample_1_unpaired.fastq trimmed_data/sample_2_paired.fastq trimmed_data/sample_2_unpaired.fastq LEADING:20 TRAILING:20 SLIDINGWINDOW:4:15 MINLEN:36 CPU のコア数に余裕があれば threads の数値を大きくすることで より高速に処理することが可能 Amelieff Corporation All Rights Reserved 39
40 TIPS CPU コア数を確認してみる $ cat /proc/cpuinfo cpu cores を確認 ここでは 2 となっている 全てのコアを使用して計算しようとすると かえって遅くなる時もあるので コマンド実行時は様子を見ながら増やす Amelieff Corporation All Rights Reserved 40
41 クオリティコントロール QC 後の品質確認 FastQC の結果確認 (QC 後 ) $ mkdir fastqc_after $ fastqc --nogroup -t 2 -o fastqc_after trimmed_data/input_1_paired.fastq trimmed_data/input_2_paired.fastq trimmed_data/sample_1_paired.fastq trimmed_data/sample_2_paired.fastq $ firefox fastqc_after/input_1_paired_fastqc/fastqc_report.html fastqc_after/input_2_paired_fastqc/fastqc_report.html fastqc_after/sample_1_paired_fastqc/fastqc_report.html fastqc_after/sample_2_paired_fastqc/fastqc_report.html Amelieff Corporation All Rights Reserved 41
42 クオリティコントロール QC 前の品質確認 FastQC summary (QC 後 ) input1 input2 sample1 sample2 Amelieff Corporation All Rights Reserved 42
43 クオリティコントロール QC 前の品質確認 input_1 input2 sample1 sample2 QC 前 QC 後 Amelieff Corporation All Rights Reserved 43
44 マッピング Bowtie2 によるマッピング (input ファイル ) $ mkdir mapping $ bowtie2 -p 2 -x /home/iu/genome/saccer3/bowtie2index/genome -1 trimmed_data/input_1_paired.fastq -2 trimmed_data/input_2_paired.fastq samtools view -Sb - > mapping/input.bam $ samtools sort mapping/input.bam -o mapping/input.sorted.bam bowtie2 のオプション -p : 使用するスレッド数 -x : bowtie2 で作成したゲノムファイルインデックス -1,-2: 入力 fastq ファイル Samtools のオプション view: SAM もしくは BAM の中身を表示 -Sb: SAM から BAM へ変換 Amelieff Corporation All Rights Reserved 44
45 マッピング Bowtie2 によるマッピング (sample ファイル ) $ bowtie2 -p 2 -x /home/iu/genome/saccer3/bowtie2index/genome -1 trimmed_data/sample_1_paired.fastq -2 trimmed_data/sample_2_paired.fastq samtools view -Sb - > mapping/sample.bam $ samtools sort mapping/sample.bam -o mapping/sample.sorted.bam Amelieff Corporation All Rights Reserved 45
46 ピーク検出 MACS2 によるピーク検出 $ macs2 callpeak -t mapping/sample.sorted.bam -c mapping/input.sorted.bam --outdir macs2_res -f BAMPE -n handson2016 -B -q g 1.2e+7 -t ターゲットサンプル (IP) のファイル -c -t に対するコントロール (input) サンプルのファイル --outdir 結果を出力するディレクトリ -f -t で指定したファイルのファイル形式 BAM SAM BED 他様々なフォーマットが指定可能 BAMPE は paired-end read をマッピングした bam ファイル ( コマンドの説明は次スライドに続きます ) Amelieff Corporation All Rights Reserved 46
47 ピーク検出 MACS2 によるピーク検出 $ macs2 callpeak -t mapping/sample.sorted.bam -c mapping/input.sorted.bam --outdir macs2_res -f BAMPE -n handson2016 -B -q g 1.2e+7 -n 出力ファイルの接頭文字 -B フラグメントの pileup control lambda 値などを BedGraph 形式で保存 -q -g peakcall するピークの閾値 (Benjamini-Hochberg による FDR の q 値 ) デフォルト 0.01 反復領域を除いたゲノムサイズ一部のモデル生物では数字ではなく ヒト :hs マウス :mm などの省略が可能 Amelieff Corporation All Rights Reserved 47
48 ピーク検出 MACS2によるピーク検出 $ ls macs2_res handson2016_control_lambda.bdg handson2016_summits.bed handson2016_peaks.narrowpeak handson2016_treat_pileup.bdg handson2016_peaks.xls 各出力ファイルの解説は NGS Surfer s Wikiが参考になる この後のモチーフ探索には ピークの領域情報が記載された handson2016_peaks.narrowpeak を用いる Amelieff Corporation All Rights Reserved 48
49 ピーク検出 先頭の 5 行を確認 $ head -5 handson2016_peaks.narrowpeak chri handson2016_peak_ chrii handson2016_peak_ chriv handson2016_peak_ chriv handson2016_peak_ chriv handson2016_peak_ Amelieff Corporation All Rights Reserved 49
50 ピーク検出 handson2016_peaks.narrowpeak の項目解説 列 1: 染色体番号 chri 2: ピーク開始位置 : ピーク終了位置 : ピークの名前 handson2016_peak_1 5: ピークのスコア 46 6: ストランド. 7:fold-change :-log10pvalue :-log10qvalue : ピーク開始位置から頂点までの距離 252 例 Amelieff Corporation All Rights Reserved 50
51 可視化 IGV でピークを確認する 検出したピークを IGV で可視化する BAMファイルのインデックスを作成 $ cd mapping $ samtools index input.sorted.bam $ samtools index sample.sorted.bam IGV で下記のファイルを表示 1. handson2016_peaks.narrowpeak 2. input.sorted.bam 3. sample.sorted.bam Amelieff Corporation All Rights Reserved 51
52 可視化 IGV でピークを確認する Gene まずは Track を見やすいように並び替える handson2016_peaks.narrowpeak input.sorted.bam sample.sorted.bam Amelieff Corporation All Rights Reserved 52
53 可視化 IGV でピークを確認する スコアの高いピークを確認 $ cd macs2_res $ cat handson2016_summits.bed sort -k 5n もっとも高いスコアを示したピーク名を IGV の検索窓に入れ検索 Amelieff Corporation All Rights Reserved 53
54 可視化 IGV でピークを確認する 検索したピークの範囲が示されている handson2016_peaks.narrowpeak Amelieff Corporation All Rights Reserved 54
55 可視化 IGV でピークを確認する ピークの頂点はこの中央 (summit) handson2016_peaks.narrowpeak Amelieff Corporation All Rights Reserved 55
56 可視化 IGV でピークを確認する それぞれのトラックの名前を右クリックし Squished を選択 Amelieff Corporation All Rights Reserved 56
57 可視化 IGV でピークを確認する 頂点の位置が空白になっている場合は ペアのリードで挟まれている領域と示唆される Amelieff Corporation All Rights Reserved 57
58 可視化 IGV でピークを確認する 今度は拡大してペアのリードを確認 Expanded を選択 Amelieff Corporation All Rights Reserved 58
59 可視化 IGV でピークを確認する 矢印が向かいあっているものはペア リード 1 リード 2 Amelieff Corporation All Rights Reserved 59
60 アノテーション handson2016_summits.bed に対して snpeff によるアノテーションを実施 アノテーション作業用のディレクトリを作成し アノテーション前のファイルを確認 $ mkdir annotation $ cd annotation $ cat../macs2_res/handson2016_summits.bed Amelieff Corporation All Rights Reserved 60
61 アノテーション handson2016_summits.bed に対して snpeff によるアノテーションを実施する $ java -jar /usr/local/bin/snpeff.jar eff -csvstats stats.txt -c /usr/local/bin/snpeff.config -i bed -o bedann R /macs2_res/handson2016_summits.bed > handson2016_summits.annotated.bed eff -csvstats 入力ファイルにアノテーションを行う csv 形式のサマリーファイルを作成する -c snpeff の設定ファイルを指定 -i 入力ファイルのフォーマット -o 出力ファイルのフォーマット ( コマンドの説明は次スライドに続きます ) Amelieff Corporation All Rights Reserved 61
62 アノテーション handson2016_summits.bed に対して snpeff によるアノテーションを実施する $ mkdir annotation $ cd annotation $ java -jar /usr/local/bin/snpeff.jar eff -csvstats stats.txt -c /usr/local/bin/snpeff.config -i bed -o bedann R /macs2_res/handson2016_summits.bed > handson2016_summits.annotated.bed R /macs2_res/handson2016_ summits.bed アノテーションに使用するゲノムバージョン 入力ファイル Amelieff Corporation All Rights Reserved 62
63 アノテーション snpeff を用いたアノテーション方法 $ less handson2016_summits.annotated.bed : # Chromo Start End Variant;Annotation Score I I:114304;EXON:ATS1 I I:114304;GENE:YAL019W-A I I:114304;UPSTREAM:FUN30 I I:114304;DOWNSTREAM:LDS1 : 検出されたピークの summit について 遺伝子名とその遺伝子に対してエクソ ン 上流 下流などの情報が付与される Amelieff Corporation All Rights Reserved 63
64 モチーフ検索 R Bioconductor package rgadem を用いた de novo モチーフ検索 1 $ mkdir../motif $ cd../motif $ R R version ( ) -- "Full of Ingredients" Copyright (C) 2015 The R Foundation for Statistical Computing Platform: x86_64-pc-linux-gnu (64-bit) R is free software and comes with ABSOLUTELY NO WARRANTY. You are welcome to redistribute it under certain conditions. Type 'license()' or 'licence()' for distribution details. : Amelieff Corporation All Rights Reserved 64
65 モチーフ検索 R Bioconductor package rgadem を用いた de novo モチーフ検索 2 > library(rgadem) > library("bsgenome.scerevisiae.ucsc.saccer3") > BED <- read.table("../macs2_res/handson2016_peaks.narrowpeak", header=false, sep=" t") > BED <- data.frame(chr=as.factor(bed[,1]), start=as.numeric(bed[,2]), end=as.numeric(bed[,3])) MACS2 から出力された BED ファイルを データフレームとして読み込む 再び handson2016_peaks.narrowpeak を使用 Amelieff Corporation All Rights Reserved 65
66 モチーフ検索 R Bioconductor package rgadem を用いた de novo モチーフ検索 3 > rgbed <- IRanges(start = BED[, 2], end = BED[, 3]) > Sequences <- RangedData(rgBED, space = BED[, 1]) > gadem <- GADEM(Sequences, verbose = 1, genome = Scerevisiae) > pdf("motif.pdf") > plot(gadem) > dev.off() > q() ピーク領域に頻出するモチーフを取得し PDF にプロット Amelieff Corporation All Rights Reserved 66
67 モチーフ検索 出力したモチーフを確認 $ evince motif.pdf この後さらに MotIVなどを使用し 検出したDNAモチーフが既知のモチーフに似ているかどうか調べることも可能 MotIV: Amelieff Corporation All Rights Reserved 67
68 まとめ ChIP-seq 解析の流れ クオリティコントロール Trimmomatic, fastqc マッピング Bowtie2 ピーク検出 MACS2 ピークアノテーション SnpEff モチーフ探索 rgadem ChIP-seq 解析の一般的な流れであり 全ての ChIP-seq で同一の解析を行うわけではない 研究の目的やデータに合わせて 最適な解析を設計 Amelieff Corporation All Rights Reserved 68
PowerPoint プレゼンテーション
 平成 28 年度 NGS ハンズオン講習会 Reseq 解析 2016 年 7 月 26 日 本講義にあたって 代表的な解析の流れを紹介します 論文でよく使用されているツールを使用します コマンドを沢山実行します スペルミスが心配な方は コマンド例がありますのでコピーして実行してください 実行が遅れてもあせらずに 課題や休憩の間に追い付いてください Amelieff Corporation All
平成 28 年度 NGS ハンズオン講習会 Reseq 解析 2016 年 7 月 26 日 本講義にあたって 代表的な解析の流れを紹介します 論文でよく使用されているツールを使用します コマンドを沢山実行します スペルミスが心配な方は コマンド例がありますのでコピーして実行してください 実行が遅れてもあせらずに 課題や休憩の間に追い付いてください Amelieff Corporation All
PowerPoint プレゼンテーション
 V1 次世代シークエンサ実習 II 本講義にあたって 代表的な解析の流れを紹介します 論文でよく使用されているツールを使用します コマンドを沢山実行します スペルミスが心配な方は コマンド例がありますのでコピーして実行してください /home/admin1409/amelieff/ngs/reseq_command.txt マークのコマンドは実行してください 実行が遅れてもあせらずに 応用や課題の間に追い付いてください
V1 次世代シークエンサ実習 II 本講義にあたって 代表的な解析の流れを紹介します 論文でよく使用されているツールを使用します コマンドを沢山実行します スペルミスが心配な方は コマンド例がありますのでコピーして実行してください /home/admin1409/amelieff/ngs/reseq_command.txt マークのコマンドは実行してください 実行が遅れてもあせらずに 応用や課題の間に追い付いてください
PowerPoint プレゼンテーション
 V1 次世代シークエンサ実習 II 本講義の内容 Reseq 解析 RNA-seq 解析 公開データ取得 クオリティコントロール マッピング 変異検出 公開データ取得 クオリティコントロール マッピング 発現定量 FPKM を算出します 2 R N A - s e q とは メッセンジャー RNA(mRNA) をキャプチャして次世代シーケンサーでシーケンシングする手法 リファレンスがある生物種の場合
V1 次世代シークエンサ実習 II 本講義の内容 Reseq 解析 RNA-seq 解析 公開データ取得 クオリティコントロール マッピング 変異検出 公開データ取得 クオリティコントロール マッピング 発現定量 FPKM を算出します 2 R N A - s e q とは メッセンジャー RNA(mRNA) をキャプチャして次世代シーケンサーでシーケンシングする手法 リファレンスがある生物種の場合
2016_RNAseq解析_修正版
 平成 28 年度 NGS ハンズオン講習会 RNA-seq 解析 2016 年 7 27 本講義にあたって n 代表的な解析の流れを紹介します 論 でよく使 されているツールを使 します n コマンドを沢 実 します タイプミスが 配な は コマンド例がありますのでコピーして実 してください 実 が遅れてもあせらずに 課題や休憩の間に追い付いてください Amelieff Corporation All
平成 28 年度 NGS ハンズオン講習会 RNA-seq 解析 2016 年 7 27 本講義にあたって n 代表的な解析の流れを紹介します 論 でよく使 されているツールを使 します n コマンドを沢 実 します タイプミスが 配な は コマンド例がありますのでコピーして実 してください 実 が遅れてもあせらずに 課題や休憩の間に追い付いてください Amelieff Corporation All
PowerPoint プレゼンテーション
 バイオインフォマティクス 講習会 V 事前準備 が完了されている方は コンテナの起動 ファイルのコピー (Windows) まで 進めておいてください メニュー 1. 環境構築の確認 2. 基本的なLinuxコマンド 3. ツールのインストール 4. NGSデータの基礎知識と前処理 5. トランスクリプトのアッセンブル 6. RNA-seqのリファレンスcDNAマッピングとFPKM 算出 7. RNA-seqのリファレンスゲノムマッピングとFPKM
バイオインフォマティクス 講習会 V 事前準備 が完了されている方は コンテナの起動 ファイルのコピー (Windows) まで 進めておいてください メニュー 1. 環境構築の確認 2. 基本的なLinuxコマンド 3. ツールのインストール 4. NGSデータの基礎知識と前処理 5. トランスクリプトのアッセンブル 6. RNA-seqのリファレンスcDNAマッピングとFPKM 算出 7. RNA-seqのリファレンスゲノムマッピングとFPKM
NGSデータ解析入門Webセミナー
 NGS データ解析入門 Web セミナー : RNA-Seq 解析編 1 RNA-Seq データ解析の手順 遺伝子発現量測定 シークエンス マッピング サンプル間比較 機能解析など 2 CLC Genomics Workbench 使用ツール シークエンスデータ メタデータのインポート NGS data import Import Metadata クオリティチェック Create Sequencing
NGS データ解析入門 Web セミナー : RNA-Seq 解析編 1 RNA-Seq データ解析の手順 遺伝子発現量測定 シークエンス マッピング サンプル間比較 機能解析など 2 CLC Genomics Workbench 使用ツール シークエンスデータ メタデータのインポート NGS data import Import Metadata クオリティチェック Create Sequencing
GWB
 NGS データ解析入門 Web セミナー : De Novo シークエンス解析編 1 NGS 新規ゲノム配列解析の手順 シークエンス 遺伝子領域の検出 アセンブル データベース検索 2 解析ワークフローと使用ソフトウェア シークエンスデータのインポート クオリティチェック 前処理 コンティグ配列の作成 CLC Genomics Workbench 遺伝子領域の検出 Blast2GO PRO データベース検索
NGS データ解析入門 Web セミナー : De Novo シークエンス解析編 1 NGS 新規ゲノム配列解析の手順 シークエンス 遺伝子領域の検出 アセンブル データベース検索 2 解析ワークフローと使用ソフトウェア シークエンスデータのインポート クオリティチェック 前処理 コンティグ配列の作成 CLC Genomics Workbench 遺伝子領域の検出 Blast2GO PRO データベース検索
シーケンサー利用技術講習会 第10回 サンプルQC、RNAseqライブラリー作製/データ解析実習講習会
 シーケンサー利用技術講習会 第 10 回サンプル QC RNAseq ライブ ラリー作製 / データ解析実習講習会 理化学研究所ライフサイエンス技術基盤研究センターゲノムネットワーク解析支援施設田上道平 次世代シーケンサー Sequencer File Format Output(Max) Read length Illumina Hiseq2500 Fastq 600 Gb 100 bp Life
シーケンサー利用技術講習会 第 10 回サンプル QC RNAseq ライブ ラリー作製 / データ解析実習講習会 理化学研究所ライフサイエンス技術基盤研究センターゲノムネットワーク解析支援施設田上道平 次世代シーケンサー Sequencer File Format Output(Max) Read length Illumina Hiseq2500 Fastq 600 Gb 100 bp Life
講義内容 ファイル形式 データの可視化 データのクオリティチェック マッピング アセンブル 資料の見方 $ pwd 実際に入力するコマンドを黄色い四角の中に示します 2
 N G S 解析基礎 講義内容 ファイル形式 データの可視化 データのクオリティチェック マッピング アセンブル 資料の見方 $ pwd 実際に入力するコマンドを黄色い四角の中に示します 2 ファイル形式 NGS 解析でよく使われるファイル形式 ファイル形式 fastq bam/sam vcf bed fasta サンプルデータの場所 /home/ ユーザ名 /Desktop/amelieff/1K_ERR038793_1.fastq
N G S 解析基礎 講義内容 ファイル形式 データの可視化 データのクオリティチェック マッピング アセンブル 資料の見方 $ pwd 実際に入力するコマンドを黄色い四角の中に示します 2 ファイル形式 NGS 解析でよく使われるファイル形式 ファイル形式 fastq bam/sam vcf bed fasta サンプルデータの場所 /home/ ユーザ名 /Desktop/amelieff/1K_ERR038793_1.fastq
リード・ゲノム・アノテーションインポート
 リード ゲノム アノテーションインポート 1 Location と Folder ロケーション フォルダ Genomics Workbenchではデータを以下のような階層構造で保存可能です フォルダの一番上位の階層を Location と呼び その下の階層を Folder と呼びます データの保存場所はロケーション毎に設定可能です たとえばあるデータは C ドライブに保存し あるデータは D ドライブに保存するといった事が可能です
リード ゲノム アノテーションインポート 1 Location と Folder ロケーション フォルダ Genomics Workbenchではデータを以下のような階層構造で保存可能です フォルダの一番上位の階層を Location と呼び その下の階層を Folder と呼びます データの保存場所はロケーション毎に設定可能です たとえばあるデータは C ドライブに保存し あるデータは D ドライブに保存するといった事が可能です
解 析 の 実 際 2 (Bismark) 1. Filtering poor quality reads, and reads with adapter sequences (TrimmomaWc) アダプターのトリミング コマンド 例 java - jar /root/bin/trimmomaw
 解 析 の 実 際 (Bismark) インストールするソフトウェア(インストール 上 の 注 意 ) Bismark (v0.12.5) インストールはダウンロードして 解 凍 するだけです BowWe2 (v2.2.3) インストールはダウンロードして 解 凍 するだけです SAMTools (v0.1.9) Makefile の curses を ncursesに 書 き 換 えてmakeします
解 析 の 実 際 (Bismark) インストールするソフトウェア(インストール 上 の 注 意 ) Bismark (v0.12.5) インストールはダウンロードして 解 凍 するだけです BowWe2 (v2.2.3) インストールはダウンロードして 解 凍 するだけです SAMTools (v0.1.9) Makefile の curses を ncursesに 書 き 換 えてmakeします
Maser RNA-seq Genome Resequencing De novo Genome Sequencing Metagenome ChIP-seq CAGE BS-seq
 NGS Maser 2013/10/17 Maser RNA-seq Genome Resequencing De novo Genome Sequencing Metagenome ChIP-seq CAGE BS-seq Maser RNA-seq Genome Resequencing De novo Genome Sequencing Metagenome ChIP-seq CAGE BS-seq
NGS Maser 2013/10/17 Maser RNA-seq Genome Resequencing De novo Genome Sequencing Metagenome ChIP-seq CAGE BS-seq Maser RNA-seq Genome Resequencing De novo Genome Sequencing Metagenome ChIP-seq CAGE BS-seq
GWB
 NGS データ解析入門 Web セミナー : 変異解析編 1 NGS 変異データ解析の手順 シークエンス 変異検出 マッピング データの精査 解釈 2 CLC Genomics Workbench 使用ツール シークエンスデータのインポート NGS data import クオリティチェック QC for Sequencing Reads Trim Reads 参照ゲノム配列へのマッピング 再アライメント
NGS データ解析入門 Web セミナー : 変異解析編 1 NGS 変異データ解析の手順 シークエンス 変異検出 マッピング データの精査 解釈 2 CLC Genomics Workbench 使用ツール シークエンスデータのインポート NGS data import クオリティチェック QC for Sequencing Reads Trim Reads 参照ゲノム配列へのマッピング 再アライメント
PowerPoint Presentation
 エピジェノミクス解析編 2016/08/10 Filgen ChIP-seq (Transfactor & Histone), Bisulfite webex seminar 株式会社キアゲンアプライドアドバンストゲノミクス宮本真理, PhD 1 アジェンダ ChIP-seq 解析 Transcription Factor ChIP-seq Histone ChIP-seq Bisulfite-seq
エピジェノミクス解析編 2016/08/10 Filgen ChIP-seq (Transfactor & Histone), Bisulfite webex seminar 株式会社キアゲンアプライドアドバンストゲノミクス宮本真理, PhD 1 アジェンダ ChIP-seq 解析 Transcription Factor ChIP-seq Histone ChIP-seq Bisulfite-seq
Microsoft PowerPoint - Ion Reporter?ソフトウェアを用いた変異解析4.6.pptx
 Ion Reporter ソフトウェアデモンストレーション Ion AmpliSeq Comprehensive Cancer Panel を用いたがん部および非がん部の体細胞変異比較解析 1 Ion Torrent システムを用いた実験例 Ion AmpliSeq Comprehensive Cancer Panel を 2 サンプル実施 ランレポート ランレポート サンプル 1 サンプル 2 2
Ion Reporter ソフトウェアデモンストレーション Ion AmpliSeq Comprehensive Cancer Panel を用いたがん部および非がん部の体細胞変異比較解析 1 Ion Torrent システムを用いた実験例 Ion AmpliSeq Comprehensive Cancer Panel を 2 サンプル実施 ランレポート ランレポート サンプル 1 サンプル 2 2
ChIP-seq
 ChIP-seq 1 ChIP-seq 解析原理 ChIP サンプルのフラグメントでは タンパク質結合部位付近にそれぞれ Forward と Reverse のリードがマップされることが予想される ChIP のサンプルでは Forward と Reverse のリードを 3 側へシフトさせ ChIP のピークを算出する コントロールサンプルでは ChIP のサンプルとは異なり 特定の場所に多くマップされないため
ChIP-seq 1 ChIP-seq 解析原理 ChIP サンプルのフラグメントでは タンパク質結合部位付近にそれぞれ Forward と Reverse のリードがマップされることが予想される ChIP のサンプルでは Forward と Reverse のリードを 3 側へシフトさせ ChIP のピークを算出する コントロールサンプルでは ChIP のサンプルとは異なり 特定の場所に多くマップされないため
Copyright 2008 NIFTY Corporation All rights reserved. 2
 Copyright 2008 NIFTY Corporation All rights reserved. 2 Copyright 2008 NIFTY Corporation All rights reserved. 3 Copyright 2008 NIFTY Corporation All rights reserved. 4 Copyright 2008 NIFTY Corporation
Copyright 2008 NIFTY Corporation All rights reserved. 2 Copyright 2008 NIFTY Corporation All rights reserved. 3 Copyright 2008 NIFTY Corporation All rights reserved. 4 Copyright 2008 NIFTY Corporation
NGSハンズオン講習会
 207.08.08 版 プラスアルファの内容です NGS 解析 ( 初 ~ 中級 ) ゲノムアセンブリ後の各種解析の補足資料 ( プラスアルファ ) 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム寺田朋子 門田幸二 Aug 29-30 207 Contents Gepard でドットプロット 連載第 8 回 W5-3 で最も長い sequence 同士のドットプロットを実行できなかったが
207.08.08 版 プラスアルファの内容です NGS 解析 ( 初 ~ 中級 ) ゲノムアセンブリ後の各種解析の補足資料 ( プラスアルファ ) 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム寺田朋子 門田幸二 Aug 29-30 207 Contents Gepard でドットプロット 連載第 8 回 W5-3 で最も長い sequence 同士のドットプロットを実行できなかったが
GettingStartedTK2
 T-Kernel 2.0 Getting Started T-Engine フォーラム ダウンロード T-Engine フォーラムの web ページから T-Kernel 2.0 Software Package をダウンロード 必要なソフトウェアや開発環境が全て含まれている 2 Copyright 2011 by T-Engine Forum, All Rights Reserved. 1 ReadMe.txt
T-Kernel 2.0 Getting Started T-Engine フォーラム ダウンロード T-Engine フォーラムの web ページから T-Kernel 2.0 Software Package をダウンロード 必要なソフトウェアや開発環境が全て含まれている 2 Copyright 2011 by T-Engine Forum, All Rights Reserved. 1 ReadMe.txt
ハピタス のコピー.pages
 Copyright (C) All Rights Reserved. 10 12,500 () ( ) ()() 1 : 2 : 3 : 2 4 : 5 : Copyright (C) All Rights Reserved. Copyright (C) All Rights Reserved. Copyright (C) All Rights Reserved. Copyright (C) All
Copyright (C) All Rights Reserved. 10 12,500 () ( ) ()() 1 : 2 : 3 : 2 4 : 5 : Copyright (C) All Rights Reserved. Copyright (C) All Rights Reserved. Copyright (C) All Rights Reserved. Copyright (C) All
Copyright 2008 All Rights Reserved 2
 Copyright 2008 All Rights Reserved 1 Copyright 2008 All Rights Reserved 2 Copyright 2008 All Rights Reserved 3 Copyright 2008 All Rights Reserved 4 Copyright 2008 All Rights Reserved 5 Copyright 2008 All
Copyright 2008 All Rights Reserved 1 Copyright 2008 All Rights Reserved 2 Copyright 2008 All Rights Reserved 3 Copyright 2008 All Rights Reserved 4 Copyright 2008 All Rights Reserved 5 Copyright 2008 All
IonTorrent RNA-Seq 解析概要 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science
 IonTorrent RNA-Seq 解析概要 2017-03 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science 資料概要 この資料は IonTorrent シーケンサーで RNA-Seq (WholeTranscriptome mrna ampliseqrna mirna) 解析を実施されるユーザー様向けの内容となっています
IonTorrent RNA-Seq 解析概要 2017-03 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science 資料概要 この資料は IonTorrent シーケンサーで RNA-Seq (WholeTranscriptome mrna ampliseqrna mirna) 解析を実施されるユーザー様向けの内容となっています
ひまわり8号データ利用手順データダウンロード方法について
 ひまわり 8 号評価用データ公開 データダウンロード手順について NICT サイエンスクラウド運営事務局 2015.02.19 目次 1 ひまわり 8 号データについて... 1 1.1 データダウンロードについて... 1 1.2 データ公開形式について... 2 1.3 データの展開について... 3 2 データダウンロード手順について... 4 2.1 ブラウザ経由のアクセス... 4 2.2
ひまわり 8 号評価用データ公開 データダウンロード手順について NICT サイエンスクラウド運営事務局 2015.02.19 目次 1 ひまわり 8 号データについて... 1 1.1 データダウンロードについて... 1 1.2 データ公開形式について... 2 1.3 データの展開について... 3 2 データダウンロード手順について... 4 2.1 ブラウザ経由のアクセス... 4 2.2
Maser - User Operation Manual
 Maser 3 Cell Innovation User Operation Manual 2013.4.1 1 目次 1. はじめに... 3 1.1. 推奨動作環境... 3 2. データの登録... 4 2.1. プロジェクトの作成... 4 2.2. Projectへのデータのアップロード... 8 2.2.1. HTTPSでのアップロード... 8 2.2.2. SFTPでのアップロード...
Maser 3 Cell Innovation User Operation Manual 2013.4.1 1 目次 1. はじめに... 3 1.1. 推奨動作環境... 3 2. データの登録... 4 2.1. プロジェクトの作成... 4 2.2. Projectへのデータのアップロード... 8 2.2.1. HTTPSでのアップロード... 8 2.2.2. SFTPでのアップロード...
GWB
 NGS データ解析入門 Web セミナー : 変異解析編 1 NGS 変異データ解析の手順 シークエンス 変異検出 マッピング データの精査 解釈 2 解析ワークフローと使用ソフトウェア シークエンスデータのインポート クオリティチェック 参照ゲノム配列へのマッピング 再アライメント 変異検出 CLC Genomics Workbench または Biomedical Genomics Workbench
NGS データ解析入門 Web セミナー : 変異解析編 1 NGS 変異データ解析の手順 シークエンス 変異検出 マッピング データの精査 解釈 2 解析ワークフローと使用ソフトウェア シークエンスデータのインポート クオリティチェック 参照ゲノム配列へのマッピング 再アライメント 変異検出 CLC Genomics Workbench または Biomedical Genomics Workbench
次世代シークエンサーを用いたがんクリニカルシークエンス解析
 次世代シークエンサーを用いた がんクリニカルシークエンス解析 フィルジェン株式会社バイオサイエンス部 ([email protected]) 1 がん遺伝子パネル がん関連遺伝子のターゲットシークエンス用のアッセイキット コストの低減や 研究プログラムの簡素化に有用 網羅的シークエンス解析の場合に比べて 1 遺伝子あたりのシークエンス量が増えるため より高感度な変異の検出が可能 2 変異データ解析パイプライン
次世代シークエンサーを用いた がんクリニカルシークエンス解析 フィルジェン株式会社バイオサイエンス部 ([email protected]) 1 がん遺伝子パネル がん関連遺伝子のターゲットシークエンス用のアッセイキット コストの低減や 研究プログラムの簡素化に有用 網羅的シークエンス解析の場合に比べて 1 遺伝子あたりのシークエンス量が増えるため より高感度な変異の検出が可能 2 変異データ解析パイプライン
PowerPoint プレゼンテーション
 Computer simulations create the future 2015 年度第 1 回 AICS 公開ソフト講習会 K MapReduce ハンズオン 滝澤真一朗松田元彦丸山直也 理化学研究所計算科学研究機構プログラム構成モデル研究チーム 1 RIKEN ADVANCED INSTITUTE FOR COMPUTATIONAL SCIENCE KMR の導入方法を学ぶ 目的 KMRRUN
Computer simulations create the future 2015 年度第 1 回 AICS 公開ソフト講習会 K MapReduce ハンズオン 滝澤真一朗松田元彦丸山直也 理化学研究所計算科学研究機構プログラム構成モデル研究チーム 1 RIKEN ADVANCED INSTITUTE FOR COMPUTATIONAL SCIENCE KMR の導入方法を学ぶ 目的 KMRRUN
Microsoft Word - SSI_Smart-Trading_QA_ja_ doc
 サイゴン証券会社 (SSI) SSI Smarttrading の設定に関する Q&A 06-2009 Q&A リスト 1. Q1 http://smarttrading.ssi.com.vn へアクセスしましたが 黒い画面になり X のマークが左上に出ている A1 原因はまだ設定していない アドミニストレータで設定しない あるいは自動設定プログラムがお客様の PC に適合しないと考えられます 解決方法アドミニストレータの権限のユーザーでログインし
サイゴン証券会社 (SSI) SSI Smarttrading の設定に関する Q&A 06-2009 Q&A リスト 1. Q1 http://smarttrading.ssi.com.vn へアクセスしましたが 黒い画面になり X のマークが左上に出ている A1 原因はまだ設定していない アドミニストレータで設定しない あるいは自動設定プログラムがお客様の PC に適合しないと考えられます 解決方法アドミニストレータの権限のユーザーでログインし
ソフトウェアについて Rev 年 1 月 16 日 このマニュアルでは標準でインストールしているソフトウェアの入手元 インストール方法の概要 インストール場所 についてご案内致します ABySS
 このマニュアルでは標準でインストールしているソフトウェアの入手元 インストール方法の概要 インストール場所 についてご案内致します ABySS http://www.bcgsc.ca/platform/bioinfo/software/abyss/ abyss 1.3.4.tar.gz tar xvzf abyss 1.3.4.tar.gz cd abyss 1.3.4./configure prefix=/usr/local/abyss
このマニュアルでは標準でインストールしているソフトウェアの入手元 インストール方法の概要 インストール場所 についてご案内致します ABySS http://www.bcgsc.ca/platform/bioinfo/software/abyss/ abyss 1.3.4.tar.gz tar xvzf abyss 1.3.4.tar.gz cd abyss 1.3.4./configure prefix=/usr/local/abyss
Slide 1
 Copyright 2008, Oracle. All rights reserved. 1 Oracle ORION version 11.1 for Linux x86-64 とりあえず ORION でテストをしたい人向け 基盤技術 SC 本部 - GRID Center 岩本知博 Create: 2008/12/04 Update: 2008/12/19
Copyright 2008, Oracle. All rights reserved. 1 Oracle ORION version 11.1 for Linux x86-64 とりあえず ORION でテストをしたい人向け 基盤技術 SC 本部 - GRID Center 岩本知博 Create: 2008/12/04 Update: 2008/12/19
Microsoft PowerPoint - set_up_bigip_ve_trial_mod.pptx[読み取り専用]
![Microsoft PowerPoint - set_up_bigip_ve_trial_mod.pptx[読み取り専用] Microsoft PowerPoint - set_up_bigip_ve_trial_mod.pptx[読み取り専用]](/thumbs/87/95528602.jpg) BIG-IP IP LTM Trial セットアップガイド F5 ネットワークスジャパン株式会社 INDEX BIG-IP VE Trial セットアップガイド 1. 準備 2. BIG-IP IP VE Trial インストール 3. BIG-IP VE Trial Setup 4. Information 5. 参考 : 構成例 Copyright 2009 All rights reserved
BIG-IP IP LTM Trial セットアップガイド F5 ネットワークスジャパン株式会社 INDEX BIG-IP VE Trial セットアップガイド 1. 準備 2. BIG-IP IP VE Trial インストール 3. BIG-IP VE Trial Setup 4. Information 5. 参考 : 構成例 Copyright 2009 All rights reserved
Slide 1
 NGS をはじめよう!RNA-Seq 入門 ( キットの選び方 実験デザイン ) April 18, 2014 米田瑞穂イルミナ株式会社テクニカルアプリケーションサイエンティスト 2012 Illumina, Inc. All rights reserved. Illumina, illuminadx, BaseSpace, BeadArray, BeadXpress, cbot, CSPro, DASL,
NGS をはじめよう!RNA-Seq 入門 ( キットの選び方 実験デザイン ) April 18, 2014 米田瑞穂イルミナ株式会社テクニカルアプリケーションサイエンティスト 2012 Illumina, Inc. All rights reserved. Illumina, illuminadx, BaseSpace, BeadArray, BeadXpress, cbot, CSPro, DASL,
HeartCoreインストールマニュアル
 HeartCore インストールマニュアル (JSP 版 ) October2013 Ver1.1-1 - 改訂履歴 改訂日 改訂内容 Ver1.0 2013 年 07 月 マニュアル改訂 Ver1.1 2013 年 10 月 フォーマット改訂 - 2 - 目次 1. 本文書の目的と対象...- 4-1.1. 概要説明... - 4-2. インストールの流れ...- 4-3. MySQL ユーザの作成...-
HeartCore インストールマニュアル (JSP 版 ) October2013 Ver1.1-1 - 改訂履歴 改訂日 改訂内容 Ver1.0 2013 年 07 月 マニュアル改訂 Ver1.1 2013 年 10 月 フォーマット改訂 - 2 - 目次 1. 本文書の目的と対象...- 4-1.1. 概要説明... - 4-2. インストールの流れ...- 4-3. MySQL ユーザの作成...-
PowerPoint Presentation
 : ソフトウェアのインストール Development Hub COBOL Server セットアップファイルのダウンロード Eclipse 版 セットアップファイルのダウンロード ソフトウェア要件の確認 ソフトウェア要件の確認 ソフトウェアのインストール ソフトウェアのインストール ライセンス認証 (DevHub COBOL Server 版のライセンスを利用 ) ライセンス認証 (Eclipse
: ソフトウェアのインストール Development Hub COBOL Server セットアップファイルのダウンロード Eclipse 版 セットアップファイルのダウンロード ソフトウェア要件の確認 ソフトウェア要件の確認 ソフトウェアのインストール ソフトウェアのインストール ライセンス認証 (DevHub COBOL Server 版のライセンスを利用 ) ライセンス認証 (Eclipse
CubePDF ユーザーズマニュアル
 CubePDF ユーザーズマニュアル 2018.11.22 第 13 版 1 1. PDF への変換手順 CubePDF は仮想プリンターとしてインストールされます そのため Web ブラウザや Microsoft Word, Excel, PowerPoint など印刷ボタンのあるアプリケーションであればどれでも 次の 3 ステップで PDF へ変換することができます 1. PDF 化したいものを適当なアプリケーションで表示し
CubePDF ユーザーズマニュアル 2018.11.22 第 13 版 1 1. PDF への変換手順 CubePDF は仮想プリンターとしてインストールされます そのため Web ブラウザや Microsoft Word, Excel, PowerPoint など印刷ボタンのあるアプリケーションであればどれでも 次の 3 ステップで PDF へ変換することができます 1. PDF 化したいものを適当なアプリケーションで表示し
Microsoft PowerPoint - InfPro_I6.pptx
 今日の学習内容 ファイルとディレクトリ ( 続 )(pp.34-36) 色々なUNIXのコマンド (pp.203-209) 今日の基礎教養セミナ講演会 Yahooの坂本さん 3コマ (13:10~) A204 ディレクトリ ( 復習 ) UNIXファイルシステムにはファイルとディレクトリがあり ツリー状の階層型構造をしている ツリー構造の頂点 ( ツリー構造の根の部分 ) 一番上の唯一のディレクトリをルートディレクトリと呼ぶ
今日の学習内容 ファイルとディレクトリ ( 続 )(pp.34-36) 色々なUNIXのコマンド (pp.203-209) 今日の基礎教養セミナ講演会 Yahooの坂本さん 3コマ (13:10~) A204 ディレクトリ ( 復習 ) UNIXファイルシステムにはファイルとディレクトリがあり ツリー状の階層型構造をしている ツリー構造の頂点 ( ツリー構造の根の部分 ) 一番上の唯一のディレクトリをルートディレクトリと呼ぶ
エンドポイント濁度測定装置 LT-16 取扱説明書
 エンドポイント濁度測定装置 LT-16 LT-16 Manager マニュアル ( 簡易マニュアル Version 2.0) LT-16 Manager のインストール LT-16 Manager は添付の CD に内蔵されています LT-16 Manager は Windows 7 Windows 8 において動作確認をしております ( 以下の図は Windows 8 使用時の表示図面です ) ただし
エンドポイント濁度測定装置 LT-16 LT-16 Manager マニュアル ( 簡易マニュアル Version 2.0) LT-16 Manager のインストール LT-16 Manager は添付の CD に内蔵されています LT-16 Manager は Windows 7 Windows 8 において動作確認をしております ( 以下の図は Windows 8 使用時の表示図面です ) ただし
POWER EGG V2.01 ユーザーズマニュアル 汎用申請編
 POWER EGG V2.0 ユーザーズマニュアル汎用申請編 Copyright 2009 D-CIRCLE,INC. All Rights Reserved 2009.4 はじめに 本書では POWER EGG 利用者向けに POWER EGG の汎用申請機能に関する操作を説明します 当マニュアルでは 汎用申請機能に関する説明のみ行います ファイルの添付など POWER EGG 全体の共通操作については
POWER EGG V2.0 ユーザーズマニュアル汎用申請編 Copyright 2009 D-CIRCLE,INC. All Rights Reserved 2009.4 はじめに 本書では POWER EGG 利用者向けに POWER EGG の汎用申請機能に関する操作を説明します 当マニュアルでは 汎用申請機能に関する説明のみ行います ファイルの添付など POWER EGG 全体の共通操作については
HeartCoreインストールマニュアル(PHP版)
 HeartCore インストールマニュアル (PHP 版 ) October 2013 Ver1.1-1 - 改訂履歴 改訂日 改訂内容 Ver1.0 2013 年 07 月 新規作成 Ver1.1 2013 年 10 月 フォーマット改訂 - 2 - 目次 1. 本文書の目的と対象... - 4-1.1. 概要説明... - 4-2. インストールの流れ... - 4-3. 定義ファイルの確認...
HeartCore インストールマニュアル (PHP 版 ) October 2013 Ver1.1-1 - 改訂履歴 改訂日 改訂内容 Ver1.0 2013 年 07 月 新規作成 Ver1.1 2013 年 10 月 フォーマット改訂 - 2 - 目次 1. 本文書の目的と対象... - 4-1.1. 概要説明... - 4-2. インストールの流れ... - 4-3. 定義ファイルの確認...
PowerPoint プレゼンテーション
 平成 28 年度 NGS ハンズオン講習会 NGS 解析基礎 2016 年 7 月 25 日 最近のシーケンサ MiniSeq MiSeq NextSeq HiSeq HiSeqX IonPGM IonProton Sequel PacBio MinION Amelieff Corporation All Rights Reserved 2 目次 NGSデータ解析で主に使用するファイル形式 データの可視化
平成 28 年度 NGS ハンズオン講習会 NGS 解析基礎 2016 年 7 月 25 日 最近のシーケンサ MiniSeq MiSeq NextSeq HiSeq HiSeqX IonPGM IonProton Sequel PacBio MinION Amelieff Corporation All Rights Reserved 2 目次 NGSデータ解析で主に使用するファイル形式 データの可視化
PowerPoint Presentation
 製品ソフトウェアのセットアップ手順 UNIX/Linux 編 1. セットアップファイルの選択開発環境 / 実行環境 / バージョン /Hotfix/ インストール先 OS 2. 対象セットアップファイルのダウンロード開発環境の場合は 2 つのファイルが対象 3. ソフトウェア要件の確認 4. ソフトウェアのインストール 5. ライセンスの認証 1 1. セットアップファイルの選択 選択項目選択肢該当チェック
製品ソフトウェアのセットアップ手順 UNIX/Linux 編 1. セットアップファイルの選択開発環境 / 実行環境 / バージョン /Hotfix/ インストール先 OS 2. 対象セットアップファイルのダウンロード開発環境の場合は 2 つのファイルが対象 3. ソフトウェア要件の確認 4. ソフトウェアのインストール 5. ライセンスの認証 1 1. セットアップファイルの選択 選択項目選択肢該当チェック
演習 レシピテキストの係り受け解析
 実習 : レシピの言語処理の現状 京都大学 笹田鉄郎 前田浩邦 森信介 2013 年 8 月 18 日 1 公開に際しての注意 必要環境 Perl KyTea Eda Firefox (ver. 14.0.1 以前のバージョン ) 著作権の関係上 係り受け解析の実習で利用した学習コーパスを公開することはできません ご了承ください 目次 1. はじめに 2. 注意事項 3. アノテーション支援ツールPNAT
実習 : レシピの言語処理の現状 京都大学 笹田鉄郎 前田浩邦 森信介 2013 年 8 月 18 日 1 公開に際しての注意 必要環境 Perl KyTea Eda Firefox (ver. 14.0.1 以前のバージョン ) 著作権の関係上 係り受け解析の実習で利用した学習コーパスを公開することはできません ご了承ください 目次 1. はじめに 2. 注意事項 3. アノテーション支援ツールPNAT
PLQ-20 取扱説明書 詳細編
 2013 Seiko Epson Corporation. All rights reserved. o n h o n n A B o C h h n h A B n C n n A B C A B C A B C D E A B C D E h o h B n C A D E F G n A C B n A B C D C n A B D F G H E n A B D C E F n A h
2013 Seiko Epson Corporation. All rights reserved. o n h o n n A B o C h h n h A B n C n n A B C A B C A B C D E A B C D E h o h B n C A D E F G n A C B n A B C D C n A B D F G H E n A B D C E F n A h
初心者にもできるアメブロカスタマイズ新2016.pages
 Copyright All Rights Reserved. 41 Copyright All Rights Reserved. 60 68 70 6 78 80 Copyright All Rights Reserved. FC2 97 Copyright All Rights Reserved. Copyright All Rights Reserved. Copyright All Rights
Copyright All Rights Reserved. 41 Copyright All Rights Reserved. 60 68 70 6 78 80 Copyright All Rights Reserved. FC2 97 Copyright All Rights Reserved. Copyright All Rights Reserved. Copyright All Rights
再起動した状態になり パスワードを入力すると 図 2 のように DEXCS2011 のアイコ ンがデスクトップ上に表示される 2 端末を準備する メニューバーにある端末の形を左クリック 図 2 デスクトップ メニューバーに端末の形がない場合 図 3 メニューバー アプリケーション アクセサリー 端末
 OpenFOAM の使い方準備編 目次 DALAB 鍔田 12/7/14 1. DEXCS2011 をインストール 2. OpenFOAM-2.1.1 を DEXCS に導入する 3. 例題を実行する 4. ParaView でのマウスによる操作方法 5. Tab キーの活用 6. 講習に使用するファイルのダウンロード この構築は Windows7(64bit メモリ :4GB) のパソコン上の仮想マシン
OpenFOAM の使い方準備編 目次 DALAB 鍔田 12/7/14 1. DEXCS2011 をインストール 2. OpenFOAM-2.1.1 を DEXCS に導入する 3. 例題を実行する 4. ParaView でのマウスによる操作方法 5. Tab キーの活用 6. 講習に使用するファイルのダウンロード この構築は Windows7(64bit メモリ :4GB) のパソコン上の仮想マシン
AJACS18_ ppt
 1, 1, 1, 1, 1, 1,2, 1,2, 1 1 DDBJ 2 AJACS3 2010 6 414:20-15:20 2231 DDBJ DDBJ DDBJ DDBJ NCBI (GenBank) DDBJ EBI (EMBL-Bank) GEO DDBJ Omics ARchive(DOR) ArrayExpress DTA (DDBJ Trace Archive) DRA (DDBJ
1, 1, 1, 1, 1, 1,2, 1,2, 1 1 DDBJ 2 AJACS3 2010 6 414:20-15:20 2231 DDBJ DDBJ DDBJ DDBJ NCBI (GenBank) DDBJ EBI (EMBL-Bank) GEO DDBJ Omics ARchive(DOR) ArrayExpress DTA (DDBJ Trace Archive) DRA (DDBJ
はじめに 面的評価支援システム操作マニュアル ( 別冊 ) 国土地理院数値地図 25000( 空間データ基盤 ) 変換編 は 国土地理院の HP よりダウンロードした数値地図 25000( 空間データ基盤 ) の地図データを 面的評価支援システム 用に変換するツールの使用方法についてまとめたものです
 面的評価支援システム 操作マニュアル ( 別冊 ) 国土地理院数値地図 25000( 空間データ基盤 ) 変換編 Ver 4.0.0 平成 28 年 3 月 環境省水 大気環境局自動車環境対策課 はじめに 面的評価支援システム操作マニュアル ( 別冊 ) 国土地理院数値地図 25000( 空間データ基盤 ) 変換編 は 国土地理院の HP よりダウンロードした数値地図 25000( 空間データ基盤
面的評価支援システム 操作マニュアル ( 別冊 ) 国土地理院数値地図 25000( 空間データ基盤 ) 変換編 Ver 4.0.0 平成 28 年 3 月 環境省水 大気環境局自動車環境対策課 はじめに 面的評価支援システム操作マニュアル ( 別冊 ) 国土地理院数値地図 25000( 空間データ基盤 ) 変換編 は 国土地理院の HP よりダウンロードした数値地図 25000( 空間データ基盤
- 2 Copyright (C) 2006. All Rights Reserved.
 - 2 Copyright (C) 2006. All Rights Reserved. 2-3 Copyright (C) 2006. All Rights Reserved. 70-4 Copyright (C) 2006. All Rights Reserved. ...1...3...7...8 1...9...14...16 2...18...20...21 3...22...23...23...24
- 2 Copyright (C) 2006. All Rights Reserved. 2-3 Copyright (C) 2006. All Rights Reserved. 70-4 Copyright (C) 2006. All Rights Reserved. ...1...3...7...8 1...9...14...16 2...18...20...21 3...22...23...23...24
thermofisher.com mirVana miRNA mimics/inhibitors 検索マニュアル
 thermofisher.com mirvana mirna mimics/inhibitors 検索マニュアル 2018 年 10 月版 The world leader in serving science mirna mimics/inhibitors 製品ラインナップ mirna mimics / inhibitors の製品ライナップ : Mimics : Gain-of-function
thermofisher.com mirvana mirna mimics/inhibitors 検索マニュアル 2018 年 10 月版 The world leader in serving science mirna mimics/inhibitors 製品ラインナップ mirna mimics / inhibitors の製品ライナップ : Mimics : Gain-of-function
MSDM_User_Manual_v0.2.1-B-1
 MAPLE Submission Data Maker Ver. 1.0 インストールマニュアル JAMSTEC 11/15/2018 目次 1. はじめに... 3 2. 動作環境... 3 3. Windows OS 上での MSDM のインストール... 4 3.1. VirtualBox のインストール... 4 3.2. MSDM 仮想サーバーのセットアップ... 6 4. Mac OS
MAPLE Submission Data Maker Ver. 1.0 インストールマニュアル JAMSTEC 11/15/2018 目次 1. はじめに... 3 2. 動作環境... 3 3. Windows OS 上での MSDM のインストール... 4 3.1. VirtualBox のインストール... 4 3.2. MSDM 仮想サーバーのセットアップ... 6 4. Mac OS
<4D F736F F F696E74202D20352D335F8D5C90AC CF909482CC90B690AC82C695D28F572E707074>
 RD_301 構成要素一覧と検索 から構成要素の編集辞書 ( 削除 ) を作る 作成 ( 編集 ) する削除辞書を開きます 構成要素を検索します ドラック & ドロップでも OK 範囲を選択して右クリック 右クリック 削除辞書に登録 ( 追加 ) したい構成要素を選択しコピーします 削除辞書に追加 ( 貼りつけ ) ます Step5. 削除辞書に構成要素が登録 ( 追加 ) されます 構成要素一覧と検索
RD_301 構成要素一覧と検索 から構成要素の編集辞書 ( 削除 ) を作る 作成 ( 編集 ) する削除辞書を開きます 構成要素を検索します ドラック & ドロップでも OK 範囲を選択して右クリック 右クリック 削除辞書に登録 ( 追加 ) したい構成要素を選択しコピーします 削除辞書に追加 ( 貼りつけ ) ます Step5. 削除辞書に構成要素が登録 ( 追加 ) されます 構成要素一覧と検索
Sophos Enterprise Console
 スタートアップガイド 製品バージョン : 5.5 次 このガイドについて...1 システム要件... 2 Linux コンピュータの保護... 3 動による Sophos Anti-Virus の新規インストール... 3 インストールパッケージの作成...3 インストールパッケージを使 した Sophos Anti-Virus のインストール...5 UNIX コンピュータの保護... 6 動による
スタートアップガイド 製品バージョン : 5.5 次 このガイドについて...1 システム要件... 2 Linux コンピュータの保護... 3 動による Sophos Anti-Virus の新規インストール... 3 インストールパッケージの作成...3 インストールパッケージを使 した Sophos Anti-Virus のインストール...5 UNIX コンピュータの保護... 6 動による
勉強会の流れ Google API の概要 デモ curl で実際に体験 Copyright 2010 SRA OSS, Inc. Japan All rights reserved. 2
 Google API 勉強会 SRA OSS, Inc. 日本支社山本博之 2010 年 12 月 13 日 勉強会の流れ Google API の概要 デモ curl で実際に体験 Copyright 2010 SRA OSS, Inc. Japan All rights reserved. 2 Sylpheed Pro 2.0 Sylpheed + Sylpheed Pro アドオンメール全文検索スケジューラ
Google API 勉強会 SRA OSS, Inc. 日本支社山本博之 2010 年 12 月 13 日 勉強会の流れ Google API の概要 デモ curl で実際に体験 Copyright 2010 SRA OSS, Inc. Japan All rights reserved. 2 Sylpheed Pro 2.0 Sylpheed + Sylpheed Pro アドオンメール全文検索スケジューラ
情報処理概論(第二日目)
 実習資料 Linux 入門講習会 九州大学情報基盤研究開発センター 注意 : この内容は najima.cc.kyushu-u.ac.jp の任意の ID で利用できますが, ファイルの削除等を含んでいるので各コマンドの意味を理解するまでは講習会用 ID で利用することをお勧めします. 1 実習 1 ログイン ファイル操作 ディレクトリの作成 ファイルの移動, コピー, 削除 ログアウト 2 ログイン
実習資料 Linux 入門講習会 九州大学情報基盤研究開発センター 注意 : この内容は najima.cc.kyushu-u.ac.jp の任意の ID で利用できますが, ファイルの削除等を含んでいるので各コマンドの意味を理解するまでは講習会用 ID で利用することをお勧めします. 1 実習 1 ログイン ファイル操作 ディレクトリの作成 ファイルの移動, コピー, 削除 ログアウト 2 ログイン
Copyright 2006 KDDI Corporation. All Rights Reserved page1
 Copyright 2006 KDDI Corporation. All Rights Reserved page1 Copyright 2006 KDDI Corporation. All Rights Reserved page2 Copyright 2006 KDDI Corporation. All Rights Reserved page3 Copyright 2006 KDDI Corporation.
Copyright 2006 KDDI Corporation. All Rights Reserved page1 Copyright 2006 KDDI Corporation. All Rights Reserved page2 Copyright 2006 KDDI Corporation. All Rights Reserved page3 Copyright 2006 KDDI Corporation.
HeartCore(PHP 版 ) インストール手順について説明いたします なお 本資料は 例として下記内容を前提として説明しております 環境情報 対象 OS: Linux ( ディストリビューション : Red Hat Enterprise Linux Server) APサーバ : Apache
 HeartCore インストール手順手順書 (PHP 版 ) Jul 2010 Ver1.0-1 - HeartCore(PHP 版 ) インストール手順について説明いたします なお 本資料は 例として下記内容を前提として説明しております 環境情報 対象 OS: Linux ( ディストリビューション : Red Hat Enterprise Linux Server) APサーバ : Apache
HeartCore インストール手順手順書 (PHP 版 ) Jul 2010 Ver1.0-1 - HeartCore(PHP 版 ) インストール手順について説明いたします なお 本資料は 例として下記内容を前提として説明しております 環境情報 対象 OS: Linux ( ディストリビューション : Red Hat Enterprise Linux Server) APサーバ : Apache
PowerPoint プレゼンテーション
 一括で商品編集する場合 オプション情報の修正 Update 2018-07 Copyright (C) Qoo10 Japan All Rights Reserved. 1 目次 どこからできますか? オプションとは - 組合型オプションと単一型オプションについて オプション一括修正 1. 登録済みオプションの情報をダウンロード 2. 編集したファイルをアップロードして修正 楽天 CSVを利用したオプション一括登録
一括で商品編集する場合 オプション情報の修正 Update 2018-07 Copyright (C) Qoo10 Japan All Rights Reserved. 1 目次 どこからできますか? オプションとは - 組合型オプションと単一型オプションについて オプション一括修正 1. 登録済みオプションの情報をダウンロード 2. 編集したファイルをアップロードして修正 楽天 CSVを利用したオプション一括登録
1. 主な機能追加項目 以下の検索項目をサポートしました 書誌 全文検索コマンド検索 国内 査定日 最新の査定日 ( 登録査定日または拒絶査定日 ) を検索します 査定種別 最新の登録 拒絶査定 または査定なしを検索します 審査最終処分日 最新の審査最終処分日を検索します 審査最終処分種別 最新の審
 日立特許情報提供サービス Sharerese arch 01-76Ver. 機能のご紹介 2011,HitachiInformationSystems,LtdAllRigh tsrese rved 1. 主な機能追加項目 以下の検索項目をサポートしました 書誌 全文検索コマンド検索 国内 査定日 最新の査定日 ( 登録査定日または拒絶査定日 ) を検索します 査定種別 最新の登録 拒絶査定 または査定なしを検索します
日立特許情報提供サービス Sharerese arch 01-76Ver. 機能のご紹介 2011,HitachiInformationSystems,LtdAllRigh tsrese rved 1. 主な機能追加項目 以下の検索項目をサポートしました 書誌 全文検索コマンド検索 国内 査定日 最新の査定日 ( 登録査定日または拒絶査定日 ) を検索します 査定種別 最新の登録 拒絶査定 または査定なしを検索します
Microsoft PowerPoint - 6_TS-0891(TS-0835(Custom TaqMan Assay Design Tool利用方法修正5.pptx
 Custom TaqMan Assay Design Tool インターネットオーダー方法 20010/06/01 Custom TaqMan Assay Design Tool 1. Custom TaqMan SNP Genotyping Assays P.5 2. Custom TaqMan Gene Expression Assays P.21 3. カスタムデザインでのオーダー P.30
Custom TaqMan Assay Design Tool インターネットオーダー方法 20010/06/01 Custom TaqMan Assay Design Tool 1. Custom TaqMan SNP Genotyping Assays P.5 2. Custom TaqMan Gene Expression Assays P.21 3. カスタムデザインでのオーダー P.30
Copyright All Rights Reserved. -2 -!
 http://ameblo.jp/admarketing/ Copyright All Rights Reserved. -2 -! Copyright All Rights Reserved. -3- Copyright All Rights Reserved. -4- Copyright All Rights Reserved. -5 - Copyright All Rights Reserved.
http://ameblo.jp/admarketing/ Copyright All Rights Reserved. -2 -! Copyright All Rights Reserved. -3- Copyright All Rights Reserved. -4- Copyright All Rights Reserved. -5 - Copyright All Rights Reserved.
Microsoft Word - CBSNet-It連携ガイドver8.2.doc
 (Net-It Central 8.2) 本ガイドでは ConceptBase Search Lite.1.1 と Net-It Central 8.2 の連携手順について説明します 目次 1 はじめに...2 1.1 本書について...2 1.2 前提条件...2 1.3 システム構成...2 2 ConceptBase のインストール...3 2.1 インストールと初期設定...3 2.2 動作確認...3
(Net-It Central 8.2) 本ガイドでは ConceptBase Search Lite.1.1 と Net-It Central 8.2 の連携手順について説明します 目次 1 はじめに...2 1.1 本書について...2 1.2 前提条件...2 1.3 システム構成...2 2 ConceptBase のインストール...3 2.1 インストールと初期設定...3 2.2 動作確認...3
XMLとXSLT
 XML と XSLT 棚橋沙弥香 目次 現場のシステム構成とXML/XSLの位置づけ XMLとは XSL/XSLTとは Xalanのインストール いろいろなXSL XMLマスター試験の紹介 現場のシステム構成 HTML 画面上のデータ 電文 電文 外部 WEB サーバー (Java) CORBA 通信 認証サーバー (C 言語 ) DB XML 電文 HTML XSL XSLT 変換今回の説明範囲
XML と XSLT 棚橋沙弥香 目次 現場のシステム構成とXML/XSLの位置づけ XMLとは XSL/XSLTとは Xalanのインストール いろいろなXSL XMLマスター試験の紹介 現場のシステム構成 HTML 画面上のデータ 電文 電文 外部 WEB サーバー (Java) CORBA 通信 認証サーバー (C 言語 ) DB XML 電文 HTML XSL XSLT 変換今回の説明範囲
Microsoft Word - XOOPS インストールマニュアルv12.doc
 XOOPS インストールマニュアル ( 第 1 版 ) 目次 1 はじめに 1 2 XOOPS のダウンロード 2 3 パッケージの解凍 4 4 FFFTP によるファイルアップロード手順 5 5 ファイルアップロード後の作業 11 6 XOOPS のインストール 15 7 インストール後の作業 22 8 XOOPS ログイン後の作業 24 愛媛県総合教育センター情報教育研究室 Ver.1.0.2
XOOPS インストールマニュアル ( 第 1 版 ) 目次 1 はじめに 1 2 XOOPS のダウンロード 2 3 パッケージの解凍 4 4 FFFTP によるファイルアップロード手順 5 5 ファイルアップロード後の作業 11 6 XOOPS のインストール 15 7 インストール後の作業 22 8 XOOPS ログイン後の作業 24 愛媛県総合教育センター情報教育研究室 Ver.1.0.2
Hphi実行環境導入マニュアル_v1.1.1
 HΦ の計算環境構築方法マニュアル 2016 年 7 月 25 日 東大物性研ソフトウェア高度化推進チーム 目次 VirtualBox を利用した HΦ の導入... 2 VirtualBox を利用した MateriAppsLive! の導入... 3 MateriAppsLive! への HΦ のインストール... 6 ISSP スパコンシステム B での HΦ の利用方法... 8 各種ファイルの置き場所...
HΦ の計算環境構築方法マニュアル 2016 年 7 月 25 日 東大物性研ソフトウェア高度化推進チーム 目次 VirtualBox を利用した HΦ の導入... 2 VirtualBox を利用した MateriAppsLive! の導入... 3 MateriAppsLive! への HΦ のインストール... 6 ISSP スパコンシステム B での HΦ の利用方法... 8 各種ファイルの置き場所...
Microsoft Word - 最終版 バックせどりismマニュアル .docx
 ism ISM ISM ISM ISM ISM ISM Copyright (c) 2010 All Rights Reserved. Copyright (c) 2010 All Rights Reserved. Copyright (c) 2010 All Rights Reserved. ISM Copyright (c) 2010 All Rights Reserved. Copyright
ism ISM ISM ISM ISM ISM ISM Copyright (c) 2010 All Rights Reserved. Copyright (c) 2010 All Rights Reserved. Copyright (c) 2010 All Rights Reserved. ISM Copyright (c) 2010 All Rights Reserved. Copyright
3.1. Velvet は velveth velvetg の 2 つのプログラムから 構 成 されており 設 定 画 面 でそれぞれのパラメーターを 設 定 可 能 です 3.2. Velvetg については より 詳 細 なパラメーターを 設 定 可 能 です 3.3. Multiplex 解
 SEQUENCHER V5.1 の 特 長 株 式 会 社 日 立 ソリューションズ SEQUENCER V5.1 で 新 規 に 追 加 された 機 能 や 改 善 された 機 能 について 以 下 にご 紹 介 いたします 追 加 項 目 および 改 善 項 目 は 項 番 14 16 27 28 32 33 34 を 除 き Windows 版 Macintosh 版 共 に 同 様 の 内
SEQUENCHER V5.1 の 特 長 株 式 会 社 日 立 ソリューションズ SEQUENCER V5.1 で 新 規 に 追 加 された 機 能 や 改 善 された 機 能 について 以 下 にご 紹 介 いたします 追 加 項 目 および 改 善 項 目 は 項 番 14 16 27 28 32 33 34 を 除 き Windows 版 Macintosh 版 共 に 同 様 の 内
ダウンロード方法 アルテラのソフトウェアをインストールするためのダウンロード ファイルには以下の種類があります.tar フォーマットのソフトウェアとデバイス ファイルがバンドルされたセット ダウンロードとインストールをカスタマイズするための個別の実行ファイル ディスクに焼いて他の場所にインストールす
 Quartus Prime ソフトウェア ダウンロードおよびインストール クイック スタート ガイド 2015 Altera Corporation. All rights reserved. ALTERA, ARRIA, CYCLONE, ENPIRION, MAX, MEGACORE, NIOS, QUARTUS and STRATIX words and logos are trademarks
Quartus Prime ソフトウェア ダウンロードおよびインストール クイック スタート ガイド 2015 Altera Corporation. All rights reserved. ALTERA, ARRIA, CYCLONE, ENPIRION, MAX, MEGACORE, NIOS, QUARTUS and STRATIX words and logos are trademarks
Solid Edge ST10 新機能紹介
 Solid Edge ST10 新機能紹介 2018 年 2 月 All Rights Reserved, Copyright ITOUCHU TECHNO-SOLUTIONS Corporation 2018 目次 パーツ ジェネレーティブデザイン ( 位相最適化 ) リバースエンジニアリング メッシュベースモデリング ボディを調節 シンクロナスでのブレンド削除を強化 シートメタル 切り抜きがある曲げ部の移動
Solid Edge ST10 新機能紹介 2018 年 2 月 All Rights Reserved, Copyright ITOUCHU TECHNO-SOLUTIONS Corporation 2018 目次 パーツ ジェネレーティブデザイン ( 位相最適化 ) リバースエンジニアリング メッシュベースモデリング ボディを調節 シンクロナスでのブレンド削除を強化 シートメタル 切り抜きがある曲げ部の移動
PowerPoint プレゼンテーション
 文献管理ツール EndNote 利用法 ユサコ株式会社 原沢竜太 Copyright(C) 2015 USACO Corporation. All Rights Reserved. EndNote basic について EndNote basic とは 文献管理 Web of Scienceなどのデータベースから検索した文献情報を保存 論文作成支援 引用表記 参考文献リストの 自動作成が可能 *EndNote
文献管理ツール EndNote 利用法 ユサコ株式会社 原沢竜太 Copyright(C) 2015 USACO Corporation. All Rights Reserved. EndNote basic について EndNote basic とは 文献管理 Web of Scienceなどのデータベースから検索した文献情報を保存 論文作成支援 引用表記 参考文献リストの 自動作成が可能 *EndNote
自己紹介 : プロフィール 石井一夫 ( 東京農工大学特任教授 ) 専門分野 : ゲノム科学 バイオインフォマティクス データマイニング 計算機統計学 経歴 : 徳島大学大学院医学研究科博士課程修了後 東京大学医科学研究所ヒトゲノム解析センターリサーチアソシエート 理化学研究所ゲノム科学総合研究セン
 フリーソフトによるゲノム科学におけるビッグデータ解析の実際 石井一夫東京農工大学農学系ゲノム科学人材育成プログラム 1 自己紹介 : プロフィール 石井一夫 ( 東京農工大学特任教授 ) 専門分野 : ゲノム科学 バイオインフォマティクス データマイニング 計算機統計学 経歴 : 徳島大学大学院医学研究科博士課程修了後 東京大学医科学研究所ヒトゲノム解析センターリサーチアソシエート 理化学研究所ゲノム科学総合研究センター研究員
フリーソフトによるゲノム科学におけるビッグデータ解析の実際 石井一夫東京農工大学農学系ゲノム科学人材育成プログラム 1 自己紹介 : プロフィール 石井一夫 ( 東京農工大学特任教授 ) 専門分野 : ゲノム科学 バイオインフォマティクス データマイニング 計算機統計学 経歴 : 徳島大学大学院医学研究科博士課程修了後 東京大学医科学研究所ヒトゲノム解析センターリサーチアソシエート 理化学研究所ゲノム科学総合研究センター研究員
目次 1. 動作環境チェック 動作必要環境 Java のインストール Java のインストール Firebird のインストール Firebird のインストール Adobe Reader のインストール
 ORCA PROJECT Linux 対応版インストールマニュアル (Version 2.0.0 対応 ) Ubuntu 10.04 Lucid 用 2.0.0 版 2013 年 3 月 8 日 目次 1. 動作環境チェック...3 1.1. 動作必要環境...3 2. Java のインストール...3 2.1. Java のインストール...3 3. Firebird のインストール...4 3.1.
ORCA PROJECT Linux 対応版インストールマニュアル (Version 2.0.0 対応 ) Ubuntu 10.04 Lucid 用 2.0.0 版 2013 年 3 月 8 日 目次 1. 動作環境チェック...3 1.1. 動作必要環境...3 2. Java のインストール...3 2.1. Java のインストール...3 3. Firebird のインストール...4 3.1.
Ontrack EasyRecovery 11 基本的な使い方
 1.Ontrack EasyRecovery 11 の基本的な使い方 1Ontrack EasyRecovery 11 を起動し 画面右下の 次へ をクリックします ここの画面 操作は Ontrack EasyRecovery 11 Home for Windows を使用した例となります 2 下記の メディアの種類を選択 の画面になりますので 復旧対象のディスクを選択し 次へ をクリックしてください
1.Ontrack EasyRecovery 11 の基本的な使い方 1Ontrack EasyRecovery 11 を起動し 画面右下の 次へ をクリックします ここの画面 操作は Ontrack EasyRecovery 11 Home for Windows を使用した例となります 2 下記の メディアの種類を選択 の画面になりますので 復旧対象のディスクを選択し 次へ をクリックしてください


