農学生命情報科学特論I
|
|
|
- たつや きせんばる
- 5 years ago
- Views:
Transcription
1 版ほぼ完成です 最終回のレポート課題はありません 基本的な考え方と解析戦略の変遷 ( スライド 3-29 あたり ) は確実に省略しますので講義前に 3-29 については自分で見ておいてください スライド についても残り時間次第です 最終回ですので アンケートのほうもよろしくお願いします 農学生命情報科学特論 I 第 4 回 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム 2 微生物科学イノベーション連携研究機構門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp July 3, 208
2 講義予定 第 回 (208 年 06 月 2 日 ) カウント情報取得の続き データの正規化 (RPK, RPM, RPKM/FPKM) 第 2 回 (208 年 06 月 9 日 ) サンプル間クラスタリング R のクラスオブジェクト R の Reference Manual の読み解き方 クラスタリング結果の客観的な評価 第 3 回 (208 年 06 月 26 日 ) 発現変動解析 ( 反復あり / なしの 2 群間比較 ) M-A plot 発現変動解析 :3 群間比較 デザイン行列 第 4 回 (208 年 07 月 03 日 ) 機能解析 ( 発現変動遺伝子セット解析 ) GSEA MSigDB GSVA の実行 July 3, 208 2
3 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3, 208 3
4 機能解析 Gene Ontology (GO) 解析 ( 発現に差のある GO term を探索 ) 基本 3 カテゴリ (Cellular Component (CC), Molecular Function (MF), Biological Process (BP)) のどれでも可能 例 : 肝臓の空腹状態 vs. 満腹状態の GO(BP) 解析の結果 脂肪酸 β 酸化 関連 GO term (GO: ) が動いていることが分かった パスウェイ解析 ( 発現に差のあるパスウェイを探索 ) KEGG Pathway, BioCarta, Reactome pathway database のどれでも可能 例 : 酸化的リン酸化パスウェイ関連遺伝子セットが糖尿病患者で動いていた モチーフ解析 ( 発現に差のあるモチーフを探索 ) 同じ 3 -UTR microrna 結合モチーフをもつ遺伝子セット 同じ転写因子結合領域 (TATA-box など ) をもつ遺伝子セット 機能解析の実体は 遺伝子セットの発現変動解析 発現に差のある遺伝子セットを探したい ということ 例 :TATA-box をもつ遺伝子セットが G 群 vs. G2 群比較で動いていた July 3, 208 4
5 機能解析の全体像 で機能解析 ( 発現変動遺伝子セット解析 ) に関する全体像を述べています July 3, 208 5
6 機能解析の全体像 第 世代の機能解析 (ORA) 2GSEA が含まれる第 2 世代の機能解析 (FCS) ORA や FCS の用語を含む詳細については後述 と書いてありますが講義では省きます 2 July 3, 208 6
7 遺伝子セット次第で ここがキモ 2 遺伝子セット次第で GO 解析や Pathway 解析にもなり得る 2 July 3, 208 7
8 MSigDB MSigDB というサイトで 遺伝子セット情報の.gmt という拡張子のついたファイルが提供されています July 3, 208 8
9 MSigDB MSigDB というサイトで 遺伝子セット情報の.gmt という拡張子のついたファイルが提供されています July 3, 208 9
10 gmt ファイルを入手 MSigDB というサイトで 遺伝子セット情報の.gmt という拡張子のついたファイルを予め入手しておかねばならない July 3, 208 0
11 入力ファイルは 2 つ 発現変動遺伝子検出の場合は 入力が つ ( 発現行列データ ) であった 発現変動遺伝子セット解析の場合は 発現データファイルに加えて どの遺伝子がどの遺伝子セットに属するかという情報を含む gmt ファイルも必要です July 3, 208
12 様々なプログラムがある 2005 年の GSEA 論文発表前後を含めて 様々な方法が提案されています July 3, 208 2
13 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3, 208 3
14 N =0,000 genes 基本的な考え方 発現変動遺伝子セット解析手法 (2 群間比較用がほとんど ) N =0,000 個の遺伝子からなる 2 群間比較用データ この中に XXX 関連遺伝子が n 個含まれている 例 : 酸化的リン酸化 (=XXX) 関連遺伝子が 7(=n) 個含まれている 酸化的リン酸化関連遺伝子セットが変動しているかどうかを調べたい という問題を考える G 群 G2 群 7 個の酸化的リン酸化関連遺伝子の位置 July 3, 208 4
15 N =0,000 genes 基本的な考え方 遺伝子ごとの発現変動の度合いを数値化 例 :t- 統計量 log2(g2/g) 相関係数 基本はデフォルトでやるようですが 様々な選択肢があります G 群 G2 群 G 群 G2 群 発現変動遺伝子 (G >> G2) 変動していない遺伝子 (G == G2) 発現変動遺伝子 (G << G2) July 3, 208 5
16 N =0,000 genes 基本的な考え方 発現変動順にソート後の酸化的リン酸化関連遺伝子セットのステレオタイプな分布 どうやって偏りを評価するのか? 変動している 変動していない G 群 G2 群 G 群 G2 群 July 3, 208 6
17 N =0,000 genes 基本的な考え方 Over-Representation Analysis (ORA) 何らかの手段で決めた上位 X(=500) 個のうち x 個が酸化的リン酸化関連遺伝子であった 基本的な考え方は 全遺伝子 と 上位のサブセット のみで 調べたい遺伝子セットの割合が不変という帰無仮説のもとで検定 酸化的リン酸化関連遺伝子セット (n =7) が変動していない場合 : x/n X/N (= 500/0000) 酸化的リン酸化関連遺伝子セット (n =7) が変動している場合 : x/n >> X/N (= 5%) G 群 G2 群 G 群 G2 群 July 3, 208 7
18 N =0,000 genes ORA Over-Representation Analysis (ORA) 何らかの手段で決めた上位 X(=500) 個のうち x 個が酸化的リン酸化関連遺伝子であった 2 2 分割表 (contingency table) に基づく方法 超幾何検定やカイ二乗検定が利用されます G 群 G2 群 G 群 G2 群 6 XXX= 酸化的リン酸化関連遺伝子セット July 3, 208 8
19 rcode_ora_basic.txt ORA( 超幾何検定 ) DEG として 500 個抽出したとき 酸化的リン酸化関連遺伝子が 6 個以上含まれる確率として算出 N=0000 個の遺伝子発現データ中に XXX= 酸化的リン酸化関連遺伝子は n=7 個含まれていた 上位 X=500 個の発現変動遺伝子 (DEG) の中に x=6 個の酸化的リン酸化関連遺伝子が含まれていた 帰無仮説 : 酸化的リン酸化関連遺伝子の割合は DEG と non-deg 間で差がない ORA として Fisher s hypergeometric test を利用 (Tavazoie et al., Nat Genet., 22: , 999) July 3, 208 9
20 rcode_ora_basic.txt ORA( 超幾何検定 )?dhyper マニュアル中の一般的な説明に置き換えるとこんな感じ m=7 個の白いボールと n=9993 個の黒いボールが入った箱があります ( トータルで N=m+n=0,000 個 ) この中から k=500 個ランダムに取り出したときに x=6 個以上白いボールが含まれる確率を計算しなさい July 3,
21 rcode_ora_basic.txt ORA( カイ二乗検定 ) DEG として 500 個抽出したとき 酸化的リン酸化関連遺伝子が 6 個以上含まれる確率として算出 July 3, 208 2
22 rcode_ora_basic.txt 直感は重要 N=0000 個の遺伝子発現データ中に XXX= 酸化的リン酸化関連遺伝子は n=7 個存在する 2 上位 X=500 個の発現変動遺伝子 (DEG) の中に x=6 個の酸化的リン酸化関連遺伝子が含まれていた 3 帰無仮説 : 酸化的リン酸化関連遺伝子の割合は DEG と non-deg 間で差がない の段階で 調べたい遺伝子セットは 7/0,000 = 0.07% の割合だと考える 2 で 6/,500 = 0.4% の割合に濃縮されていると考える 3 今やっているのは ある 2 群間比較 もし比較している群間で この遺伝子セットが全体として発現変動していなかったとしたら ランダムで,500 個とった時に この遺伝子セット中の遺伝子が含まれる割合は 0.07% なので 個数だと % =.05 個程度しか含まれないはず 実際に得られたのは 6 個なので 偶然こんな結果が得られたとは考えにくい 起こるとしたら 4 くらい低い確率なんだね だから 発現変動遺伝子セット と考えよう という思考回路 4 July 3,
23 N =0,000 genes ORA Over-Representation Analysis (ORA) 上位 500 個のうち 酸化的リン酸化関連遺伝子が 7 個中 4 つ以上含まれていれば p < 0.05 で検出可能ということを意味する G 群 G2 群 G 群 G2 群 p < 0.05 を灰色で示した July 3,
24 ORA Over-Representation Analysis (ORA) GenMAPP (Dahlquist et al., Nature Genet., 3: 9-20, 2002) FatiGO (Al-Shahrour et al., Bioinformatics, 20: , 2004) GOstat (Beissbarth et al., Bioinformatics, 20: , 2004) GOFFA (Sun et al., BMC Bioinformatics, 7 Suppl 2: S23, 2006) agrigo (Du et al., Nucleic Acids Res., 38: W64-W70, 200) GenMAPP は比較的有名 July 3,
25 N =0,000 genes 第 世代 (ORA) の短所 全体的には動いているものの 個々の発現変動の度合いが弱い場合に検出困難 2 上位 X 個の X 次第で結果が変わる 3 情報量低下 ( 発現変動の度合い カウント情報 ) 3 G 群 G2 群 2 G 群 G2 群 July 3,
26 第 2 世代 (FCS) もちろん分割表ベースの方法 (ORA) ではない第 2 世代以降の方法があります Functional Class Scoring (FCS). 遺伝子ごとの統計量を算出 ( 発現変動の度合いを数値化 ) 例 :t- 統計量 log(g2/g) 相関係数 2. 目的の遺伝子セット XXX(= 酸化的リン酸化関連遺伝子 ) の偏りを何らかの方法で評価 t 検定 (XXX 中の遺伝子群の統計量 vs. それ以外の遺伝子群の統計量 ) Wilcoxon rank sum test (XXX 中の遺伝子群の発現変動の順位 vs. それ以外 ) XXX 中の n 個の遺伝子群の何らかの要約統計量 S XXX を計算しておき N 個の全遺伝子の中からランダムに n 個を抽出して同じ統計量を計算する ( 例えば 0 万回 ) 0 万回のうち S XXX 以上 ( 大きければ大きいほど発現変動していることを意味する場合 ; その逆のときは 以下 ) だった回数 ( 例えば j 回 ) に基づいて p 値 (= j / 00,000) を算出 ( いわゆる gene set permutation というアプローチ ) 本来の G 群 vs. G2 群のラベル情報を用いて得られた XXX 中の n 個の遺伝子群の何らかの要約統計量 S XXX を計算しておく ランダムにラベル情報を入れ替えて 同じ統計量を計算することを何回も繰り返して p 値を算出 ( いわゆる Phenotype permutation というアプローチ ) July 3,
27 N =0,000 genes 第 2 世代 (FCS) 第一世代 (ORA) の欠点が改善遺伝子ごとの log 比で考えると 遺伝子を等価に取り扱うのではなく log 比そのものを足し込むことで 発現変動の度合いが大きいほどより大きな重みをかけるようなイメージ 全体的には動いているものの 個々の発現変動の度合いが弱い場合に検出困難 2 上位 X 個の X 次第で結果が変わる 3 情報量低下 ( 発現変動の度合い カウント情報 ) 3 G 群 G2 群 2 G 群 G2 群 July 3,
28 第 2 世代 (FCS) Functional Class Scoring (FCS) GSEA (Subramanian et al., PNAS, 02: , 2005) PAGE (Kim and Volsky, BMC Bioinformatics, 6: 44, 2005) sigpathway (Tian et al., PNAS, 02: , 2005) GSA (Efron and Tibshirani, Ann. Appl. Stat., : 07-29, 2007) GeneTrail (Backes et al., Nucleic Acids Res., 35: W86-W92, 2007) SAM-GS (Dinu et al., BMC Bioinformatics, 8: 242, 2007) 最も有名なのが GSEA ここでリストアップされているのは 基本的にマイクロアレイデータ解析用なので情報としては古い よって (R で ) 塩基配列解析ではほとんどリストアップしていない July 3,
29 遺伝子セット解析の課題 突っ込みどころは満載だが ネガティブなことばかりいってもしょうがないし この種の機能解析が目的の場合も多い ( 知識ベースの解析法なので ) 解析対象がアノテーションの情報の豊富な生物種に限定 それ以外の生物種は まずは地道にアノテーション情報を増やしていくことが先決 ( ではないだろうか ) アノテーションの解像度を上げる努力も大事 アノテーション情報の信頼度が高いとはいえない なんらかの GO term がついていたとしても その大部分の evidence code が自動でつけられたもの (IEA, inferrred from electronic annotations) である 遺伝子セット間の独立性の問題 数百個程度の遺伝子セットの中から 比較するサンプル間で動いている遺伝子セットはどれか? という解析を遺伝子セット間の独立性を仮定して調べるが そもそも独立ではない (GO term 間の親子関係などから明らか ) いくつくらいの遺伝子セットが動いているのか? という問いに答えるすべがない 評価に用いられる よく研究されているデータセット は答えが完全に分かっているものではない (the actual biology is never fully known!) 感度が高い と謳っているだけの方法は ( 全部の遺伝子セットが動いている 感度 00%) July 3, 208 Maciejewski H., Brief Bioinform., 4: ,
30 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
31 GO 解析用 遺伝子セットとして GO の情報を用いる場合 July 3, 208 3
32 GO 解析用 SeqGSA はマニュアルがないに等しい 2GSAR は 発現変動遺伝子セットを探すというよりは 興味ある遺伝子セットを与えてネットワーク図を描き どの遺伝子が hub(hub genes) かを返すのがメイン の講義資料でも解説しています でページ内検索してください 2 July 3,
33 EGSEA EGSEA は 2 複数のツールを組み合わせるやり方 様々な分野でこの種の戦略がよいことは実証されており おそらく妥当 しかしその分だけ依存関係が複雑になるため 私もまだ試してはいない 2 July 3,
34 SeqGSEA SeqGSEA は 手順が煩雑な上 ものすごく計算時間がかかる の講習会資料作成当時の個人の感想です July 3,
35 GSVA GSVA は 2EGSEA でも利用されている また 3 引用回数も多い ( 使いやすい ) ので 後半はこれをベースに説明 2 July 3,
36 パスウェイ解析用 遺伝子セットとして KEGG Pathway や Reactome などの情報を用いる場合 July 3,
37 パスウェイ解析用 GO 解析もできるのに ここにしか記載していないものもいくつかあるはずですのでご注意ください July 3,
38 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
39 遺伝子セット情報取得 遺伝子セット情報は GMT という形式 ( 拡張子が.gmt) で提供されており 23 つの手段で取得可能です 2 July 3,
40 MSigDB ver. 6. 最も有名なのは MSigDB 2208 年 6 月現在の ver. 6. では 8 個の主要なコレクションが提供されている 2 July 3,
41 MSigDB ver. 6.. H: hallmark gene sets (50 gene sets) 2. c: positional gene sets (326 gene sets) ヒト染色体の位置ごとの遺伝子セットリストファイル (326 gene sets) 3. c2: curated gene sets (4,738 gene sets) CGP: chemical and genetic perturbations (3,409 gene sets) CP: canonical pathways (,329 gene sets) CP:BIOCARTA: BioCarta gene sets (27 gene sets) CP:KEGG: KEGG gene sets (86 gene sets) CP:REACTOME: Reactome gene sets (674 gene sets) 4. c3: motif gene sets (836 gene sets) MIR: microrna targets (22 gene sets) TFT: transcription factor targets (65 gene sets) 5. c4: computational gene sets (858 gene sets) CGM: cancer gene neighborhoods (427 gene sets) CM: cancer modules (43 gene sets) 6. c5: gene ontology (GO) gene sets (5,97 gene sets) BP: biological process (4,436 gene sets) CC: cellular component (580 gene sets) MF: molecular function (90 gene sets) 7. c6: oncogenic signatures gene sets (89 gene sets) 8. c7: immunologic signatures gene sets (4,872 gene sets) 発現変動と関連する KEGG パスウェイを調べたいときは 3 番目の c2 というカテゴリーに属する CP:KEGG というところの 86 個の遺伝子セットが含まれる.gmt ファイルを予めダウンロードしておく July 3, 208 Subramanian et al., PNAS, 02: ,
42 MSigDB ver. 6.. H: hallmark gene sets (50 gene sets) 2. c: positional gene sets (326 gene sets) ヒト染色体の位置ごとの遺伝子セットリストファイル (326 gene sets) 3. c2: curated gene sets (4,738 gene sets) CGP: chemical and genetic perturbations (3,409 gene sets) CP: canonical pathways (,329 gene sets) CP:BIOCARTA: BioCarta gene sets (27 gene sets) CP:KEGG: KEGG gene sets (86 gene sets) CP:REACTOME: Reactome gene sets (674 gene sets) 4. c3: motif gene sets (836 gene sets) MIR: microrna targets (22 gene sets) TFT: transcription factor targets (65 gene sets) 5. c4: computational gene sets (858 gene sets) CGM: cancer gene neighborhoods (427 gene sets) CM: cancer modules (43 gene sets) 6. c5: gene ontology (GO) gene sets (5,97 gene sets) BP: biological process (4,436 gene sets) CC: cellular component (580 gene sets) MF: molecular function (90 gene sets) 7. c6: oncogenic signatures gene sets (89 gene sets) 8. c7: immunologic signatures gene sets (4,872 gene sets) 発現変動と関連する GO の biological process を調べたいときは 6 番目の c5 というカテゴリーに属する BP というところの 4,436 個の遺伝子セットが含まれる.gmt ファイルを予めダウンロードしておく July 3,
43 MSigDB ver. 6.. H: hallmark gene sets (50 gene sets) 2. c: positional gene sets (326 gene sets) ヒト染色体の位置ごとの遺伝子セットリストファイル (326 gene sets) 3. c2: curated gene sets (4,738 gene sets) CGP: chemical and genetic perturbations (3,409 gene sets) CP: canonical pathways (,329 gene sets) CP:BIOCARTA: BioCarta gene sets (27 gene sets) CP:KEGG: KEGG gene sets (86 gene sets) CP:REACTOME: Reactome gene sets (674 gene sets) 4. c3: motif gene sets (836 gene sets) MIR: microrna targets (22 gene sets) TFT: transcription factor targets (65 gene sets) 5. c4: computational gene sets (858 gene sets) CGM: cancer gene neighborhoods (427 gene sets) CM: cancer modules (43 gene sets) 6. c5: gene ontology (GO) gene sets (5,97 gene sets) BP: biological process (4,436 gene sets) CC: cellular component (580 gene sets) MF: molecular function (90 gene sets) 7. c6: oncogenic signatures gene sets (89 gene sets) 8. c7: immunologic signatures gene sets (4,872 gene sets) 講義で利用する解析プログラム GSVA の検証用としては 326 遺伝子セットが最適なので これをダウンロードしておきます July 3,
44 ダウンロード MSigDB 本家からもダウンロードできますが 一気にやると迷惑をかけるので 2 から c.all.v6..entrez.gmt をダウンロードしておいてください Entrez gene ID のヒト染色体の位置ごとの遺伝子セットリストファイル (326 gene sets) です 3 c.all.v6..symbols.gmt もダウンロードしておきましょう 3 2 July 3,
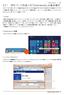
45 c.all.v6..entrez.gmt c.all.v6..entrez.gmt を Excel で眺めるとこんな感じ GMT 形式は 2 列目が gene set 名 32 列目が情報源の URL そして 43 列目以降が遺伝子セットを構成する Entrez gene IDs July 3,
46 c.all.v6..entrez.gmt chr5q23 が 番目のデータセット 2 赤枠部分が chr5q23 という遺伝子セットに含まれる遺伝子群の Entrez gene IDs 2 July 3,
47 chr5q23 最後の Entrez gene ID である 272 まで ずーっと右のほうに移動したところ 遺伝子セットによって 構成メンバー数が異なることがわかります 22 番目の遺伝子セット (chr6q24) は 番目の遺伝子の遺伝子セット (chr5q23) よりも構成メンバー数が多いことがわかります 2 July 3,
48 chr5q23 2 列目の情報源の URL を眺めると July 3,
49 chr5q23 2 列目の情報源の URL を眺めると 2 こんな感じで詳細情報が得られます 3 遺伝子セット chr5q23 のメンバー数は 84 genes であることがわかります 2 3 July 3,
50 Show members で をクリックすると July 3,
51 Show members で こんな感じになって Entrez gene ID に対応する 2gene symbol や 3gene description が見られます 2 3 July 3, 208 5
52 5759 は PTMAP2 番目のデータセット chr5q23 に含まれるメンバーで Entrez gene ID が の 3gene symbol は PTMAP2 であることがわかります July 3,
53 5759 は PTMAP2 参考 c.all.v6..symbols.gmt だとこんな感じになります このファイルは遺伝子セット情報を gene symbols で提供しているものなので妥当ですね July 3,
54 参考 326 gene sets なので 326 行目までで終わりです July 3,
55 326 gene sets なので c.all.v6..entrez.gmt でも当然同じです July 3,
56 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
57 MSigDB 提供ファイル MSigDB は Entrez gene IDs(.entrez.gmt) と Gene symbols(.symbols.gmt) の 2 種類を提供 当然 本家もそうなっています July 3,
58 機能解析の全体像 機能解析の全体像に関する説明では 2 のあたりで記載しています 2 July 3,
59 機能解析の全体像 手元の発現データの gene ID の種類に応じて 利用する遺伝子セットファイル (.entrez.gmt or.symbols.gmt ) を切り替えます この後で実行する GSVA パッケージの例題の発現データは Entrez gene ID なので c.all.v6..entrez.gmt をダウンロードしたのです July 3,
60 参考 gene symbols の場合 手元の発現データが Entrez gene ID 以外であり 例えば Ensembl gene ID だった場合は Ensembl gene ID と gene symbols の対応情報を取得しておきます そして 発現データのほうを gene symbols に変換しておいて.symbols.gmt ファイルを用いて遺伝子セット解析を行います July 3,
61 参考 gene symbols の場合 様々な ID の変換を行う際によく用いられる BioMart というデータベースを R 経由で実行可能な biomart という R パッケージもあります が話がそれていくので深入りしません July 3, 208 6
62 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
63 検証用データ 以前のスライドで遺伝子セット解析を実行するプログラムとして 2GSVA を採択した根拠を示した際に 赤枠内のプログラムについては言及したが 3 については言及しなかった 2 3 July 3,
64 Pickrell data 3 2 講義資料作成当時の私は 説明しやすい検証用データセットも探していた AbsFilterGSEA 論文中の 2 のあたりで 3Pickrell data という 2 群間比較用 (29 males vs. 40 females) のカウントデータが存在することを発見 July 3, 208 AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 64
65 MSigDB C この Pickrell data と 2MSigDB C カテゴリーの発現変動遺伝子セット解析結果として 3chryq がおそらく最上位に近い有意性を示し 4male 群で高発現であることまで分かった この段階で c.all.v6..entrez.gmt または c.all.v6..symbols.gmt が有望株だと認識 July 3, 208 AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 65
66 AbsFilterGSEA AbsFilterGSEA が使いやすければよいが July 3, 208 AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 66
67 AbsFilterGSEA 一般に CRAN 提供パッケージは Bioconductor 提供パッケージに比べて利用法の解読が難解です 実際私は 2 の Reference manual をみてガッカリし 他にわかりやすいパッケージはないか探した結果として GSVA を眺め 採用しました 2 July 3, 208 AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 67
68 GSVA 2GSVA は引用回数も多く 3Bioconductor のパッケージであったことが決め手 さらに解説 PDF を読むと Pickrell データも例題として使われていることが判明 3 をクリック 3 2 July 3,
69 GSVA をクリックすると 2Bioconductor から提供されているパッケージの場合はこんな感じになります 2 July 3,
70 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
71 GSVA 左側の赤枠部分を拡大表示 July 3, 208 7
72 GSVA GSVA のインストール手順の説明 2Reference Manual の PDF これは CRAN( しーらん ) にもあるやつです 2 July 3,
73 GSVA の解説 PDF GSVA の解説 PDF(bignettes; ビニェット ) これは Bioconductor のサイトに存在するものです GSVA インストール後は R 起動直後に 2 をコピペでも ( 手元の PC 内に存在する ) 解説 PDF を開けます 2 July 3,
74 GSVA の解説 PDF GSVA の解説 PDF(bignettes; ビニェット ) これは Bioconductor のサイトに存在するものです GSVA インストール後は R 起動直後に 2 をコピペでも ( 手元の PC 内に存在する ) 解説 PDF を開けます 3 こんな感じ 3 2 July 3,
75 GSVA の解説 PDF GSVA の解説 PDF(bignettes; ビニェット ) を起動して 利用法を学んでいく 例えば 2 のところを押すと 3 のところに飛びます 基本的には解説 PDF 内を順番にざっと読んで このパッケージの使用法のノリに慣れる 2 3 July 3,
76 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
77 MSigDB の C2 Application のあたりまでページ下部に移動 このパッケージは 遺伝子セット情報として 2MSigDB の C2 コレクションを利用していることがわかる 2 July 3,
78 MSigDB の C2 それは GSVAdata というパッケージに含まれる 2 c2broadsets という名前の 3GeneSetCollection オブジェクトだということがわかる 3 2 July 3,
79 MSigDB の C2 これをコピーして R コンソール画面上で コマンドのみペースト すれば確認できる July 3,
80 c2broadsets の確認 こんな感じでコピーして 2 コマンドのみペースト 2 July 3,
81 c2broadsets の確認 こんな感じになり GeneSetCollection という形式の c2broadsets を無事読み込めました July 3, 208 8
82 Tips: コマンドのみペースト コマンドのみペースト とすることで > などの余分な文字が誤ってコマンドとして認識されないようにすることができます July 3,
83 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
84 c.all.v6..entrez.gmt 今手元にあるのは c.all.v6..entrez.gmt gmt ファイルを読み込ませ 遺伝子セット情報を自在に変えて解析できるようになりたい! July 3,
85 ?c2broadsets?c2broadsets と打ち込んで このデータがどのような手順で作成されたのか手がかりを探る July 3,
86 ?c2broadsets こんな感じになります GSVAdata パッケージ中の 2c2BroadSets の説明のページという意味 2 July 3,
87 ?c2broadsets c2broadsets という名前の 2GeneSetCollection 形式のオブジェクトは 3GSEABase パッケージ内の 4 getgmt 関数を用いて 5c2.all.v3.0.entrez.gmt ファイルを読み込んで得られたものだということが分かる July 3,
88 gmt ファイルの読込 2 例題 の gmt ファイル読込の基本形を実行してみましょう 2 July 3,
89 例題 : 基本形 コピペ実行結果 無事 GeneSetCollection 形式の geneset オブジェクトが得られていることがわかります 2 確かに C コレクションは 326 遺伝子セットでした 3 つめの遺伝子セットが chr5q23 で 42 つめが chr6q24 となっています July 3,
90 c.all.v6..symbols.gmt 入力ファイル (c.all.v6..symbols.gmt) を Excel で眺めたところ 2 最初の 2 行の遺伝子セット名と同じであり 妥当ですね 2 July 3,
91 c.all.v6..symbols.gmt これは 入力が c.all.v6..symbols.gmt なので 2PTMAP2 や 3FTMT のような gene symbols で遺伝子セット情報が記載されている 2 3 July 3, 208 9
92 c.all.v6..symbols.gmt c.all.v6..symbols.gmt 内にある gene symbols は 2PTMAP2や3FTMTを含めて全部で何種類あるのだろうか? そのあたりの情報は 2 3 July 3,
93 例題 : 基本形 c.all.v6..symbols.gmt 内には 2PTMAP2 や 3 FTMT を含めて 4 全部で 30,00 種類あるのだろう July 3,
94 まだ不十分か?! 赤下線部分に着目! この部分が NullIdentifier や NullCollection となっている GSVAdata パッケージの 2c2BroadSets ではそうなっていなかった 2 July 3,
95 両者を比較 gmt ファイルから読み込んだ geneset では 2NullIdentifier や NullCollection となっている その一方で GSVAdata パッケージの 3c2BroadSets では 4EntrezIdentifier や BroadCollection となっている ここまでやっておく必要性については今のところ不明ではあるが 念のためやったのが例題 July 3,
96 例題 3 2 例題 3 です 2 July 3,
97 例題 3 例題 3 の 2 入力は c.all.v6..entrez.gmt 2 July 3,
98 例題 3 getgmt 関数実行時に 2geneIdType と 3collectionType オプションを与えて Entrez gene ID であることや Broad institute 提供の C コレクションであることを明示しておけば 2 3 July 3,
99 例題 3 c.all.v6..entrez.gmt を読み込んで得られた 2geneset オブジェクトの中身が 3EntrezIdentifier や BroadCollection になります これで見た目上は GSVAdata パッケージの c2broadsets 同じような見栄えになりました 実際問題としてここまでやっておく必要があるかどうかはわかりません ここまでで gmt ファイルを読み込んで GeneSetCollection オブジェクトを作成するところまで完了 2 3 July 3,
100 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
101 GSVA の解説 PDF 4. Functional enrichment のところ 最初に発現データとして 2 マイクロアレイデータの leukemia_eset を見せている これは 3ExpressionSet という発現データを格納する形式です 42,626 features 37 samples のデータのようですね July 3, 208 0
102 ExpressionSet GSVA パッケージでは RNA-seq カウントデータも ExpressionSet 形式になっています まだクリックしない! July 3,
103 重複除去時の入力 このあと行う同一 gene ID の重複除去時の入力として ExpressionSet 形式の 2leukemia_eset が与えられています 3 今は 6 ページのあたり 2 3 July 3,
104 重複除去時の入力 7~8 ページにかけて 2ExpressionSet オブジェクトの leukemia_eset を入力として 3nsFilter 関数を用いた重複除去が行われています 3 2 July 3,
105 nsfilter で重複除去 マイクロアレイ時代を知るヒトは AFFX という文字のみで 2leukemia_eset が Affymetrix GeneChip データであることがわかる また 3 の記述から Entrez gene ID であることを前提とし 4 で重複した Entrez gene ID の除去を行っているらしいことがわかる この段階で 重複除去を nsfilter 関数を用いて行うためには RNA-seq カウントデータの場合も ExpressionSet オブジェクトにしないといけないので 若干テンションが下がる July 3,
106 GSVA の入力 重複除去の実行結果は filtered_eset 28 ページ目の上のほうです 2 July 3,
107 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
108 GSVA の入力 重複除去の実行結果は filtered_eset 28 ページ目の下のほうです 2 July 3,
109 ExpressionSet 形式 重複除去の実行結果は filtered_eset filtered_eset オブジェクト中の 2eset という部分の情報を抜き出した 3leukemia_filtered_eset が 4gsva 関数実行時の 5 入力のようです 3 leukemia_filtered_eset は ExpressionSet 形式です July 3,
110 GeneSetCollection 形式 c2broadsetsは GeneSetCollectionという形式の遺伝子セット情報です 2と3で解析する遺伝子セットのフィルタリングを指定しています 2は遺伝子セットを構成するメンバー数の下限 (minimum size) 3は上限 (maximum size) です どの遺伝子セット解析プログラムも 大抵このような遺伝子セットのフィルタリングを行います 従って 解析結果で見られる遺伝子セット数は 入力時よりも減るのが普通 2 3 July 3, 208 0
111 GSVA の入力形式 gsva の入力が 2ExpressionSet および 3GeneSetCollection という形式に限定されているかを 4?gsva で確認 July 3, 208
112 ?gsva こんな感じになります GSVA パッケージ中の 2gsva 関数の説明のページという意味 2 4 July 3, 208 2
113 ?gsva 何を書いてるのか (S4 method って何よ? とか ) 分かりづらいだろうが と 2 の比較から 2 4 July 3, 208 3
114 ?gsva 4 何を書いてるのか (S4 method って何よ? とか ) 分かりづらいだろうが と 2 の比較から 遺伝子セット情報は 3GeneSetCollection 形式以外に 4list 形式でもよいのだろう ということがわかる 3 2 July 3, 208 4
115 ?gsva 発現情報もまた ExpressionSet 以外に 2 matrix 形式でもよいことがわかる この結果から RNA-seq カウントデータの入力が通常のタブ区切りテキストファイルの場合は 基本そのまま読み込むのでよい ( 正確には as.matrix しないといけない ) と判断する 2 July 3, 208 5
116 おまけ 参考 遺伝子セットのフィルタリングは デフォルトでは行わない設定になっていることがわかる は遺伝子セットを構成するメンバー数の下限 ( minimum size) が 上限 (maximum size) が Inf になっているからです Inf は無限大の意味です July 3, 208 6
117 ?gsva 参考 このあたりにもちゃんと書いてますね July 3, 208 7
118 ?gsva kcdf オプションは この後の GSVA for RNA-seq data の記述を見てから気づくのが実際のところかもしれない 結論のみ述べると 2RNA-seq のカウントデータを入力とする場合は デフォルトの kcdf= Gaussian ではなく kcdf= Poisson で実行せねばならない 2 July 3, 208 8
119 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3, 208 9
120 おさらい 2GSVA の解説 PDF には Pickrell データも例題として使われています 実際に Pickrell データを眺めます 3 2 July 3,
121 GSVA for RNA-seq data をクリックすると 2GSVA for RNA-seq data のところに飛びます 2 July 3, 208 2
122 p4 この画面内で実際に行うのは GSVAdata パッケージをロードした後 2 をコピペ実行することのみ 3 は無視でよい 2 3 July 3,
123 補足説明 の論文ではマイクロアレイデータが そして 2 の論文では RNAseq データが取得されており 両者は比較可能な状態にあります そして 2 の RNA-seq データはさらに Argonne sequencing center と Yale sequencing center の 2 か所で独立に取得されています 3 は単純に 4 アレイデータと ( 画面上では見えていませんが )p6 の 行目で見られる Argonne sequencing center で得られた RNA-seq データの gene ID が完全に一致しているかどうかを featurenames 関数で gene ID 情報を取り出した後 identical 関数で比較しているだけです 若干説明が不十分かもしれませんし私も誤解している部分があるかもしれませんが このあたりは深入りする価値はありません July 3,
124 p5~6 参考 のあたりが p6 の最初のほうの記述内容です これもさらっと流すところ July 3,
125 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
126 p6 の上のほう 参考 のあたりも c2broadsets の中から 一部を抜き取っているだけ 個人的には なぜこれをやる必要があるのか理解不能 July 3,
127 C2 ではなく C 参考 c2broadsets の中から一部を抜き取って作成した canonicalc2broadsets は あくまでも C2 コレクションのデータの一部 その一方で 2 発現データの Pickrell data と MSigDB C コレクションの遺伝子セット解析で有意な発現変動を示したのは chryq であった July 3, AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 27
128 参考 性特異的セットを追加 canonicalc2broadsets のセットだけでは Pickrell data の解析結果を示しづらいので 22 つの sex-specific expression を示す遺伝子セットを追加すると書いています 2 July 3,
129 参考 性特異的セット つめ つめの sex-specific expression を示す遺伝子セットを MSY というセット名で作成したところ July 3,
130 参考 性特異的セット 2 つめ つめの sex-specific expression を示す遺伝子セットを MSY というセット名で作成したところ 22 つめの sex-specific expression を示す遺伝子セットを XiE というセット名で作成したところ 2 July 3,
131 参考 性特異的セットマージ 22つめのsex-specific expressionを示す遺伝子セットをxieというセット名で作成したところ 3 既存のcanonicalC2BroadSetsに対して 作成した2 つのsex-specific expressionを示す4msyと5 XiEを 6GeneSetCollectionという形式でマージした結果を 7 新たなcanonicalC2BroadSetsとする July 3, 208 3
132 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
133 GSVA の実行準備完了 やっと GSVA を実行できる状況になったようです July 3,
134 p7 の上のほう が GSVA 実行コマンドです July 3,
135 p7 の上のほう Argonne sequence center で取得された RNAseq カウントデータの ExpressionSet オブジェクトと 2sex-specific expression を示す 2 つの遺伝子セット (MSY と XiE) を追加した MSigDB C2 コレクションの GeneSetCollection オブジェクトが入力 2 July 3,
136 p7 の上のほう 3 GSVA 実行結果である esrnaseq は 806 行 36 列からなるデータ 2 この後は RNA-seq カウントデータを RPKM 値に変換してから 3 マイクロアレイデータの結果である esmicro との比較を行って ほら似た結果になってるでしょ でオシマイ sexspecific expression を示す 2 つの遺伝子セット (MSY と XiE) については 一応サンプルごとに算出した Enrichment Scores (GSVA scores) の散布図を示している そして男女間でサンプルごとのスコアが確かに異なっており その傾向はマイクロアレイデータでも RPKM データでも同じですね ということは述べられている しかしながら 有意な発現変動遺伝子セットはどれかを調べる枠組みはガイドラインも示されておらず残念 2 July 3,
137 p7 の中央あたり RPKM 値を算出する最初のほうを示しているところ のあたりで RPKM 値への変換に必要な配列長情報を含む annoentrez22022 を取得 2 がまず RPM(Reads Per Million) 値を算出しているところ cpm 関数を用いていますが これは (Counts Per Million) 値を算出するものであり 実質的に同じです 3 は 4cpm と annotentrez22022 で共通の gene ID のもの (intersection) を抽出しているだけです July 3,
138 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
139 発現行列データを整形して取得 順を追って説明をしますが 2 例題 7を実行しましょう 2 July 3,
140 例題 7 実行の下準備 話がややこしくなるので 一旦 R を再起動し 作業ディレクトリを Desktop に変更してから ls() を実行 これは利用可能なオブジェクトが何もないことを確認しているだけです 単に全員の環境を揃えているだけなので 意味が分かるヒトはやらなくてもよい July 3,
141 例題 7 赤枠内をコピペ GSVAdata パッケージ中にある発現データ commonpickrellhuang をロードし 2 どのようなオブジェクトが利用可能かを見るところまでです commonpickrellhuang には 3 種類の発現データが含まれています 3 アレイデータ および 4Argonne sequencing center と 5Yale sequencing center で取得された RNA-seq カウントデータです July 3, 208 4
142 列の並びはバラバラ 赤枠内をコピペ July 3,
143 列の並びはバラバラ Yale sequencing center で取得された RNA-seq カウントデータのオブジェクトを eset という名前で取り扱い 2 発現行列の列 (phenotype という意味で pdata) に関するメタデータ情報を確認したところ Gender 列が各サンプルの性別情報を含んでいると学習し そこを表示させた結果が 3 赤枠の最後のコマンド 列の並びは性別できれいに分離されていないことがわかる この段階で GSVA 実行結果を見やすくするために 予めソートしておこうと考える 2 3 July 3,
144 女 23 人 男 3 人 table 関数で内訳を調査 Female が 23 人 Male が 3 人 G 群 23 サンプル vs. G2 群 3 サンプル の 2 群間比較データだと読み替えてもよい July 3,
145 コード下部に移動 一番下までカーソル移動して最後の部分まで表示 July 3,
146 重複 gene ID の除去 オリジナルの発現行列 exprs(eset) に対して その行名からなるベクトル rownames(exprs(eset)) の要素数と 行名をユニークにした後のベクトルの要素数を表示 このデータの場合は = 26 個分の重複があったことがわかる この重複を除去してユニークな gene ID にしておかないと 保存 ( hoge7.txt の作成 ) 自体はできるが 次にそれを ( 私のウェブページ上の通常のやり方で ) 読み込むときに 重複した row.names は許されない! と言われる July 3,
147 nsfilter 関数の実行 重複除去を行う nsfilter 関数を実行 2 var.filter=f オプションがついているが こうしておかないと重複除去のみにならないからです 3 思い描いた通りの結果になっています 実際問題としては重複した Entrez gene IDs があった場合にどのような取り扱い ( 平均しているのかどうかなど ) を行っているかまでは調べきれていません 2 3 July 3,
148 列名変更の必要性 の後処理として列名変更をしなかった場合は 出力ファイル中の列名が 2 の実行結果のようになってしまいます 今は男女間での発現変動遺伝子セット解析を行いたいので 列名は Female or Male で充分 2 July 3,
149 列名変更後 赤枠内コピペ実行後 確かに列名変更できていることがわかります ただ 以前 pdata(eset)$gender でも確認したように Female と Male が性別順に並んでいないことがわかります July 3,
150 列名でソート 赤枠内コピペ実行後 確かに列名でソートできていることがわかります order 関数を用いた行列のソートテクは よく利用します July 3,
151 例題 7 を最後までコピペ後 hoge7.txt が作成されます July 3, 208 5
152 ,482 gene IDs hoge7.txt これが Entrez gene ID の重複除去を行って 性別ごとに列をソートした後の発現行列データ G 群 :23 females G2 群 :3 males July 3,
153 Contents 機能解析 ( 発現変動遺伝子セット解析 ) 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 July 3,
154 C コレクションで GSVA 2 遺伝子セット情報は MSigDB C コレクションの c.all.v6..entrez.gmt を利用します これは 326 遺伝子セットからなります 2 July 3,
155 C コレクションで GSVA 発現データファイルの中身は July 3,
156 ,482 gene IDs SRP00540_23_3.txt Entrez gene ID の重複除去を行い 性別ごとに列をソートした後の発現行列データ G 群 :23 females G2 群 :3 males July 3,
157 コピペ実行 コード全体をコピペ実行後 全部で 326 個の遺伝子セットの発現変動解析を行うべく 入力として与えたが 2 遺伝子セットのメンバー数が 5 以上 500 以下という条件でフィルタリングすると 3298 個の遺伝子セットになったようです 3 2 July 3,
158 実行結果ファイル 実行結果ファイルの hoge.txt をエクセルで眺めてみましょう July 3,
159 hoge.txt GSVA 自体は Enrichment score を返すだけのプログラム そのスコアからなる数値ベクトルを入力として ノンパラメトリックな Wilcoxon rank sum test (Mann-Whitney U test と同じもの ) で得られた p 値が最も低い発現変動遺伝子セットは 2chryq でした この結果は 2 July 3,
160 同じ結果が得られた AbsFilterGSEA の論文の結果と同じですね 2 chryq がおそらく最上位だと思われます 3 解析データがちょっと異なりますが それでも結果は同じ 今回の我々の実行結果も 確かに male で高発現になっています July 3, 208 AbsFilterGSEA(Yoon et al., PLoS One, : e06599, 206) 60
161 hoge.txt male で高発現というのは の部分の Enrichment score が male 群で高いという理解で正しいはずです July 3, 208 6
162 Contents 機能解析 ( 発現変動遺伝子セット解析 ) July 3, 208 全体像 基本的な考え方と解析戦略の変遷 様々なプログラム 遺伝子セット情報の取得 (gmtファイルの取得) 発現データ情報と遺伝子セット情報のIDの対応付け 検証用 RNA-seqカウントデータセットPickrell data( なぜGSVAにしたか ) GSVAの解説 PDFを読み解く ( 手元のc.all.v6..entrez.gmt をどう読み込ませるか ) GSVAdataパッケージ提供の MSigDB c2コレクションであるc2broadsetsを理解する 手元のgmtファイルを読み込ませて GeneSetCollection 形式で取り扱えるようにする GSVA の解説 PDF を読み解く ( 手元の発現データファイルをどう取り扱うか ) ExpressionSetの取り扱い nsfilter 関数を用いた同一 IDの重複除去 メインプログラムgsva 関数が入力として受け付けるデータ形式 (ExpressionSetとMatrix) 検証用 RNA-seqカウントデータセットPickrell dataのイントロ スルーしていいところ MSigDB c2コレクションに2つの性特異的遺伝子セットを追加したものでgsvaを実行 ユニークなEntrez gene IDで グループごとに分離させた発現データファイル作成 整形後の発現データファイルとc.all.v6..entrez.gmtを入力としてGSVAを実行 最後に 62
163 最後に rcode_pickrell.txt 内で Pickrell カウントデータファイル (SRP00540_23_3.txt) を入力として以下の三つを行っています : サンプル間クラスタリング ( 雄雌入り混じった状態 ) シルエットスコアの計算 ( 非常に 0 に近い値 ) DEG 検出 (0%FDR で 85 個を DEG と判定 ) このように一見 hopeless な印象を受ける結果であったとしても 遺伝子セット解析によってカナリ説得力のある結果が得られることもあるというのが面白いですね July 3,
機能ゲノム学
 USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 機能ゲノム学 第 4 回 前回 (5/26) の hoge フォルダがデスクトップに残っているかもしれないのでご注意ください 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/
USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 機能ゲノム学 第 4 回 前回 (5/26) の hoge フォルダがデスクトップに残っているかもしれないのでご注意ください 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/
機能ゲノム学(第6回)
 講義室後ろにある USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 機能ゲノム学第 4 回 東京大学大学院農学生命科学研究科 アグリバイオインフォマティクス教育研究ユニット 門田幸二 kadota@iu.a.u-tokyo.ac.jp 前回 (5/28) のhogeフォルダがデスクトップに残っているかもしれないのでご注意ください Jun 04, 2014 1 講義予定
講義室後ろにある USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 機能ゲノム学第 4 回 東京大学大学院農学生命科学研究科 アグリバイオインフォマティクス教育研究ユニット 門田幸二 kadota@iu.a.u-tokyo.ac.jp 前回 (5/28) のhogeフォルダがデスクトップに残っているかもしれないのでご注意ください Jun 04, 2014 1 講義予定
基本的な利用法
 (R で ) 塩基配列解析 基本的な利用法 Macintosh 版 到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける 1. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール 2. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握
(R で ) 塩基配列解析 基本的な利用法 Macintosh 版 到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける 1. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール 2. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握
Rインストール手順
 R 本体は最新のリリース版 R パッケージは 必要最小限プラスアルファ の推奨インストール手順を示します R 本体とパッケージのインストール Windows 版 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/
R 本体は最新のリリース版 R パッケージは 必要最小限プラスアルファ の推奨インストール手順を示します R 本体とパッケージのインストール Windows 版 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/
基本的な利用法
 到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握 ) 4. ありがちなミス のところで示しているエラーメッセージとその原因をきっちり理解
到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握 ) 4. ありがちなミス のところで示しているエラーメッセージとその原因をきっちり理解
NGS速習コース
 バイオインフォマティクス人材育成カリキュラム ( 次世代シークエンサ ) 速習コース 3. データ解析基礎 3-3. R 各種パッケージ 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 1 Contents
バイオインフォマティクス人材育成カリキュラム ( 次世代シークエンサ ) 速習コース 3. データ解析基礎 3-3. R 各種パッケージ 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 1 Contents
NGSハンズオン講習会
 207.08.08 版 プラスアルファの内容です NGS 解析 ( 初 ~ 中級 ) ゲノムアセンブリ後の各種解析の補足資料 ( プラスアルファ ) 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム寺田朋子 門田幸二 Aug 29-30 207 Contents Gepard でドットプロット 連載第 8 回 W5-3 で最も長い sequence 同士のドットプロットを実行できなかったが
207.08.08 版 プラスアルファの内容です NGS 解析 ( 初 ~ 中級 ) ゲノムアセンブリ後の各種解析の補足資料 ( プラスアルファ ) 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム寺田朋子 門田幸二 Aug 29-30 207 Contents Gepard でドットプロット 連載第 8 回 W5-3 で最も長い sequence 同士のドットプロットを実行できなかったが
NGSデータ解析入門Webセミナー
 NGS データ解析入門 Web セミナー : RNA-Seq 解析編 1 RNA-Seq データ解析の手順 遺伝子発現量測定 シークエンス マッピング サンプル間比較 機能解析など 2 CLC Genomics Workbench 使用ツール シークエンスデータ メタデータのインポート NGS data import Import Metadata クオリティチェック Create Sequencing
NGS データ解析入門 Web セミナー : RNA-Seq 解析編 1 RNA-Seq データ解析の手順 遺伝子発現量測定 シークエンス マッピング サンプル間比較 機能解析など 2 CLC Genomics Workbench 使用ツール シークエンスデータ メタデータのインポート NGS data import Import Metadata クオリティチェック Create Sequencing
1. はじめに 1. はじめに 1-1. KaPPA-Average とは KaPPA-Average は KaPPA-View( でマイクロアレイデータを解析する際に便利なデータ変換ソフトウェアです 一般のマイクロアレイでは 一つのプロー
 KaPPA-Average 1.0 マニュアル 第 1.0 版 制作者 : かずさ DNA 研究所櫻井望 制作日 : 2010 年 1 月 12 日 目次 1. はじめに 2 1-1. KaPPA-Average とは 2 1-2. 動作環境 3 1-3. インストールと起動 3 2. 操作説明 4 2-1. メイン機能 - Calc. Average 4 2-1-1. データの準備 4 2-1-2.
KaPPA-Average 1.0 マニュアル 第 1.0 版 制作者 : かずさ DNA 研究所櫻井望 制作日 : 2010 年 1 月 12 日 目次 1. はじめに 2 1-1. KaPPA-Average とは 2 1-2. 動作環境 3 1-3. インストールと起動 3 2. 操作説明 4 2-1. メイン機能 - Calc. Average 4 2-1-1. データの準備 4 2-1-2.
計算機生命科学の基礎II_
 Ⅱ 1.4 atsushi_doi@cell-innovator.com 812-8582 3-1-1 8 806 http://www.cell-innovator.com BioGPS Connectivity Map The Cancer Genome Atlas (TCGA); cbioportal GO DAVID, GSEA WCGNA BioGPS http://biogps.org/
Ⅱ 1.4 atsushi_doi@cell-innovator.com 812-8582 3-1-1 8 806 http://www.cell-innovator.com BioGPS Connectivity Map The Cancer Genome Atlas (TCGA); cbioportal GO DAVID, GSEA WCGNA BioGPS http://biogps.org/
機能ゲノム学(第6回)
 トランスクリプトーム解析の今昔 なぜマイクロアレイ? なぜRNA-Seq? 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 Contents トランスクリプトーム解析の概要 各手法の長所 短所 マイクロアレイ
トランスクリプトーム解析の今昔 なぜマイクロアレイ? なぜRNA-Seq? 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 Contents トランスクリプトーム解析の概要 各手法の長所 短所 マイクロアレイ
Microsoft Word - 1 color Normalization Document _Agilent version_ .doc
 color 実験の Normalization color 実験で得られた複数のアレイデータを相互比較するためには Normalization( 正規化 ) が必要です 2 つのサンプルを異なる色素でラベル化し 競合ハイブリダイゼーションさせる 2color 実験では 基本的に Dye Normalization( 色素補正 ) が適用されますが color 実験では データの特徴と実験の目的 (
color 実験の Normalization color 実験で得られた複数のアレイデータを相互比較するためには Normalization( 正規化 ) が必要です 2 つのサンプルを異なる色素でラベル化し 競合ハイブリダイゼーションさせる 2color 実験では 基本的に Dye Normalization( 色素補正 ) が適用されますが color 実験では データの特徴と実験の目的 (
PrimerArray® Analysis Tool Ver.2.2
 研究用 PrimerArray Analysis Tool Ver.2.2 説明書 v201801 PrimerArray Analysis Tool Ver.2.2 は PrimerArray( 製品コード PH001 ~ PH007 PH009 ~ PH015 PN001 ~ PN015) で得られたデータを解析するためのツールで コントロールサンプルと 1 種類の未知サンプル間の比較が可能です
研究用 PrimerArray Analysis Tool Ver.2.2 説明書 v201801 PrimerArray Analysis Tool Ver.2.2 は PrimerArray( 製品コード PH001 ~ PH007 PH009 ~ PH015 PN001 ~ PN015) で得られたデータを解析するためのツールで コントロールサンプルと 1 種類の未知サンプル間の比較が可能です
Rインストール手順
 R 本体は最新のリリース版 R パッケージは 必要最小限プラスアルファ の推奨インストール手順を示します スライドは Windows0 環境でのスクリーンショットです ウェブブラウザによって挙動が多少異なるのでご注意ください 私は Chrome を使っています R 本体とパッケージのインストール Windows 版 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二
R 本体は最新のリリース版 R パッケージは 必要最小限プラスアルファ の推奨インストール手順を示します スライドは Windows0 環境でのスクリーンショットです ウェブブラウザによって挙動が多少異なるのでご注意ください 私は Chrome を使っています R 本体とパッケージのインストール Windows 版 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二
Microsoft Word - apstattext04.docx
 4 章母集団と指定値との量的データの検定 4.1 検定手順今までは質的データの検定の方法を学んで来ましたが これからは量的データについてよく利用される方法を説明します 量的データでは データの分布が正規分布か否かで検定の方法が著しく異なります この章ではまずデータの分布の正規性を調べる方法を述べ 次にデータの平均値または中央値がある指定された値と違うかどうかの検定方法を説明します 以下の図 4.1.1
4 章母集団と指定値との量的データの検定 4.1 検定手順今までは質的データの検定の方法を学んで来ましたが これからは量的データについてよく利用される方法を説明します 量的データでは データの分布が正規分布か否かで検定の方法が著しく異なります この章ではまずデータの分布の正規性を調べる方法を述べ 次にデータの平均値または中央値がある指定された値と違うかどうかの検定方法を説明します 以下の図 4.1.1
機能ゲノム学(第6回)
 RNA-Seqデータ解析における正規化法の選択 :RPKM 値でサンプル間比較は危険?! 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 よりよい正規化法とは? その正規化法によって得られたデータを用いて発現変動の度合いでランキングしたときに
RNA-Seqデータ解析における正規化法の選択 :RPKM 値でサンプル間比較は危険?! 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 よりよい正規化法とは? その正規化法によって得られたデータを用いて発現変動の度合いでランキングしたときに
ゲノム情報解析基礎 ~ Rで塩基配列解析 ~
 ネット接続できないヒトも ダブルクリックでローカルに r_seq.html を起動可能です 実習は デスクトップ上にある hoge フォルダの中身が以下の状態を想定して行います (R で ) 塩基配列解析の利用法 : GC 含量計算から発現変動解析まで東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp
ネット接続できないヒトも ダブルクリックでローカルに r_seq.html を起動可能です 実習は デスクトップ上にある hoge フォルダの中身が以下の状態を想定して行います (R で ) 塩基配列解析の利用法 : GC 含量計算から発現変動解析まで東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp
データ科学2.pptx
 データ科学 多重検定 2 mul%ple test False Discovery Rate 藤博幸 前回の復習 1 多くの検定を繰り返す時には 単純に個々の検定を繰り返すだけでは不十分 5% 有意水準ということは, 1000 回検定を繰り返すと, 50 回くらいは帰無仮説が正しいのに 間違って棄却されてすまうじちがあるということ ex) 1 万個の遺伝子について 正常細胞とガン細胞で それぞれの遺伝子の発現に差があるかどうかを検定
データ科学 多重検定 2 mul%ple test False Discovery Rate 藤博幸 前回の復習 1 多くの検定を繰り返す時には 単純に個々の検定を繰り返すだけでは不十分 5% 有意水準ということは, 1000 回検定を繰り返すと, 50 回くらいは帰無仮説が正しいのに 間違って棄却されてすまうじちがあるということ ex) 1 万個の遺伝子について 正常細胞とガン細胞で それぞれの遺伝子の発現に差があるかどうかを検定
ChIP-seq
 ChIP-seq 1 ChIP-seq 解析原理 ChIP サンプルのフラグメントでは タンパク質結合部位付近にそれぞれ Forward と Reverse のリードがマップされることが予想される ChIP のサンプルでは Forward と Reverse のリードを 3 側へシフトさせ ChIP のピークを算出する コントロールサンプルでは ChIP のサンプルとは異なり 特定の場所に多くマップされないため
ChIP-seq 1 ChIP-seq 解析原理 ChIP サンプルのフラグメントでは タンパク質結合部位付近にそれぞれ Forward と Reverse のリードがマップされることが予想される ChIP のサンプルでは Forward と Reverse のリードを 3 側へシフトさせ ChIP のピークを算出する コントロールサンプルでは ChIP のサンプルとは異なり 特定の場所に多くマップされないため
基本的な利用法
 到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握 ) 4. ありがちなミス のところで示しているエラーメッセージとその原因をきっちり理解
到達目標 : このスライドに書かれている程度のことは自在にできるようにしてエラーへの対処法を身につける. 必要なパッケージのインストールが正しくできているかどうかの自力での判定 および個別のパッケージのインストール. 作業ディレクトリの変更 3. テキストエディタで自在に入出力ファイル名の変更 ( どんなファイル名のものがどこに生成されるかという全体像の把握 ) 4. ありがちなミス のところで示しているエラーメッセージとその原因をきっちり理解
Medical3
 1.4.1 クロス集計表の作成 -l m 分割表 - 3つ以上のカテゴリを含む変数を用いて l mのクロス集計表による分析を行います この例では race( 人種 ) によってlow( 低体重出生 ) に差が認められるかどうかを分析します 人種には3つのカテゴリ 低体重出生には2つのカテゴリが含まれています 2つの変数はともにカテゴリ変数であるため クロス集計表によって分析します 1. 分析メニュー
1.4.1 クロス集計表の作成 -l m 分割表 - 3つ以上のカテゴリを含む変数を用いて l mのクロス集計表による分析を行います この例では race( 人種 ) によってlow( 低体重出生 ) に差が認められるかどうかを分析します 人種には3つのカテゴリ 低体重出生には2つのカテゴリが含まれています 2つの変数はともにカテゴリ変数であるため クロス集計表によって分析します 1. 分析メニュー
Microsoft PowerPoint - R-intro-04.ppt
 R で学ぶデータ解析とシミュレーション 4 ~ データハンドリング入門 ~ 4 時間目のメニュー パッケージについて パッケージとは パッケージの呼び出し 追加パッケージのインストール データハンドリング入門 データフレームとは 種々のテキストファイルを R に読み込ませる方法 データハンドリング手法一覧 演習 2 パッケージとは R は関数とデータを機能別に分類して パッケージ という形にまとめている
R で学ぶデータ解析とシミュレーション 4 ~ データハンドリング入門 ~ 4 時間目のメニュー パッケージについて パッケージとは パッケージの呼び出し 追加パッケージのインストール データハンドリング入門 データフレームとは 種々のテキストファイルを R に読み込ませる方法 データハンドリング手法一覧 演習 2 パッケージとは R は関数とデータを機能別に分類して パッケージ という形にまとめている
統計的データ解析
 統計的データ解析 011 011.11.9 林田清 ( 大阪大学大学院理学研究科 ) 連続確率分布の平均値 分散 比較のため P(c ) c 分布 自由度 の ( カイ c 平均値 0, 標準偏差 1の正規分布 に従う変数 xの自乗和 c x =1 が従う分布を自由度 の分布と呼ぶ 一般に自由度の分布は f /1 c / / ( c ) {( c ) e }/ ( / ) 期待値 二乗 ) 分布 c
統計的データ解析 011 011.11.9 林田清 ( 大阪大学大学院理学研究科 ) 連続確率分布の平均値 分散 比較のため P(c ) c 分布 自由度 の ( カイ c 平均値 0, 標準偏差 1の正規分布 に従う変数 xの自乗和 c x =1 が従う分布を自由度 の分布と呼ぶ 一般に自由度の分布は f /1 c / / ( c ) {( c ) e }/ ( / ) 期待値 二乗 ) 分布 c
Qlucore_seminar_slide_180604
 シングルセル RNA-Seq のための 情報解析 フィルジェン株式会社バイオサイエンス部 (biosupport@filgen.jp) 1 シングルセル RNA-Seq シングルセル RNA-Seq のデータ解析では 通常の RNA-Seq データの解析手法に加え データセット内の各細胞の遺伝子発現プロファイルの違いを俯瞰できるような 強力な情報解析アルゴリズムと データのビジュアライズ機能を利用する必要がある
シングルセル RNA-Seq のための 情報解析 フィルジェン株式会社バイオサイエンス部 (biosupport@filgen.jp) 1 シングルセル RNA-Seq シングルセル RNA-Seq のデータ解析では 通常の RNA-Seq データの解析手法に加え データセット内の各細胞の遺伝子発現プロファイルの違いを俯瞰できるような 強力な情報解析アルゴリズムと データのビジュアライズ機能を利用する必要がある
スライド 1
 ラベル屋さん HOME かんたんマニュアル リンクコース 目次 STEP 1-2 : ( 基礎編 ) 用紙の選択と文字の入力 STEP 3 : ( 基礎編 ) リンクの設定 STEP 4 : ( 基礎編 ) リンクデータの入力と印刷 STEP 5 : ( 応用編 ) リンクデータの入力 1 STEP 6 : ( 応用編 ) リンクデータの入力 2 STEP 7-8 : ( 応用編 ) リンク機能で使ったデータをコピーしたい場合
ラベル屋さん HOME かんたんマニュアル リンクコース 目次 STEP 1-2 : ( 基礎編 ) 用紙の選択と文字の入力 STEP 3 : ( 基礎編 ) リンクの設定 STEP 4 : ( 基礎編 ) リンクデータの入力と印刷 STEP 5 : ( 応用編 ) リンクデータの入力 1 STEP 6 : ( 応用編 ) リンクデータの入力 2 STEP 7-8 : ( 応用編 ) リンク機能で使ったデータをコピーしたい場合
JMP による 2 群間の比較 SAS Institute Japan 株式会社 JMP ジャパン事業部 2008 年 3 月 JMP で t 検定や Wilcoxon 検定はどのメニューで実行できるのか または検定を行う際の前提条件の評価 ( 正規性 等分散性 ) はどのメニューで実行できるのかと
 JMP による 2 群間の比較 SAS Institute Japan 株式会社 JMP ジャパン事業部 2008 年 3 月 JMP で t 検定や Wilcoxon 検定はどのメニューで実行できるのか または検定を行う際の前提条件の評価 ( 正規性 等分散性 ) はどのメニューで実行できるのかというお問い合わせがよくあります そこで本文書では これらについて の回答を 例題を用いて説明します 1.
JMP による 2 群間の比較 SAS Institute Japan 株式会社 JMP ジャパン事業部 2008 年 3 月 JMP で t 検定や Wilcoxon 検定はどのメニューで実行できるのか または検定を行う際の前提条件の評価 ( 正規性 等分散性 ) はどのメニューで実行できるのかというお問い合わせがよくあります そこで本文書では これらについて の回答を 例題を用いて説明します 1.
Microsoft Word - index.html
 R のインストールと超入門 R とは? R ダウンロード R のインストール R の基本操作 R 終了データの読み込みパッケージの操作 R とは? Rはデータ解析 マイニングを行うフリーソフトである Rはデータ解析の環境でもあり 言語でもある ニュージーランドのオークランド (Auckland) 大学の統計学科のRobert Gentlemanと Ross Ihakaにより開発がはじめられ 1997
R のインストールと超入門 R とは? R ダウンロード R のインストール R の基本操作 R 終了データの読み込みパッケージの操作 R とは? Rはデータ解析 マイニングを行うフリーソフトである Rはデータ解析の環境でもあり 言語でもある ニュージーランドのオークランド (Auckland) 大学の統計学科のRobert Gentlemanと Ross Ihakaにより開発がはじめられ 1997
Chapter カスタムテーブルの概要 カスタムテーブル Custom Tables は 複数の変数に基づいた多重クロス集計テーブルや スケール変数を用いた集計テーブルなど より複雑な集計表を自由に設計することができるIBM SPSS Statisticsのオプション製品です テーブ
 カスタムテーブル入門 1 カスタムテーブル入門 カスタムテーブル Custom Tables は IBM SPSS Statisticsのオプション機能の1つです カスタムテーブルを追加することで 基本的な度数集計テーブルやクロス集計テーブルの作成はもちろん 複数の変数を積み重ねた多重クロス集計テーブルや スケール変数を用いた集計テーブルなど より複雑で柔軟な集計表を作成することができます この章では
カスタムテーブル入門 1 カスタムテーブル入門 カスタムテーブル Custom Tables は IBM SPSS Statisticsのオプション機能の1つです カスタムテーブルを追加することで 基本的な度数集計テーブルやクロス集計テーブルの作成はもちろん 複数の変数を積み重ねた多重クロス集計テーブルや スケール変数を用いた集計テーブルなど より複雑で柔軟な集計表を作成することができます この章では
初めてのプログラミング
 Excel の使い方 2 ~ 数式の入力 グラフの作成 ~ 0. データ処理とグラフの作成 前回は エクセルを用いた表の作成方法について学びました 今回は エクセルを用いたデータ処理方法と グラフの作成方法について学ぶことにしましょう 1. 数式の入力 1 ここでは x, y の値を入力していきます まず 前回の講義を参考に 自動補間機能を用いて x の値を入力してみましょう 補間方法としては A2,
Excel の使い方 2 ~ 数式の入力 グラフの作成 ~ 0. データ処理とグラフの作成 前回は エクセルを用いた表の作成方法について学びました 今回は エクセルを用いたデータ処理方法と グラフの作成方法について学ぶことにしましょう 1. 数式の入力 1 ここでは x, y の値を入力していきます まず 前回の講義を参考に 自動補間機能を用いて x の値を入力してみましょう 補間方法としては A2,
第21章 表計算
 第 3 部 第 3 章 Web サイトの作成 3.3.1 WEB ページ作成ソフト Dreamweaver の基本操作 Web ページは HTML CSS という言語で作成されており これらは一般的なテキストエディタで作成できるのが特徴ですが その入 力 編集は時に煩雑なものです そこで それらの入力 編集作業など Web ページの作成を補助するソフトウェアである Dreamweaver の使い方について解説していきます
第 3 部 第 3 章 Web サイトの作成 3.3.1 WEB ページ作成ソフト Dreamweaver の基本操作 Web ページは HTML CSS という言語で作成されており これらは一般的なテキストエディタで作成できるのが特徴ですが その入 力 編集は時に煩雑なものです そこで それらの入力 編集作業など Web ページの作成を補助するソフトウェアである Dreamweaver の使い方について解説していきます
ビジネス統計 統計基礎とエクセル分析 正誤表
 ビジネス統計統計基礎とエクセル分析 ビジネス統計スペシャリスト エクセル分析スペシャリスト 公式テキスト正誤表と学習用データ更新履歴 平成 30 年 5 月 14 日現在 公式テキスト正誤表 頁場所誤正修正 6 知識編第 章 -3-3 最頻値の解説内容 たとえば, 表.1 のデータであれば, 最頻値は 167.5cm というたとえば, 表.1 のデータであれば, 最頻値は 165.0cm ということになります
ビジネス統計統計基礎とエクセル分析 ビジネス統計スペシャリスト エクセル分析スペシャリスト 公式テキスト正誤表と学習用データ更新履歴 平成 30 年 5 月 14 日現在 公式テキスト正誤表 頁場所誤正修正 6 知識編第 章 -3-3 最頻値の解説内容 たとえば, 表.1 のデータであれば, 最頻値は 167.5cm というたとえば, 表.1 のデータであれば, 最頻値は 165.0cm ということになります
Microsoft PowerPoint ppt
 情報科学第 07 回データ解析と統計代表値 平均 分散 度数分布表 1 本日の内容 データ解析とは 統計の基礎的な値 平均と分散 度数分布表とヒストグラム 講義のページ 第 7 回のその他の欄に 本日使用する教材があります 171025.xls というファイルがありますので ダウンロードして デスクトップに保存してください 2/45 はじめに データ解析とは この世の中には多くのデータが溢れています
情報科学第 07 回データ解析と統計代表値 平均 分散 度数分布表 1 本日の内容 データ解析とは 統計の基礎的な値 平均と分散 度数分布表とヒストグラム 講義のページ 第 7 回のその他の欄に 本日使用する教材があります 171025.xls というファイルがありますので ダウンロードして デスクトップに保存してください 2/45 はじめに データ解析とは この世の中には多くのデータが溢れています
配付資料 自習用テキスト 解析サンプル配布ページ 2
 分子系統樹推定法 理論と応用 2009年11月6日 筑波大 院 生命環境 田辺晶史 配付資料 自習用テキスト 解析サンプル配布ページ http://www.fifthdimension.jp/documents/molphytextbook/ 2 参考書籍 分子系統学 3 参考書籍 統計的モデル選択とベイジアンMCMC 4 祖先的な形質 問題 OTU左の の色は表現型形質の状態を表している 赤と青
分子系統樹推定法 理論と応用 2009年11月6日 筑波大 院 生命環境 田辺晶史 配付資料 自習用テキスト 解析サンプル配布ページ http://www.fifthdimension.jp/documents/molphytextbook/ 2 参考書籍 分子系統学 3 参考書籍 統計的モデル選択とベイジアンMCMC 4 祖先的な形質 問題 OTU左の の色は表現型形質の状態を表している 赤と青
数量的アプローチ 年 6 月 11 日 イントロダクション データ分析をマスターする 12 のレッスン ウェブサポートページ ( 有斐閣 ) 水落研究室 R http:
 イントロダクション データ分析をマスターする 12 のレッスン ウェブサポートページ ( 有斐閣 ) http://yuhikaku-nibu.txt-nifty.com/blog/2017/09/22103.html 水落研究室 R http://depts.nanzan-u.ac.jp/ugrad/ps/mizuochi/r.html 1 この授業では統計ソフト R を使って分析を行います データを扱うソフトとして
イントロダクション データ分析をマスターする 12 のレッスン ウェブサポートページ ( 有斐閣 ) http://yuhikaku-nibu.txt-nifty.com/blog/2017/09/22103.html 水落研究室 R http://depts.nanzan-u.ac.jp/ugrad/ps/mizuochi/r.html 1 この授業では統計ソフト R を使って分析を行います データを扱うソフトとして
ANOVA
 3 つ z のグループの平均を比べる ( 分散分析 : ANOVA: analysis of variance) 分散分析は 全体として 3 つ以上のグループの平均に差があるか ということしかわからないために, どのグループの間に差があったかを確かめるには 多重比較 という方法を用います これは Excel だと自分で計算しなければならないので, 分散分析には統計ソフトを使った方がよいでしょう 1.
3 つ z のグループの平均を比べる ( 分散分析 : ANOVA: analysis of variance) 分散分析は 全体として 3 つ以上のグループの平均に差があるか ということしかわからないために, どのグループの間に差があったかを確かめるには 多重比較 という方法を用います これは Excel だと自分で計算しなければならないので, 分散分析には統計ソフトを使った方がよいでしょう 1.
> usdata01 と打ち込んでエンター キーを押すと V1 V2 V : : : : のように表示され 読み込まれていることがわかる ここで V1, V2, V3 は R が列のデータに自 動的につけた変数名である ( variable
 R による回帰分析 ( 最小二乗法 ) この資料では 1. データを読み込む 2. 最小二乗法によってパラメーターを推定する 3. データをプロットし 回帰直線を書き込む 4. いろいろなデータの読み込み方について簡単に説明する 1. データを読み込む 以下では read.table( ) 関数を使ってテキストファイル ( 拡張子が.txt のファイル ) のデー タの読み込み方を説明する 1.1
R による回帰分析 ( 最小二乗法 ) この資料では 1. データを読み込む 2. 最小二乗法によってパラメーターを推定する 3. データをプロットし 回帰直線を書き込む 4. いろいろなデータの読み込み方について簡単に説明する 1. データを読み込む 以下では read.table( ) 関数を使ってテキストファイル ( 拡張子が.txt のファイル ) のデー タの読み込み方を説明する 1.1
Excelによる統計分析検定_知識編_小塚明_5_9章.indd
 第7章57766 検定と推定 サンプリングによって得られた標本から, 母集団の統計的性質に対して推測を行うことを統計的推測といいます 本章では, 推測統計の根幹をなす仮説検定と推定の基本的な考え方について説明します 前章までの知識を用いて, 具体的な分析を行います 本章以降の知識は操作編での操作に直接関連していますので, 少し聞きなれない言葉ですが, 帰無仮説 有意水準 棄却域 などの意味を理解して,
第7章57766 検定と推定 サンプリングによって得られた標本から, 母集団の統計的性質に対して推測を行うことを統計的推測といいます 本章では, 推測統計の根幹をなす仮説検定と推定の基本的な考え方について説明します 前章までの知識を用いて, 具体的な分析を行います 本章以降の知識は操作編での操作に直接関連していますので, 少し聞きなれない言葉ですが, 帰無仮説 有意水準 棄却域 などの意味を理解して,
IonTorrent RNA-Seq 解析概要 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science
 IonTorrent RNA-Seq 解析概要 2017-03 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science 資料概要 この資料は IonTorrent シーケンサーで RNA-Seq (WholeTranscriptome mrna ampliseqrna mirna) 解析を実施されるユーザー様向けの内容となっています
IonTorrent RNA-Seq 解析概要 2017-03 サーモフィッシャーサイエンティフィックライフテクノロジーズジャパンテクニカルサポート The world leader in serving science 資料概要 この資料は IonTorrent シーケンサーで RNA-Seq (WholeTranscriptome mrna ampliseqrna mirna) 解析を実施されるユーザー様向けの内容となっています
Microsoft Word - Stattext12.doc
 章対応のない 群間の量的データの検定. 検定手順 この章ではデータ間に 対 の対応のないつの標本から推定される母集団間の平均値や中央値の比較を行ないます 検定手法は 図. のようにまず正規に従うかどうかを調べます 但し この場合はつの群が共に正規に従うことを調べる必要があります 次に 群とも正規ならば F 検定を用いて等分散であるかどうかを調べます 等分散の場合は t 検定 等分散でない場合はウェルチ
章対応のない 群間の量的データの検定. 検定手順 この章ではデータ間に 対 の対応のないつの標本から推定される母集団間の平均値や中央値の比較を行ないます 検定手法は 図. のようにまず正規に従うかどうかを調べます 但し この場合はつの群が共に正規に従うことを調べる必要があります 次に 群とも正規ならば F 検定を用いて等分散であるかどうかを調べます 等分散の場合は t 検定 等分散でない場合はウェルチ
Microsoft PowerPoint - R-stat-intro_12.ppt [互換モード]
![Microsoft PowerPoint - R-stat-intro_12.ppt [互換モード] Microsoft PowerPoint - R-stat-intro_12.ppt [互換モード]](/thumbs/101/151921093.jpg) R で統計解析入門 (12) 生存時間解析 中篇 準備 : データ DEP の読み込み 1. データ DEP を以下からダウンロードする http://www.cwk.zaq.ne.jp/fkhud708/files/dep.csv /fkh /d 2. ダウンロードした場所を把握する ここでは c:/temp とする 3. R を起動し,2. 2 の場所に移動し, データを読み込む 4. データ
R で統計解析入門 (12) 生存時間解析 中篇 準備 : データ DEP の読み込み 1. データ DEP を以下からダウンロードする http://www.cwk.zaq.ne.jp/fkhud708/files/dep.csv /fkh /d 2. ダウンロードした場所を把握する ここでは c:/temp とする 3. R を起動し,2. 2 の場所に移動し, データを読み込む 4. データ
<4D F736F F D2091E C C6791D B28DB88DEC90AC D836A B2E646F63>
 改訂第 2 版携帯電話 PC 用調査作成システム Ver. 2 利用マニュアル 多くの先生のご協力のおかげで, 昨年の Ver. 1 の問題点が明らかとなり, 今年はほぼすべてを改良しました Ver. 2 での改良点は以下の通りです 1. 携帯電話用と PC 用が同時に作成されるようにしました 2. 携帯電話の調査の URL を, 文字だけでなく QR コードでも表示できるようにしました 3. 回答法として,2
改訂第 2 版携帯電話 PC 用調査作成システム Ver. 2 利用マニュアル 多くの先生のご協力のおかげで, 昨年の Ver. 1 の問題点が明らかとなり, 今年はほぼすべてを改良しました Ver. 2 での改良点は以下の通りです 1. 携帯電話用と PC 用が同時に作成されるようにしました 2. 携帯電話の調査の URL を, 文字だけでなく QR コードでも表示できるようにしました 3. 回答法として,2
PowerPoint プレゼンテーション
 FLEXSCHE Excel 帳票 入門ガイド 1 目次 2 EXCEL 帳票とは EDIF を用いて出力された一時データを元に それを EXCEL 形式の帳票として出力する機能です 利用するには FLEXSCHE EDIF の他 Microsoft Excel 2003 以降が必要です レイアウトデザインも EXCEL で行うので 多くの方に操作に抵抗なく編集していただけます この入門ガイドでは
FLEXSCHE Excel 帳票 入門ガイド 1 目次 2 EXCEL 帳票とは EDIF を用いて出力された一時データを元に それを EXCEL 形式の帳票として出力する機能です 利用するには FLEXSCHE EDIF の他 Microsoft Excel 2003 以降が必要です レイアウトデザインも EXCEL で行うので 多くの方に操作に抵抗なく編集していただけます この入門ガイドでは
EBNと疫学
 推定と検定 57 ( 復習 ) 記述統計と推測統計 統計解析は大きく 2 つに分けられる 記述統計 推測統計 記述統計 観察集団の特性を示すもの 代表値 ( 平均値や中央値 ) や ばらつきの指標 ( 標準偏差など ) 図表を効果的に使う 推測統計 観察集団のデータから母集団の特性を 推定 する 平均 / 分散 / 係数値などの推定 ( 点推定 ) 点推定値のばらつきを調べる ( 区間推定 ) 検定統計量を用いた検定
推定と検定 57 ( 復習 ) 記述統計と推測統計 統計解析は大きく 2 つに分けられる 記述統計 推測統計 記述統計 観察集団の特性を示すもの 代表値 ( 平均値や中央値 ) や ばらつきの指標 ( 標準偏差など ) 図表を効果的に使う 推測統計 観察集団のデータから母集団の特性を 推定 する 平均 / 分散 / 係数値などの推定 ( 点推定 ) 点推定値のばらつきを調べる ( 区間推定 ) 検定統計量を用いた検定
Outlook2010 の メール 連絡先 に関連する内容を解説します 注意 :Outlook2007 と Outlook2010 では 基本操作 基本画面が違うため この資料では Outlook2010 のみで参考にしてください Outlook2010 の画面構成について... 2 メールについて
 Outlook2010 - メール 連絡先など - Outlook2010 の メール 連絡先 に関連する内容を解説します 注意 :Outlook2007 と Outlook2010 では 基本操作 基本画面が違うため この資料では Outlook2010 のみで参考にしてください Outlook2010 の画面構成について... 2 メールについて... 3 画面構成と操作... 3 人物情報ウィンドウ...
Outlook2010 - メール 連絡先など - Outlook2010 の メール 連絡先 に関連する内容を解説します 注意 :Outlook2007 と Outlook2010 では 基本操作 基本画面が違うため この資料では Outlook2010 のみで参考にしてください Outlook2010 の画面構成について... 2 メールについて... 3 画面構成と操作... 3 人物情報ウィンドウ...
年齢別人数計算ツールマニュアル
 年齢別人数計算ツールの使用手引 本ツールは 学校基本調査の調査項目である 年齢別入学者数 を 学生名簿等の既存データを利用して集計するものです < 対象となる調査票 > 1 学校調査票 ( 大学 ) 学部学生内訳票 ( 様式第 8 号 ) 2 学校調査票 ( 短期大学 ) 本科学生内訳票 ( 様式第 10 号 ) < 年齢別人数計算ツールの使用の流れは以下のとおりです > 巻末に補足事項の記載がございます
年齢別人数計算ツールの使用手引 本ツールは 学校基本調査の調査項目である 年齢別入学者数 を 学生名簿等の既存データを利用して集計するものです < 対象となる調査票 > 1 学校調査票 ( 大学 ) 学部学生内訳票 ( 様式第 8 号 ) 2 学校調査票 ( 短期大学 ) 本科学生内訳票 ( 様式第 10 号 ) < 年齢別人数計算ツールの使用の流れは以下のとおりです > 巻末に補足事項の記載がございます
win版8日目
 8 日目 : 項目のチェック (2) 1 日 30 分くらい,30 日で何とか R をそこそこ使えるようになるための練習帳 :Win 版 昨日は, 平均値などの基礎統計量を計算する試行錯誤へご招待しましたが (?), 今日は簡 単にやってみます そのためには,psych というパッケージが必要となりますが, パッケー ジのインストール & 読み込みの詳しい方法は, 後で説明します 以下の説明は,psych
8 日目 : 項目のチェック (2) 1 日 30 分くらい,30 日で何とか R をそこそこ使えるようになるための練習帳 :Win 版 昨日は, 平均値などの基礎統計量を計算する試行錯誤へご招待しましたが (?), 今日は簡 単にやってみます そのためには,psych というパッケージが必要となりますが, パッケー ジのインストール & 読み込みの詳しい方法は, 後で説明します 以下の説明は,psych
Microsoft PowerPoint - prog03.ppt
 プログラミング言語 3 第 03 回 (2007 年 10 月 08 日 ) 1 今日の配布物 片面の用紙 1 枚 今日の課題が書かれています 本日の出欠を兼ねています 2/33 今日やること http://www.tnlab.ice.uec.ac.jp/~s-okubo/class/java06/ にアクセスすると 教材があります 2007 年 10 月 08 日分と書いてある部分が 本日の教材です
プログラミング言語 3 第 03 回 (2007 年 10 月 08 日 ) 1 今日の配布物 片面の用紙 1 枚 今日の課題が書かれています 本日の出欠を兼ねています 2/33 今日やること http://www.tnlab.ice.uec.ac.jp/~s-okubo/class/java06/ にアクセスすると 教材があります 2007 年 10 月 08 日分と書いてある部分が 本日の教材です
図 1 アドインに登録する メニューバーに [BAYONET] が追加されます 登録 : Excel 2007, 2010, 2013 の場合 1 Excel ブックを開きます Excel2007 の場合 左上の Office マークをクリックします 図 2 Office マーク (Excel 20
![図 1 アドインに登録する メニューバーに [BAYONET] が追加されます 登録 : Excel 2007, 2010, 2013 の場合 1 Excel ブックを開きます Excel2007 の場合 左上の Office マークをクリックします 図 2 Office マーク (Excel 20 図 1 アドインに登録する メニューバーに [BAYONET] が追加されます 登録 : Excel 2007, 2010, 2013 の場合 1 Excel ブックを開きます Excel2007 の場合 左上の Office マークをクリックします 図 2 Office マーク (Excel 20](/thumbs/92/109963601.jpg) BayoLink Excel アドイン使用方法 1. はじめに BayoLink Excel アドインは MS Office Excel のアドインツールです BayoLink Excel アドインは Excel から API を利用して BayoLink と通信し モデルのインポートや推論の実行を行います BayoLink 本体ではできない 複数のデータを一度に推論することができます なお現状ではソフトエビデンスを指定して推論を行うことはできません
BayoLink Excel アドイン使用方法 1. はじめに BayoLink Excel アドインは MS Office Excel のアドインツールです BayoLink Excel アドインは Excel から API を利用して BayoLink と通信し モデルのインポートや推論の実行を行います BayoLink 本体ではできない 複数のデータを一度に推論することができます なお現状ではソフトエビデンスを指定して推論を行うことはできません
スクールCOBOL2002
 3. 関連資料 - よく使われる機能の操作方法 - (a) ファイルの入出力処理 - 順ファイル等を使ったプログラムの実行 - - 目次 -. はじめに 2. コーディング上の指定 3. 順ファイルの使用方法 4. プリンタへの出力方法 5. 索引ファイルの使用方法 6. 終わりに 2 . はじめに 本説明書では 簡単なプログラム ( ファイル等を使わないプログラム ) の作成からコンパイル 実行までの使用方法は既に理解しているものとして
3. 関連資料 - よく使われる機能の操作方法 - (a) ファイルの入出力処理 - 順ファイル等を使ったプログラムの実行 - - 目次 -. はじめに 2. コーディング上の指定 3. 順ファイルの使用方法 4. プリンタへの出力方法 5. 索引ファイルの使用方法 6. 終わりに 2 . はじめに 本説明書では 簡単なプログラム ( ファイル等を使わないプログラム ) の作成からコンパイル 実行までの使用方法は既に理解しているものとして
データ構造
 アルゴリズム及び実習 7 馬青 1 表探索 定義表探索とは 表の形で格納されているデータの中から条件に合ったデータを取り出してくる操作である 但し 表は配列 ( 連結 ) リストなどで実現できるので 以降 表 の代わりに直接 配列 や リスト などの表現を用いる場合が多い 表探索をただ 探索 と呼ぶ場合が多い 用語レコード : 表の中にある個々のデータをレコード (record) と呼ぶ フィールド
アルゴリズム及び実習 7 馬青 1 表探索 定義表探索とは 表の形で格納されているデータの中から条件に合ったデータを取り出してくる操作である 但し 表は配列 ( 連結 ) リストなどで実現できるので 以降 表 の代わりに直接 配列 や リスト などの表現を用いる場合が多い 表探索をただ 探索 と呼ぶ場合が多い 用語レコード : 表の中にある個々のデータをレコード (record) と呼ぶ フィールド
Microsoft PowerPoint - 統数研シンポジウム_R_ ppt
 でデータハンドリング ~ データフレーム 30 分クッキング ~ 本日のメニュー データフレームとは データフレームの作成 データハンドリングの方法と例 2 データフレームとは 統計解析を行うデータの形式は様々 ( R 上で ) データを手で入力して テキストファイル,EXCEL,ACCESS,SAS などの形式 R でデータ解析を行う際は, データフレームという形式にデータを変換することが多い (
でデータハンドリング ~ データフレーム 30 分クッキング ~ 本日のメニュー データフレームとは データフレームの作成 データハンドリングの方法と例 2 データフレームとは 統計解析を行うデータの形式は様々 ( R 上で ) データを手で入力して テキストファイル,EXCEL,ACCESS,SAS などの形式 R でデータ解析を行う際は, データフレームという形式にデータを変換することが多い (
2011 年度春学期基礎ゼミナール ( コンピューティングクラス ) A コース 1 / 18 コンピュータリテラシー A コース 第 10 講 [ 全 15 講 ] 2011 年度春学期 基礎ゼミナール ( コンピューティングクラス ) 同志社大学経済学部 DIGITAL TEXT コンピュータリ
![2011 年度春学期基礎ゼミナール ( コンピューティングクラス ) A コース 1 / 18 コンピュータリテラシー A コース 第 10 講 [ 全 15 講 ] 2011 年度春学期 基礎ゼミナール ( コンピューティングクラス ) 同志社大学経済学部 DIGITAL TEXT コンピュータリ 2011 年度春学期基礎ゼミナール ( コンピューティングクラス ) A コース 1 / 18 コンピュータリテラシー A コース 第 10 講 [ 全 15 講 ] 2011 年度春学期 基礎ゼミナール ( コンピューティングクラス ) 同志社大学経済学部 DIGITAL TEXT コンピュータリ](/thumbs/88/116922111.jpg) A コース 1 / 18 コンピュータリテラシー A コース 第 10 講 [ 全 15 講 ] 2011 年度春学期 基礎ゼミナール ( コンピューティングクラス ) 第 10 講データ処理 5 10-1 ブック ( ファイル ) を開く第 8 講で保存した meibo.xlsx を開きましょう 2 / 18 10-2 行列の非表示と再表示 E 列 と F 列 を非表示にしましょう 1. 列番号
A コース 1 / 18 コンピュータリテラシー A コース 第 10 講 [ 全 15 講 ] 2011 年度春学期 基礎ゼミナール ( コンピューティングクラス ) 第 10 講データ処理 5 10-1 ブック ( ファイル ) を開く第 8 講で保存した meibo.xlsx を開きましょう 2 / 18 10-2 行列の非表示と再表示 E 列 と F 列 を非表示にしましょう 1. 列番号
クエリエディターを活用した公開データのデータ整形
 クエリエディターを活用した 公開データのデータ整形 山形大学学術研究院 (ICT IR 担当 ) 鈴木達哉 suzukit@jm.kj.yamagata-u.ac.jp February 26, 2018 データマネジメント に関する勉強会 ( 於 : 東京工業大学キャンパスイノベーションセンター (CIC)509 号室 ) 本日の内容 1. 目標の確認 2. クエリエディターについての説明 起動方法
クエリエディターを活用した 公開データのデータ整形 山形大学学術研究院 (ICT IR 担当 ) 鈴木達哉 suzukit@jm.kj.yamagata-u.ac.jp February 26, 2018 データマネジメント に関する勉強会 ( 於 : 東京工業大学キャンパスイノベーションセンター (CIC)509 号室 ) 本日の内容 1. 目標の確認 2. クエリエディターについての説明 起動方法
Microsoft Word - 操作マニュアル-Excel-2.doc
 Excel プログラム開発の練習マニュアルー 1 ( 関数の学習 ) 作成 2015.01.31 修正 2015.02.04 本マニュアルでは Excel のプログラム開発を行なうに当たって まずは Excel の関数に関する学習 について記述する Ⅰ.Excel の関数に関する学習 1. 初めに Excel は単なる表計算のソフトと思っている方も多いと思います しかし Excel には 一般的に使用する
Excel プログラム開発の練習マニュアルー 1 ( 関数の学習 ) 作成 2015.01.31 修正 2015.02.04 本マニュアルでは Excel のプログラム開発を行なうに当たって まずは Excel の関数に関する学習 について記述する Ⅰ.Excel の関数に関する学習 1. 初めに Excel は単なる表計算のソフトと思っている方も多いと思います しかし Excel には 一般的に使用する
目 次 1. はじめに ソフトの起動と終了 環境設定 発助 SMS ファイルの操作 電話番号設定 運用条件 回線情報 SMS 送信の開始と停止 ファイル出力... 16
 発助 SMS 操作マニュアル Ver1.2 2018.7.21 ソフトプラン合同会社 1/18 目 次 1. はじめに... 3 2. ソフトの起動と終了... 3 3. 環境設定... 5 4. 発助 SMS ファイルの操作... 7 5. 電話番号設定... 9 6. 運用条件... 11 7. 回線情報... 12 8.SMS 送信の開始と停止... 13 9. ファイル出力... 16 10.
発助 SMS 操作マニュアル Ver1.2 2018.7.21 ソフトプラン合同会社 1/18 目 次 1. はじめに... 3 2. ソフトの起動と終了... 3 3. 環境設定... 5 4. 発助 SMS ファイルの操作... 7 5. 電話番号設定... 9 6. 運用条件... 11 7. 回線情報... 12 8.SMS 送信の開始と停止... 13 9. ファイル出力... 16 10.
PowerPoint プレゼンテーション
 かんたんマニュアル 差し込み印刷編 目次 STEP:1 STEP:2 STEP:3 STEP:4 元となるラベル カードのデータを作ろうP.2 差し込みデータの関連付けを設定しよう P.7 データの差し込みをしよう P.11 印刷しよう P.17 STEP1: 画面の確認をしよう 差し込み印刷とは 表計算ソフトで作った住所録を宛名ラベルに印刷したり 名簿をも とに同じ形式のカードを作ったりするときに便利な機能です
かんたんマニュアル 差し込み印刷編 目次 STEP:1 STEP:2 STEP:3 STEP:4 元となるラベル カードのデータを作ろうP.2 差し込みデータの関連付けを設定しよう P.7 データの差し込みをしよう P.11 印刷しよう P.17 STEP1: 画面の確認をしよう 差し込み印刷とは 表計算ソフトで作った住所録を宛名ラベルに印刷したり 名簿をも とに同じ形式のカードを作ったりするときに便利な機能です
コンテンツ作成基本編
 コンテンツ作成マニュアル基本編 もくじ コンテンツとは 公開する物件検索サイト内の情報の一つ一つを指します 3~8 サイト作成の流れ 物件検索一覧ページ 物件検索を行うためのページを作成するための一覧の流れです 9~4 その他コンテンツについて 各々のページを作成するための コンテンツ管理画面の項目です 5~7 コンテンツとは 3 コンテンツとは コンテンツとは 公開する Web サイトのページ つ
コンテンツ作成マニュアル基本編 もくじ コンテンツとは 公開する物件検索サイト内の情報の一つ一つを指します 3~8 サイト作成の流れ 物件検索一覧ページ 物件検索を行うためのページを作成するための一覧の流れです 9~4 その他コンテンツについて 各々のページを作成するための コンテンツ管理画面の項目です 5~7 コンテンツとは 3 コンテンツとは コンテンツとは 公開する Web サイトのページ つ
生物工学会誌 第95巻 第5号 バイオインフォマティクスを使い尽くす秘訣教えます!【第5回】
 生物工学会誌第 95 巻第 5 号 Cytoscape Cytoscape Cytoscape 1) 15 Cytoscape Web 2) Mac Windows Linux OS Cytoscape GML BioPAX PSI-MI KGML SBML Pathway Commons IntAct BioMart Igraph Bioconductor PDF PS SVG PNG JPEG
生物工学会誌第 95 巻第 5 号 Cytoscape Cytoscape Cytoscape 1) 15 Cytoscape Web 2) Mac Windows Linux OS Cytoscape GML BioPAX PSI-MI KGML SBML Pathway Commons IntAct BioMart Igraph Bioconductor PDF PS SVG PNG JPEG
Microsoft PowerPoint - e-stat(OLS).pptx
 経済統計学 ( 補足 ) 最小二乗法について 担当 : 小塚匡文 2015 年 11 月 19 日 ( 改訂版 ) 神戸大学経済学部 2015 年度後期開講授業 補足 : 最小二乗法 ( 単回帰分析 ) 1.( 単純 ) 回帰分析とは? 標本サイズTの2 変数 ( ここではXとY) のデータが存在 YをXで説明する回帰方程式を推定するための方法 Y: 被説明変数 ( または従属変数 ) X: 説明変数
経済統計学 ( 補足 ) 最小二乗法について 担当 : 小塚匡文 2015 年 11 月 19 日 ( 改訂版 ) 神戸大学経済学部 2015 年度後期開講授業 補足 : 最小二乗法 ( 単回帰分析 ) 1.( 単純 ) 回帰分析とは? 標本サイズTの2 変数 ( ここではXとY) のデータが存在 YをXで説明する回帰方程式を推定するための方法 Y: 被説明変数 ( または従属変数 ) X: 説明変数
機能ゲノム学(第6回)
 マイクロアレイデータ解 析結果の正しい?! 解釈 について 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 自己紹介 2002 年 3 月 東京大学 大学院農学生命科学研究科博士課程修了 学位論文 : cdna
マイクロアレイデータ解 析結果の正しい?! 解釈 について 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) http://www.iu.a.u-tokyo.ac.jp/~kadota/ kadota@iu.a.u-tokyo.ac.jp 1 自己紹介 2002 年 3 月 東京大学 大学院農学生命科学研究科博士課程修了 学位論文 : cdna
分析のステップ Step 1: Y( 目的変数 ) に対する値の順序を確認 Step 2: モデルのあてはめ を実行 適切なモデルの指定 Step 3: オプションを指定し オッズ比とその信頼区間を表示 以下 このステップに沿って JMP の操作をご説明します Step 1: Y( 目的変数 ) の
 JMP によるオッズ比 リスク比 ( ハザード比 ) の算出と注意点 SAS Institute Japan 株式会社 JMP ジャパン事業部 2011 年 10 月改定 1. はじめに 本文書は JMP でロジスティック回帰モデルによるオッズ比 比例ハザードモデルによるリスク比 それぞれに対する信頼区間を求める操作方法と注意点を述べたものです 本文書は JMP 7 以降のバージョンに対応しております
JMP によるオッズ比 リスク比 ( ハザード比 ) の算出と注意点 SAS Institute Japan 株式会社 JMP ジャパン事業部 2011 年 10 月改定 1. はじめに 本文書は JMP でロジスティック回帰モデルによるオッズ比 比例ハザードモデルによるリスク比 それぞれに対する信頼区間を求める操作方法と注意点を述べたものです 本文書は JMP 7 以降のバージョンに対応しております
I
 別紙 LG-WAN 決算統計データの操作手順 LG-WANに掲載されている決算統計データをワークシート H24(2012)~S44(1969) の所定の場所にデータを貼り付けることにより 貸借対照表の 有形固定資産 に数値が反映されます LG-WAN 決算統計データは 調査データ閲覧 ダウンロード のページに掲載されています URL: http://llb.k3tokei.asp.lgwan.jp/soumu-app/contents/index.html
別紙 LG-WAN 決算統計データの操作手順 LG-WANに掲載されている決算統計データをワークシート H24(2012)~S44(1969) の所定の場所にデータを貼り付けることにより 貸借対照表の 有形固定資産 に数値が反映されます LG-WAN 決算統計データは 調査データ閲覧 ダウンロード のページに掲載されています URL: http://llb.k3tokei.asp.lgwan.jp/soumu-app/contents/index.html
生存確認調査ツール
 Hos-CanR.0 独自項目運用マニュアル FileMaker pro を使用 登録作業者用 Ver. バージョン改訂日付改訂内容 Ver. 00//5 初版 Ver. 0// FileMaker Pro の動作確認の追加 はじめに 本マニュアルについて Hos-CanR.0 院内がん登録システム ( 以降は Hos-CanR.0 と記述します ) では 独自項目の作成 登録 サポートはなくなり
Hos-CanR.0 独自項目運用マニュアル FileMaker pro を使用 登録作業者用 Ver. バージョン改訂日付改訂内容 Ver. 00//5 初版 Ver. 0// FileMaker Pro の動作確認の追加 はじめに 本マニュアルについて Hos-CanR.0 院内がん登録システム ( 以降は Hos-CanR.0 と記述します ) では 独自項目の作成 登録 サポートはなくなり
Microsoft Word - appendix_b
 付録 B エクセルの使い方 藪友良 (2019/04/05) 統計学を勉強しても やはり実際に自分で使ってみないと理解は十分ではあ りません ここでは 実際に統計分析を使う方法のひとつとして Microsoft Office のエクセルの使い方を解説します B.1 分析ツールエクセルについている分析ツールという機能を使えば さまざまな統計分析が可能です まず この機能を使えるように設定をします もし
付録 B エクセルの使い方 藪友良 (2019/04/05) 統計学を勉強しても やはり実際に自分で使ってみないと理解は十分ではあ りません ここでは 実際に統計分析を使う方法のひとつとして Microsoft Office のエクセルの使い方を解説します B.1 分析ツールエクセルについている分析ツールという機能を使えば さまざまな統計分析が可能です まず この機能を使えるように設定をします もし
 GenBank クイックスタート GenBank は NLM/NCBI にて維持管理されている核酸配列データベースです また GenBank は EMBL, DDBJ と三極間で連携しながら国際核酸配列データベースを共同で構築しています これら三機関はデータを日々交換し続けており その規模は 160000 種にも及ぶ生物種の塩基配列から成り立つまでになっています この GenBank クイックスタートでは
GenBank クイックスタート GenBank は NLM/NCBI にて維持管理されている核酸配列データベースです また GenBank は EMBL, DDBJ と三極間で連携しながら国際核酸配列データベースを共同で構築しています これら三機関はデータを日々交換し続けており その規模は 160000 種にも及ぶ生物種の塩基配列から成り立つまでになっています この GenBank クイックスタートでは
情報工学概論
 確率と統計 中山クラス 第 11 週 0 本日の内容 第 3 回レポート解説 第 5 章 5.6 独立性の検定 ( カイ二乗検定 ) 5.7 サンプルサイズの検定結果への影響練習問題 (4),(5) 第 4 回レポート課題の説明 1 演習問題 ( 前回 ) の解説 勉強時間と定期試験の得点の関係を無相関検定により調べる. データ入力 > aa
確率と統計 中山クラス 第 11 週 0 本日の内容 第 3 回レポート解説 第 5 章 5.6 独立性の検定 ( カイ二乗検定 ) 5.7 サンプルサイズの検定結果への影響練習問題 (4),(5) 第 4 回レポート課題の説明 1 演習問題 ( 前回 ) の解説 勉強時間と定期試験の得点の関係を無相関検定により調べる. データ入力 > aa
地理情報システム論 外部データとのやりとり レイヤのエクスポートによるシェープファイルの作成金沢区を題材とした操作練習では, すでにマップ上に表示されているレイヤを複製することで, 同一のシェープファイルを, 一方は階級区分図に用い, 他方はチャート表示に用いてオーバーレイ表現
 外部データとのやりとり レイヤのエクスポートによるシェープファイルの作成金沢区を題材とした操作練習では, すでにマップ上に表示されているレイヤを複製することで, 同一のシェープファイルを, 一方は階級区分図に用い, 他方はチャート表示に用いてオーバーレイ表現できることを体験した 同様の操作は, 同一のシェープファイルに対して異なる条件のフィルタを設定することで, レイヤを複数使用したい場合にも有用である
外部データとのやりとり レイヤのエクスポートによるシェープファイルの作成金沢区を題材とした操作練習では, すでにマップ上に表示されているレイヤを複製することで, 同一のシェープファイルを, 一方は階級区分図に用い, 他方はチャート表示に用いてオーバーレイ表現できることを体験した 同様の操作は, 同一のシェープファイルに対して異なる条件のフィルタを設定することで, レイヤを複数使用したい場合にも有用である
参考資料 分析ツールからダウンロードするデータについて < 行為明細 > 行為明細データを使いましょう (EVE をお使いの場合は ヘルプの行為明細ダウンロードを参照 ) 各分析ツールのマニュアルを参考にしてください
 参考資料 分析ツールからダウンロードするデータについて < 症例 > 様式 1 をベースに 1 症例 =1 行のデータを使いましょう (EVE をお使いの場合は ヘルプの症例ダウンロードを参照 ) 各分析ツールのマニュアルを参考にしてください 参考資料 分析ツールからダウンロードするデータについて < 行為明細 > 行為明細データを使いましょう (EVE をお使いの場合は ヘルプの行為明細ダウンロードを参照
参考資料 分析ツールからダウンロードするデータについて < 症例 > 様式 1 をベースに 1 症例 =1 行のデータを使いましょう (EVE をお使いの場合は ヘルプの症例ダウンロードを参照 ) 各分析ツールのマニュアルを参考にしてください 参考資料 分析ツールからダウンロードするデータについて < 行為明細 > 行為明細データを使いましょう (EVE をお使いの場合は ヘルプの行為明細ダウンロードを参照
コンテンツ作成基本編
 コンテンツ作成マニュアル基本編 もくじ コンテンツとは 公開する求人検索サイト内の情報の一つ一つを指します 3~7 サイト作成の流れ 求人検索一覧ページ 求人検索を行うためのページを作成するための一覧の流れです 8~8 その他コンテンツについて 各々のページを作成するための コンテンツ管理画面の項目です 9~0 コンテンツとは 3 コンテンツとは コンテンツとは 公開するWebサイトのページつつを指します
コンテンツ作成マニュアル基本編 もくじ コンテンツとは 公開する求人検索サイト内の情報の一つ一つを指します 3~7 サイト作成の流れ 求人検索一覧ページ 求人検索を行うためのページを作成するための一覧の流れです 8~8 その他コンテンツについて 各々のページを作成するための コンテンツ管理画面の項目です 9~0 コンテンツとは 3 コンテンツとは コンテンツとは 公開するWebサイトのページつつを指します
<4D F736F F D F4390B394C5816A8C B835E C835A AA90CD82A982E78CA982E990B68A888F4B8AB595618AC7979D312D332E646F63>
 3 も 飲酒習慣 に替えておきましょう( 図 12) その上で 飲酒分類 をフィールドリストにドラッグして消します 同じように 高血圧判定 もグループ化を図り 1 を 正常血圧 2-4 を 血圧異常 とします 高血圧判定 2 を作り もとの 高血圧判定 を消します これで飲酒と血圧のクロス集計が完成しました ページの選択で 男女の結果 ( 図 13) 男女別の結果( 図 1 4 15) が得られます
3 も 飲酒習慣 に替えておきましょう( 図 12) その上で 飲酒分類 をフィールドリストにドラッグして消します 同じように 高血圧判定 もグループ化を図り 1 を 正常血圧 2-4 を 血圧異常 とします 高血圧判定 2 を作り もとの 高血圧判定 を消します これで飲酒と血圧のクロス集計が完成しました ページの選択で 男女の結果 ( 図 13) 男女別の結果( 図 1 4 15) が得られます
Microsoft Word Proself-guide4STD+Prof.docx
 ファイル共有システム利用の手引き 全学基本メール事業室 1. はじめにメールでファイルを送りたい時に ファイルが大きすぎて送れなかったことはないでしょうか あるいはファイルはそれほど大きくないけれどもファイル数が多くて添付するのに手間がかかったり 届いたメールにたくさんのファイルが添付されていて 一つずつ保存するのが面倒だったことはないでしょうか ここで紹介するファイル共有システムを使うと そうした悩みを一気に解決できます
ファイル共有システム利用の手引き 全学基本メール事業室 1. はじめにメールでファイルを送りたい時に ファイルが大きすぎて送れなかったことはないでしょうか あるいはファイルはそれほど大きくないけれどもファイル数が多くて添付するのに手間がかかったり 届いたメールにたくさんのファイルが添付されていて 一つずつ保存するのが面倒だったことはないでしょうか ここで紹介するファイル共有システムを使うと そうした悩みを一気に解決できます
2. オプション設定画面で, 必要事項を記入 選択します. 少なくとも, タイトル に課題の見出しとなる文章を入力する他, 種別 を アンケート( 無記名式 ) に設定する必要があります. また, アクセス制限はここでは コースメニューで非表示にする に設定します. その他設定は必要に応じて行って下
 (WebClass チュートリアル ) 公開アンケートの実施 ここではアンケート, 特にメンバーを限定せず広く実施する無記名アンケート ( 以下, 公開アンケート ) の実施方法について解説します. 公開アンケートでは, 回答者が WebClass にログインすることなく回答できるというメリットがありますが, 回答資格の判別や, 同一人による複数回の回答をチェックすることが出来ない欠点がありますのでご注意下さい.
(WebClass チュートリアル ) 公開アンケートの実施 ここではアンケート, 特にメンバーを限定せず広く実施する無記名アンケート ( 以下, 公開アンケート ) の実施方法について解説します. 公開アンケートでは, 回答者が WebClass にログインすることなく回答できるというメリットがありますが, 回答資格の判別や, 同一人による複数回の回答をチェックすることが出来ない欠点がありますのでご注意下さい.
Microsoft PowerPoint - Borland C++ Compilerの使用方法(v1.1).ppt [互換モード]
![Microsoft PowerPoint - Borland C++ Compilerの使用方法(v1.1).ppt [互換モード] Microsoft PowerPoint - Borland C++ Compilerの使用方法(v1.1).ppt [互換モード]](/thumbs/92/109692094.jpg) Borland C++ Compiler の 使用方法 解説書 (v1.1) 1 準備 (1/2) 1. スタートメニューから コントロールパネル を開いて その中に デスクトップのカスタマイズ フォルダーオプション があるので開く エクスプローラー内の ツール フォルダーオプション などからも開ける 2. 表示 タブにある 登録されている拡張子は表示しない のチェックを外して OKを押す これでファイルの拡張子が表示されるようになった
Borland C++ Compiler の 使用方法 解説書 (v1.1) 1 準備 (1/2) 1. スタートメニューから コントロールパネル を開いて その中に デスクトップのカスタマイズ フォルダーオプション があるので開く エクスプローラー内の ツール フォルダーオプション などからも開ける 2. 表示 タブにある 登録されている拡張子は表示しない のチェックを外して OKを押す これでファイルの拡張子が表示されるようになった
Microsoft Word - A04 - Configuring Launch In Context_jp-ReviewedandCorrected a.doc
 Launch in Context ( コンテキスト起動 ) の構成 執筆 :Leandro Cassa 本書では Tivoli プロセス自動化エンジンをベースにした製品において Launch In Context (LIC: コンテキスト起動 ) を構成する方法について説明します コンテキスト起動とは コンテキストが割り当てられた外部 Web サイトを起動するアクション サービスを指します 本書では
Launch in Context ( コンテキスト起動 ) の構成 執筆 :Leandro Cassa 本書では Tivoli プロセス自動化エンジンをベースにした製品において Launch In Context (LIC: コンテキスト起動 ) を構成する方法について説明します コンテキスト起動とは コンテキストが割り当てられた外部 Web サイトを起動するアクション サービスを指します 本書では
Microsoft Word - _ ‘C’³_V1.6InstManual.doc
 厚生労働省版医薬品等電子申請ソフト ダウンロードマニュアル 平成 23 年 6 月 17 日版 厚生労働省医薬食品局 はじめに 本マニュアルの利用方法 本マニュアルは 医薬品等電子申請ソフト ( 以下 申請ソフト と呼びます ) のダウンロード方法について説明したものです 申請ソフトのダウンロードは ウェブブラウザを用いたインターネットからの一般のドキュメントやプログラムのダウンロードと大きく異なるところはありません
厚生労働省版医薬品等電子申請ソフト ダウンロードマニュアル 平成 23 年 6 月 17 日版 厚生労働省医薬食品局 はじめに 本マニュアルの利用方法 本マニュアルは 医薬品等電子申請ソフト ( 以下 申請ソフト と呼びます ) のダウンロード方法について説明したものです 申請ソフトのダウンロードは ウェブブラウザを用いたインターネットからの一般のドキュメントやプログラムのダウンロードと大きく異なるところはありません
ゲノム情報解析基礎
 講義資料 PDF が講義のページからダウンロード可能です 印刷物はありません ゲノム情報解析基礎 ~ R で塩基配列解析 ~ 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム 微生物科学イノベーション連携研究機構門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 講義予定
講義資料 PDF が講義のページからダウンロード可能です 印刷物はありません ゲノム情報解析基礎 ~ R で塩基配列解析 ~ 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究プログラム 微生物科学イノベーション連携研究機構門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 講義予定
Microsoft Word - Stattext13.doc
 3 章対応のある 群間の量的データの検定 3. 検定手順 この章では対応がある場合の量的データの検定方法について学びます この場合も図 3. のように最初に正規に従うかどうかを調べます 正規性が認められた場合は対応がある場合の t 検定 正規性が認められない場合はウィルコクソン (Wlcoxo) の符号付き順位和検定を行ないます 章で述べた検定方法と似ていますが ここでは対応のあるデータ同士を引き算した値を用いて判断します
3 章対応のある 群間の量的データの検定 3. 検定手順 この章では対応がある場合の量的データの検定方法について学びます この場合も図 3. のように最初に正規に従うかどうかを調べます 正規性が認められた場合は対応がある場合の t 検定 正規性が認められない場合はウィルコクソン (Wlcoxo) の符号付き順位和検定を行ないます 章で述べた検定方法と似ていますが ここでは対応のあるデータ同士を引き算した値を用いて判断します
【第一稿】論文執筆のためのワード活用術 (1).docx.docx
 ワード活用マニュアル レポート 論文の作成に欠かせない Word の使い方を勉強しましょう ワードはみんなの味方です 使いこなせればレポート 論文の強い味方になってくれます 就職してからも必要とされるスキルなのでこの機会に基本的なところをおさえちゃいましょう 各セクションの最後に練習問題があるので HP に添付されているワークシート (http://www.tufs.ac.jp/common/library/lc/word_work.docx)
ワード活用マニュアル レポート 論文の作成に欠かせない Word の使い方を勉強しましょう ワードはみんなの味方です 使いこなせればレポート 論文の強い味方になってくれます 就職してからも必要とされるスキルなのでこの機会に基本的なところをおさえちゃいましょう 各セクションの最後に練習問題があるので HP に添付されているワークシート (http://www.tufs.ac.jp/common/library/lc/word_work.docx)
プロジェクト概要 ー ヒト全遺伝子 データベース(H-InvDB)の概要と進展
 個別要素技術 2 疾患との関連情報の抽出 予測のための 技術開発 平成 20 年 11 月 18 日産業技術総合研究所バイオメディシナル情報研究センター分子システム情報統合チーム 今西規 1 個別要素技術 2 課題一覧 1 大量文献からの自動知識抽出と文献からの既知疾患原因遺伝子情報の網羅的収集 2 疾患遺伝子情報整備と新規疾患遺伝子候補の予測 3 遺伝子多型情報整備 1 大量文献からの自動知識抽出と
個別要素技術 2 疾患との関連情報の抽出 予測のための 技術開発 平成 20 年 11 月 18 日産業技術総合研究所バイオメディシナル情報研究センター分子システム情報統合チーム 今西規 1 個別要素技術 2 課題一覧 1 大量文献からの自動知識抽出と文献からの既知疾患原因遺伝子情報の網羅的収集 2 疾患遺伝子情報整備と新規疾患遺伝子候補の予測 3 遺伝子多型情報整備 1 大量文献からの自動知識抽出と
Eclipse マニュアル <作成目的> Eclipse のインストール方法などを紹介したページはいろいろありますが 専門用語がわからない初心者でもわか りやすく Eclipse のインストール方法 基本操作などをまとめたマニュアル作成を目的としています <目次> 1 Eclipse のインストール
 Eclipse マニュアル 作成者 桑野和子 藤島梓 Eclipse マニュアル Eclipse のインストール方法などを紹介したページはいろいろありますが 専門用語がわからない初心者でもわか りやすく Eclipse のインストール方法 基本操作などをまとめたマニュアル作成を目的としています 1 Eclipse のインストール Win 版 Linux 版 1 1 J2SE
Eclipse マニュアル 作成者 桑野和子 藤島梓 Eclipse マニュアル Eclipse のインストール方法などを紹介したページはいろいろありますが 専門用語がわからない初心者でもわか りやすく Eclipse のインストール方法 基本操作などをまとめたマニュアル作成を目的としています 1 Eclipse のインストール Win 版 Linux 版 1 1 J2SE
目次 1. システム概要 設置手順 注意事項 動作環境 初期設定 システム設定 ( 環境設定 ) システム設定 ( ログインパスワード変更 ) システム設定 ( ファイルのパスワード変
 厚生労働省版ストレスチェック実施プログラム設置 設定マニュアル Ver.1.1 目次 1. システム概要... 2 2. 設置手順... 3 3. 注意事項... 5 4. 動作環境... 8 5. 初期設定... 9 6. システム設定 ( 環境設定 )... 15 7. システム設定 ( ログインパスワード変更 )... 18 8. システム設定 ( ファイルのパスワード変更 )... 20 9.
厚生労働省版ストレスチェック実施プログラム設置 設定マニュアル Ver.1.1 目次 1. システム概要... 2 2. 設置手順... 3 3. 注意事項... 5 4. 動作環境... 8 5. 初期設定... 9 6. システム設定 ( 環境設定 )... 15 7. システム設定 ( ログインパスワード変更 )... 18 8. システム設定 ( ファイルのパスワード変更 )... 20 9.
SpreadSheet Interface
 CHAPTER 11 この章では (SSI) 変換プラグインについて説明します これは ネットワーク設計情報を NMT と Microsoft Excel 互換フォーマット間で変換するものです SSI では Microsoft Excel のバージョン 6.2 以降を使うことを前提にしています この章の内容は次のとおりです NMT から Microsoft Excel への変換 Microsoft
CHAPTER 11 この章では (SSI) 変換プラグインについて説明します これは ネットワーク設計情報を NMT と Microsoft Excel 互換フォーマット間で変換するものです SSI では Microsoft Excel のバージョン 6.2 以降を使うことを前提にしています この章の内容は次のとおりです NMT から Microsoft Excel への変換 Microsoft
Microsoft PowerPoint - KanriManual.ppt
 環境一般教育 マニュアル 管理者用 1 管理者機能一覧管理者が利用できる機能は以下の通りです 印がついている操作手順について後のページでご説明いたします (p.10) 受講者の学習状況に応じメールを送信することができますメール設定 (p.16) 登録されている受講者の削除が出来ます受講者の削除受講者に関する操作 (p.5) 個人を指定して学習進捗を確認できます学習進捗 (p.3) 受講者を一覧にした受講状況を確認します
環境一般教育 マニュアル 管理者用 1 管理者機能一覧管理者が利用できる機能は以下の通りです 印がついている操作手順について後のページでご説明いたします (p.10) 受講者の学習状況に応じメールを送信することができますメール設定 (p.16) 登録されている受講者の削除が出来ます受講者の削除受講者に関する操作 (p.5) 個人を指定して学習進捗を確認できます学習進捗 (p.3) 受講者を一覧にした受講状況を確認します
Agilent 1色法 2条件比較 繰り返し実験なし
 GeneSpring GX11.0.2 ビギナーズガイド Agilent 1 色法 2 条件の比較繰り返し実験あり 適用 薬剤非投与と投与の解析 Wild type と Knock out の解析 正常細胞と病態細胞の解析 など ビギナーズガイドは 様々なマイクロアレイの実験デザインがあるなかで 実験デザインの種類ごとに適切なデータ解析の流れを 実例とともに紹介するガイドブックです ご自分の実験デザインに適合したガイドをお使いください
GeneSpring GX11.0.2 ビギナーズガイド Agilent 1 色法 2 条件の比較繰り返し実験あり 適用 薬剤非投与と投与の解析 Wild type と Knock out の解析 正常細胞と病態細胞の解析 など ビギナーズガイドは 様々なマイクロアレイの実験デザインがあるなかで 実験デザインの種類ごとに適切なデータ解析の流れを 実例とともに紹介するガイドブックです ご自分の実験デザインに適合したガイドをお使いください
1. MEGA 5 をインストールする 1.1 ダウンロード手順 MEGA のホームページ (http://www.megasoftware.net/index.php) から MEGA 5 software をコンピュータにインストールする 2. 塩基配列を決定する 2.1 Alignment E
 MEGA 5 を用いた塩基配列解析法および分子系統樹作成法 Ver.1 Update: 2012.04.01 ウイルス 疫学研究領域井関博 < 内容 > 1. MEGA 5 をインストールする 1.1 ダウンロード手順 2. 塩基配列を決定する 2.1 Alignment Explorer の起動 2.2 シークエンスデータの入力 2.2.1 テキストファイルから読み込む場合 2.2.2 波形データから読み込む場合
MEGA 5 を用いた塩基配列解析法および分子系統樹作成法 Ver.1 Update: 2012.04.01 ウイルス 疫学研究領域井関博 < 内容 > 1. MEGA 5 をインストールする 1.1 ダウンロード手順 2. 塩基配列を決定する 2.1 Alignment Explorer の起動 2.2 シークエンスデータの入力 2.2.1 テキストファイルから読み込む場合 2.2.2 波形データから読み込む場合
生命情報学
 生命情報学 5 隠れマルコフモデル 阿久津達也 京都大学化学研究所 バイオインフォマティクスセンター 内容 配列モチーフ 最尤推定 ベイズ推定 M 推定 隠れマルコフモデル HMM Verアルゴリズム EMアルゴリズム Baum-Welchアルゴリズム 前向きアルゴリズム 後向きアルゴリズム プロファイル HMM 配列モチーフ モチーフ発見 配列モチーフ : 同じ機能を持つ遺伝子配列などに見られる共通の文字列パターン
生命情報学 5 隠れマルコフモデル 阿久津達也 京都大学化学研究所 バイオインフォマティクスセンター 内容 配列モチーフ 最尤推定 ベイズ推定 M 推定 隠れマルコフモデル HMM Verアルゴリズム EMアルゴリズム Baum-Welchアルゴリズム 前向きアルゴリズム 後向きアルゴリズム プロファイル HMM 配列モチーフ モチーフ発見 配列モチーフ : 同じ機能を持つ遺伝子配列などに見られる共通の文字列パターン
目次 3 14P Wordpressテンプレートの設定方法 15P 17P livedoorテンプレートの設定方法 18P 21P FC2テンプレートの設定方法
 特典テンプレートの設定方法 目次 3 14P Wordpressテンプレートの設定方法 15P 17P livedoorテンプレートの設定方法 18P 21P FC2テンプレートの設定方法 Wordpressテンプレートの設定方法 Wordpressテンプレートの設定方法 この作業を行う前に wordpressのインストールを済ませておいてください 1.テーマのインストール wordpressのインストール後
特典テンプレートの設定方法 目次 3 14P Wordpressテンプレートの設定方法 15P 17P livedoorテンプレートの設定方法 18P 21P FC2テンプレートの設定方法 Wordpressテンプレートの設定方法 Wordpressテンプレートの設定方法 この作業を行う前に wordpressのインストールを済ませておいてください 1.テーマのインストール wordpressのインストール後
次に示す数値の並びを昇順にソートするものとする このソートでは配列の末尾側から操作を行っていく まず 末尾の数値 9 と 8 に着目する 昇順にソートするので この値を交換すると以下の数値の並びになる 次に末尾側から 2 番目と 3 番目の 1
 4. ソート ( 教科書 p.205-p.273) 整列すなわちソートは アプリケーションを作成する際には良く使われる基本的な操作であり 今までに数多くのソートのアルゴリズムが考えられてきた 今回はこれらソートのアルゴリズムについて学習していく ソートとはソートとは与えられたデータの集合をキーとなる項目の値の大小関係に基づき 一定の順序で並べ替える操作である ソートには図 1 に示すように キーの値の小さいデータを先頭に並べる
4. ソート ( 教科書 p.205-p.273) 整列すなわちソートは アプリケーションを作成する際には良く使われる基本的な操作であり 今までに数多くのソートのアルゴリズムが考えられてきた 今回はこれらソートのアルゴリズムについて学習していく ソートとはソートとは与えられたデータの集合をキーとなる項目の値の大小関係に基づき 一定の順序で並べ替える操作である ソートには図 1 に示すように キーの値の小さいデータを先頭に並べる
基礎統計
 基礎統計 第 11 回講義資料 6.4.2 標本平均の差の標本分布 母平均の差 標本平均の差をみれば良い ただし, 母分散に依存するため場合分けをする 1 2 3 分散が既知分散が未知であるが等しい分散が未知であり等しいとは限らない 1 母分散が既知のとき が既知 標準化変量 2 母分散が未知であり, 等しいとき 分散が未知であるが, 等しいということは分かっているとき 標準化変量 自由度 の t
基礎統計 第 11 回講義資料 6.4.2 標本平均の差の標本分布 母平均の差 標本平均の差をみれば良い ただし, 母分散に依存するため場合分けをする 1 2 3 分散が既知分散が未知であるが等しい分散が未知であり等しいとは限らない 1 母分散が既知のとき が既知 標準化変量 2 母分散が未知であり, 等しいとき 分散が未知であるが, 等しいということは分かっているとき 標準化変量 自由度 の t
. 起動 目次 P.. ログイン 画面 P.. メニュー 画面 P.. POS 開示 _ 指定店舗 アイテム別 期間合計 画面 ( レポート A) P. 5. POS 開示 _ 店舗別 指定アイテム 期間合計 画面 ( レポート B) ----
 操作手順書 0 年 0 月 情報システム部 . 起動 目次 ------ P.. ログイン 画面 ------ P.. メニュー 画面 ------ P.. POS 開示 _ 指定店舗 アイテム別 期間合計 画面 ( レポート A) ------ P. 5. POS 開示 _ 店舗別 指定アイテム 期間合計 画面 ( レポート B) ------ P.0 6. POS 開示 _ 指定店舗 指定アイテム
操作手順書 0 年 0 月 情報システム部 . 起動 目次 ------ P.. ログイン 画面 ------ P.. メニュー 画面 ------ P.. POS 開示 _ 指定店舗 アイテム別 期間合計 画面 ( レポート A) ------ P. 5. POS 開示 _ 店舗別 指定アイテム 期間合計 画面 ( レポート B) ------ P.0 6. POS 開示 _ 指定店舗 指定アイテム
eYACHO 管理者ガイド
 eyacho 管理者ガイド 第 1 版 - ios は Cisco の米国およびその他の国における商標または登録商標であり ライセンスに基づき使用されています - Apple ipad は Apple Inc. の商標です - Microsoft, Excel および Internet Explorer は 米国 Microsoft Corporation の 米国およびその他の国における登録商標または商標です
eyacho 管理者ガイド 第 1 版 - ios は Cisco の米国およびその他の国における商標または登録商標であり ライセンスに基づき使用されています - Apple ipad は Apple Inc. の商標です - Microsoft, Excel および Internet Explorer は 米国 Microsoft Corporation の 米国およびその他の国における登録商標または商標です
Python-statistics5 Python で統計学を学ぶ (5) この内容は山田 杉澤 村井 (2008) R によるやさしい統計学 (
 http://localhost:8888/notebooks/... Python で統計学を学ぶ (5) この内容は山田 杉澤 村井 (2008) R によるやさしい統計学 (http://shop.ohmsha.co.jp/shop /shopdetail.html?brandcode=000000001781&search=978-4-274-06710-5&sort=) を参考にしています
http://localhost:8888/notebooks/... Python で統計学を学ぶ (5) この内容は山田 杉澤 村井 (2008) R によるやさしい統計学 (http://shop.ohmsha.co.jp/shop /shopdetail.html?brandcode=000000001781&search=978-4-274-06710-5&sort=) を参考にしています
正誤表(FPT1004)
 1 Introduction 本書で学習を進める前に ご一読ください 1 第 1 章関数の利用 第 章表作成の活用 第 3 章グラフの活用 第 章グラフィックの利用 SmartArt 第 5 章複数ブックの操作 第 章データベースの活用 第 7 章ピボットテーブルとピボットグラフの作成 第 章マクロの作成 第 9 章便利な機能 総合問題 Excel 付録 1 ショートカットキー一覧 Excel 付録
1 Introduction 本書で学習を進める前に ご一読ください 1 第 1 章関数の利用 第 章表作成の活用 第 3 章グラフの活用 第 章グラフィックの利用 SmartArt 第 5 章複数ブックの操作 第 章データベースの活用 第 7 章ピボットテーブルとピボットグラフの作成 第 章マクロの作成 第 9 章便利な機能 総合問題 Excel 付録 1 ショートカットキー一覧 Excel 付録
PowerPoint Presentation
 5. スマホ版導入マニュアル 5-1. はじめに これまでスマートフォンでアクセスした場合 PC 版 が表示されておりましたが スマートフォン版 ( 以下スマホ版 ) が表示されます 基本的には PC 用に登録いただいている情報を流用してスマホ版でも表示されるようになりますが 一部スマホ用に登録いただく必要があります スマホ用の情報登録方法と開店までの流れをマニュアルにてご説明いたします 以前 携帯用
5. スマホ版導入マニュアル 5-1. はじめに これまでスマートフォンでアクセスした場合 PC 版 が表示されておりましたが スマートフォン版 ( 以下スマホ版 ) が表示されます 基本的には PC 用に登録いただいている情報を流用してスマホ版でも表示されるようになりますが 一部スマホ用に登録いただく必要があります スマホ用の情報登録方法と開店までの流れをマニュアルにてご説明いたします 以前 携帯用
論文題目 腸管分化に関わるmiRNAの探索とその発現制御解析
 論文題目 腸管分化に関わる microrna の探索とその発現制御解析 氏名日野公洋 1. 序論 microrna(mirna) とは細胞内在性の 21 塩基程度の機能性 RNA のことであり 部分的相補的な塩基認識を介して標的 RNA の翻訳抑制や不安定化を引き起こすことが知られている mirna は細胞分化や増殖 ガン化やアポトーシスなどに関与していることが報告されており これら以外にも様々な細胞諸現象に関与していると考えられている
論文題目 腸管分化に関わる microrna の探索とその発現制御解析 氏名日野公洋 1. 序論 microrna(mirna) とは細胞内在性の 21 塩基程度の機能性 RNA のことであり 部分的相補的な塩基認識を介して標的 RNA の翻訳抑制や不安定化を引き起こすことが知られている mirna は細胞分化や増殖 ガン化やアポトーシスなどに関与していることが報告されており これら以外にも様々な細胞諸現象に関与していると考えられている
Microsoft PowerPoint - 6_TS-0891(TS-0835(Custom TaqMan Assay Design Tool利用方法修正5.pptx
 Custom TaqMan Assay Design Tool インターネットオーダー方法 20010/06/01 Custom TaqMan Assay Design Tool 1. Custom TaqMan SNP Genotyping Assays P.5 2. Custom TaqMan Gene Expression Assays P.21 3. カスタムデザインでのオーダー P.30
Custom TaqMan Assay Design Tool インターネットオーダー方法 20010/06/01 Custom TaqMan Assay Design Tool 1. Custom TaqMan SNP Genotyping Assays P.5 2. Custom TaqMan Gene Expression Assays P.21 3. カスタムデザインでのオーダー P.30
特論I
 講義室後ろにある USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 農学生命情報科学特論 I 第 4 回 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 kadota@iu.a.u-tokyo.ac.jp 1 前回の課題と正答 アダプター配列除去前後の small RNA-seq データをカイコゲノムにマップし マップ率 ( マップされたリード数
講義室後ろにある USB メモリ中の hoge フォルダをデスクトップにコピーしておいてください 農学生命情報科学特論 I 第 4 回 東京大学大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 kadota@iu.a.u-tokyo.ac.jp 1 前回の課題と正答 アダプター配列除去前後の small RNA-seq データをカイコゲノムにマップし マップ率 ( マップされたリード数
様々なミクロ計量モデル†
 担当 : 長倉大輔 ( ながくらだいすけ ) この資料は私の講義において使用するために作成した資料です WEB ページ上で公開しており 自由に参照して頂いて構いません ただし 内容について 一応検証してありますが もし間違いがあった場合でもそれによって生じるいかなる損害 不利益について責任を負いかねますのでご了承ください 間違いは発見次第 継続的に直していますが まだ存在する可能性があります 1 カウントデータモデル
担当 : 長倉大輔 ( ながくらだいすけ ) この資料は私の講義において使用するために作成した資料です WEB ページ上で公開しており 自由に参照して頂いて構いません ただし 内容について 一応検証してありますが もし間違いがあった場合でもそれによって生じるいかなる損害 不利益について責任を負いかねますのでご了承ください 間違いは発見次第 継続的に直していますが まだ存在する可能性があります 1 カウントデータモデル
NGS速習コース
 バイオインフォマティクス人材育成カリキュラム ( 次世代シークエンサ ) 速習コース 3. データ解析基礎 3-4. R Bioconductor I 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 1 Contents
バイオインフォマティクス人材育成カリキュラム ( 次世代シークエンサ ) 速習コース 3. データ解析基礎 3-4. R Bioconductor I 東京大学 大学院農学生命科学研究科アグリバイオインフォマティクス教育研究ユニット門田幸二 ( かどたこうじ ) kadota@iu.a.u-tokyo.ac.jp http://www.iu.a.u-tokyo.ac.jp/~kadota/ 1 Contents
